Complementación de la actividad de empalme por un complejo de galectina-3 - U1 snRNP en perlas
Summary
Este artículo describe los procedimientos experimentales para (a) el agotamiento de U1 snRNP de extractos nucleares, con pérdida concomitante de la actividad de empalme; y (b) reconstitución de la actividad de empalme en el extracto agotado de U1 por partículas de galectina-3 – U1 snRNP unidas a perlas covalentemente acopladas con anticuerpos anti-galectina-3.
Abstract
Los experimentos clásicos de agotamiento-reconstitución indican que la galectina-3 es un factor de empalme requerido en los extractos nucleares. El mecanismo de incorporación de galectina-3 en la vía de empalme se aborda en este trabajo. La sedimentación de extractos nucleares de células HeLa en gradientes de glicerol del 12% al 32% produce fracciones enriquecidas en una partícula endógena ~ 10S que contiene galectina-3 y U1 snRNP. Ahora describimos un protocolo para agotar los extractos nucleares de U1 snRNP con pérdida concomitante de la actividad de empalme. La actividad de empalme en el extracto agotado de U1 puede ser reconstituida por la partícula snRNP de galectina-3 – U1 atrapada en las perlas de agarosa acoplada covalentemente con anticuerpos anti-galectina-3. Los resultados indican que el complejo ternario galectina-3 – U1 snRNP – pre-ARNm es un complejo E funcional que conduce a intermediarios y productos de la reacción de empalme y que la galectina-3 entra en la vía de empalme a través de su asociación con U1 snRNP. El esquema de uso de complejos de afinidad o inmunoseleccionados en perlas para reconstituir la actividad de empalme en extractos agotados de un factor de empalme específico puede ser generalmente aplicable a otros sistemas.
Introduction
La producción de la mayoría de los ARN mensajeros eucariotas (ARNm) implica la eliminación de intrones y la ligadura de exones en un proceso nuclear denominado empalme pre-ARNm1. Dos clases de complejos ARN-proteína (RNPs) dirigen el procesamiento del ARN pre-mensajero en ARNm maduro a través de complejos espliceosómicos. Una clase, los RNPs pre-mensajeros nacientes, se forma co-transcripcionalmente por la unión de proteínas RNP nucleares heterogéneas y otras proteínas de unión a ARN, incluyendo algunos miembros de la familia SR, produciendo complejos hnRNP2. Los RNPs nucleares pequeños ricos en uracilo de segunda clase (U snRNPs con SnRNAs U1, U2, U4, U5 y U6) se asocian con proteínas específicas de U y del núcleo3,4. Los snRNPs U interactúan de manera ordenada con regiones específicas de RNPs pre-mensajeros en una vía de remodelación dinámica a medida que los intrones se extirpan y los exones se ligan para producir mRNPs maduros5. Muchas proteínas nucleares adicionales participan en estos eventos de procesamiento6.
La galectina-1 (Gal1) y la galectina-3 (Gal3) son dos proteínas que son factores necesarios en la vía de empalme como lo demuestran los estudios de agotamiento-reconstitución7,8. La eliminación de ambas galectinas del empalme de extractos nucleares competentes (NE) suprime el ensamblaje del espliceosoma y la actividad de empalme en un paso temprano. La adición de cualquiera de las dos galectinas a un NE tan doblemente agotado restaura ambas actividades. Gal1 y Gal3 son componentes de los empaliceosomas activos, como lo demuestra la inmunoprecipitación específica de pre-ARNm, intermedios de empalme y ARNm maduro por antisuero específico para Gal1 o Gal39. Es importante destacar que Gal3 se asocia con partículas endógenas U snRNA que contienen partículas en el NE fuera de la vía de empalme, como lo demuestra la precipitación de snRNPs por anti-Gal3 antisera10. Finalmente, el silenciamiento de Gal3 en células HeLa altera los patrones de empalme de numerosos genes11.
En NE preincubado para desmontar spliceosomas preformados12, los snRNPs se encuentran en múltiples complejos sedimentando en gradientes de glicerol desde 7S hasta mayores de 60S. Aunque el fraccionamiento del gradiente de glicerol es una técnica común para el aislamiento de complejos y componentes espliceosómicos (ver referencias13,14,15 por ejemplo), hemos ampliado este método caracterizando fracciones específicas mediante inmunoprecipitaciones de anticuerpos. Un sedimento snRNP a 10S contiene solo SnRNA U1 junto con Gal3. La inmunoprecipitación de la fracción 10S con antisueros específicos para Gal3 o U1 snRNP co-precipita tanto U1 como Gal3 indicando que algunas de las monopartículas U1 snRNP están unidas a Gal310. Como U1 snRNP es el primer complejo que se une a pre-mRNP en el ensamblaje espliceosómico1,5, este paso representa un sitio de entrada potencial para Gal3 en la vía de empalme. Sobre esta base, demostramos que las monopartículas 10S Gal3-U1 snRNP unidas a perlas que contienen anti-Gal3 restauraron la actividad de empalme a un NE agotado U1 snRNP, estableciendo este complejo como un mecanismo por el cual Gal3 es reclutado en la vía espliceosomal16. Esto contrasta con los intentos de aislar los espliceosomas en etapas específicas de la reacción de empalme y catalogar los factores asociados17,18. En tales estudios, se determina la presencia de ciertos factores en algún momento, pero no el mecanismo por el cual se cargaron.
Anteriormente habíamos descrito en detalle la preparación de NE, el sustrato de empalme, el ensamblaje de la mezcla de reacción de empalme y el análisis de productos en nuestra documentación del papel de las galectinas en el empalme pre-ARNm19. Ahora describimos los procedimientos experimentales para el fraccionamiento de extractos nucleares para obtener una fracción enriquecida en el complejo Gal3 – U1 snRNP y para la inmunoselección de este último complejo para reconstituir la actividad de empalme en un extracto nuclear agotado de U1.
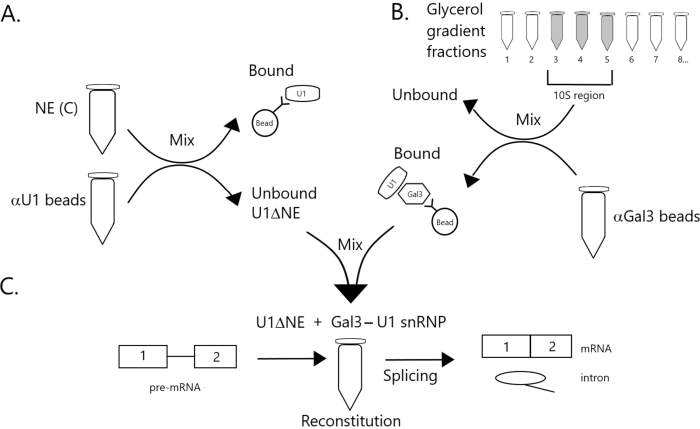
Figura 1: Diagrama esquemático que ilustra la complementación de la actividad de empalme en extracto nuclear agotado de U1 snRNP por un complejo Gal3-U1 snRNP en perlas. (A) NE en Buffer C (NE(C)) se incuba con perlas de proteína A-sefalrosa covalentemente acopladas con snRNP anti-U1 (perlas αU1). La fracción no unida se agota de U1 snRNP (U1ΔNE). (B) NE en el tampón D (NE(D)) se fracciona sobre un gradiente de glicerol de 12%-32% por ultracentrifugación. Las fracciones correspondientes a la región 10S (fracciones 3-5) se combinan y mezclan con perlas covalentemente acopladas con anticuerpos anti-Gal3 (perlas αGal3). El material unido a las perlas contiene una monopartícula Gal3-U1 snRNP. (C) El complejo snRNP Gal3-U1 de la Parte (B) se mezcla con U1ΔNE de la Parte (A) en un ensayo de empalme utilizando sustrato de pre-ARNm MINX marcado con 32P y los intermedios y productos de la reacción de empalme se analizan mediante electroforesis en gel y autorradiografía. Haga clic aquí para ver una versión más grande de esta figura.
Protocol
Representative Results
Discussion
Este informe proporciona los detalles experimentales que documentan que un complejo snRNP Gal3 – U1 atrapado en perlas recubiertas anti-Gal3 puede unirse al sustrato pre-ARNm y este complejo ternario puede restaurar la actividad de empalme a un NE agotado de U1 snRNP. Gal3 es un miembro de una familia de proteínas originalmente aisladas sobre la base de su actividad de unión a carbohidratos específicos de galactosa23 . Los estudios tempranos de inmunofluorescencia y fraccionamiento subcelular p…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Este trabajo ha sido apoyado por national Science Foundation Grant MCB-0092919 y Michigan State University Intramural Research Grant 09-CDFP-2001 (a RJP) y por National Institutes of Health Grant GM-38740 y Michigan AgBioResearch Project MICL02455 (a JLW).
El sustrato minx de pre-ARNm utilizado en los ensayos de empalme fue un amable regalo de la Dra. Susan Berget (Baylor College of Medicine, Houston, TX, EE. UU.).
Materials
| anti-U1 snRNP | The Binding Site | Hu ENA-RNP #33471 | human autoimmune serum specific for U1 snRNP |
| bottle top vacuum filter | Fisher Scientific | Corning 431153 (0.22 μm; PES 150 ml) | for filtering solutions containing Tris |
| centrifuge | International Equipment Company | IEC Model PR-6 | for pelletting Sepharose beads in immunoprecipitation |
| diethylpyrocarbonate (DEPC) | Sigma-Aldrich | 159220-5G | for treatment of water used in preparation of all solutions |
| dimethylpimelimidate (DMP) | Sigma-Aldrich | 80490-5G | for cross-linking antibody to Sepharose beads |
| electrophoresis cell | BioRad Laboratories, Inc | Mini-Protean II | for SDS-PAGE separation of proteins |
| ethanolamine | Sigma-Aldrich | 411000-100ml | for blocking after the cross-linking reaction |
| gel electrophoresis system | Hoefer, Inc | HSI SE 500 Series | for separating snRNAs by gel electrophoresis |
| gel slab dryer | BioRad | Model 224 | for drying gel slabs for autoradiography |
| Hybond ECL membrane | GE Healthcare | RPN3032D (0.2 μm; 30 cm x 3 m) | for immunoblotting of proteins on membrane |
| microdialyzer (12 x 100 μl sample capacity) | Pierce | Microdialyzer System 100 | for exchanging the buffer of nuclear extract |
| microdialyzer membranes (8K cutoff) | Pierce | 66310 | for exchanging the buffer of nuclear extract |
| non-fat dry milk | Spartan Stores | Spartan Instant Non-fat Dry Milk | |
| Protein A Sepharose CL-4B | Millipore-Sigma | GE 17-0780-01 | for coupling antibody to beads |
| Proteinase K | Millipore-Sigma | P2308-5mg | for stopping the splicing reaction to isolate the RNAs |
| RNasin | Promega | N2111 | for inhibiting ribonuclease activity |
| rocker/rotator | Lab Industries, Inc | Labquake Shaker 400-110 | for mixing protein solutions in coupling reactions and in immunoprecipitation |
| Safety-Solve | Research Products International Corp. | No. 111177 | scintillation counting cocktail for determination of radioactivity in splicing substrate |
| scintillation counter | Beckman Instruments | LS6000SC | scintillation counter for determination of radioactivity |
| speed vaccum concentrator | Savant | SVC 100H | for drying ethanol-precipitated RNA pellets |
| Transphor electrophoresis unit | Hoefer, Inc | Hoefer TE Series Transphor | for protein transfer from SDS-PAGE to blotting membrane |
Riferimenti
- Hoskins, A. A., Moore, M. J. The spliceosome: a flexible, reversible macromolecular machine. Trends In Biochemical Sciences. 37, 179-188 (2012).
- Choi, Y. D., Grabowski, P., Sharp, P. A., Dreyfuss, G. Heterogeneous nuclear ribonucleoproteins: role in RNA splicing. Science. 231, 1534-1539 (1986).
- Lerner, M., Steitz, J. A. Snurps and scyrps. Cell. 25, 298-300 (1981).
- Maniatis, T., Reed, R. The role of small nuclear ribonucleoprotein particles in pre-mRNA splicing. Nature. 325, 673-678 (1987).
- Hoskins, A. A., et al. Ordered and dynamic assembly of single spliceosomes. Science. 331, 1289-1295 (2011).
- Coppin, L., Leclerc, J., Vincent, A., Porchet, N., Pigny, P. Messenger RNA life-cycle in cancer: emerging role of conventional and non-conventional RNA-binding proteins. International Journal of Molecular Sciences. 19, 650-676 (2018).
- Dagher, S. F., Wang, J. L., Patterson, R. J. Identification of galectin-3 as a factor in pre-mRNA splicing. Proceedings of the National Academy of Sciences of the United States of America. 92, 1213-1217 (1995).
- Vyakarnam, A., Dagher, S. F., Wang, J. L., Patterson, R. J. Evidence for a role for galectin-1 in pre-mRNA splicing. Molecular and Cellular Biology. 17, 4730-4737 (1997).
- Wang, W., Park, J. W., Wang, J. L., Patterson, R. J. Immunoprecipitation of spliceosomal RNAs by antisera to galectin-1 and galectin-3. Nucleic Acids Research. 34, 5166-5174 (2006).
- Haudek, K. C., Voss, P. G., Locascio, L. E., Wang, J. L., Patterson, R. J. A mechanism for incorporation of galectin-3 into the spliceosome through its association with U1 snRNP. Biochimica. 48, 7705-7712 (2009).
- Fritsch, K., et al. Galectin-3 interacts with components of the nuclear ribonucleoprotein complex. BMC Cancer. 16, 502-511 (2016).
- Conway, G. C., Krainer, A. R., Spector, D. L., Roberts, R. J. Multiple splicing factors are released from endogenous complexes during in vitro pre-mRNA splicing. Molecular and Cellular Biology. 9, 5273-5280 (1989).
- Dery, K. J., Yean, S. L., Lin, R. J. Assembly and glycerol gradient isolation of yeast spliceosomes containing transcribed or synthetic U6 snRNA. Methods in Molecular Biology. 488, 41-63 (2008).
- Yoshimoto, R., Kataoka, N., Okawa, K., Ohno, M. Isolation and characterization of post-splicing lariat-intron complexes. Nucleic Acids Research. 37, 891-902 (2009).
- Malca, H., Shomron, N., Ast, G. The U1 snRNP base pairs with the 5′ splice site within a penta-snRNP complex. Molecular and Cellular Biology. 23, 3442-3455 (2003).
- Haudek, K. C., Voss, P. G., Wang, J. L., Patterson, R. J. A 10S galectin-3 – snRNP complex assembles into active spliceosomes. Nucleic Acids Research. 44, 6391-6397 (2016).
- Rappsilber, J., Ryder, U., Lamond, A. I., Mann, M. Large-scale proteomic analysis of the human spliceosome. Genome Research. 12, 1231-1245 (2002).
- Jurica, M. S., Moore, M. J. Capturing splicing complexes to study structure and mechanism. Methods. 28, 336-345 (2002).
- Patterson, R. J., Haudek, K. C., Voss, P. G., Wang, J. L. Examination of the role of galectins in pre-mRNA splicing. Methods in Molecular Biology. 1207, 431-449 (2015).
- Dignam, J. D., Lebovitz, R. M., Roeder, R. G. Accurate transcription initiation by RNA polymerase II in a soluble extract from isolated mammalian nuclei. Nucleic Acids Research. 11, 1475-1489 (1983).
- Agarwal, N., Sun, Q., Wang, S. Y., Wang, J. L. Carbohydrate-binding protein 35. I. Properties of the recombinant polypeptide and the individuality of the domains. Journal of Biological Chemistry. 268, 14932 (1993).
- Zillmann, M., Zapp, M. I., Berget, S. M. Gel electrophoretic isolation of splicing complexes containing U1 small nuclear ribonucleoprotein particles. Molecular and Cellular Biology. 8, 814-821 (1988).
- Barondes, S. H., et al. Galectins: a family of animal β-galactoside-binding proteins. Cell. 76, 597-598 (1994).
- Laing, J. G., Wang, J. L. Identification of carbohydrate binding protein 35 in heterogeneous nuclear ribonucleoprotein complex. Biochimica. 27, 5329-5334 (1988).
- Vyakarnam, A., Lenneman, A. J., Lakkides, K. M., Patterson, R. J., Wang, J. L. A comparative nuclear localization study of galectin-1 with other splicing components. Experimental Cell Research. 242, 419-428 (1998).
- Michaud, S., Reed, R. An ATP-independent complex commits pre-mRNA to the mammalian spliceosome assembly pathway. Genes & Development. 5, 2534-2546 (1991).
- Chiu, Y. -. F., et al. Cwc25 is a novel splicing factor required after Prp2 and Yju2 to facilitate the first catalytic reaction. Molecular and Cellular Biology. 29, 5671-5678 (2009).
- Krishnan, R., et al. Biased Brownian ratcheting leads to pre-mRNA remodeling and capture prior to first-step splicing. Nature Structural and Molecular Biology. 20, 1450-1457 (2013).
- Gray, R. M., et al. Distinct effects on splicing of two monoclonal antibodies directed against the amino-terminal domain of galectin-3. Archives of Biochemistry and Biophysics. 475, 100-108 (2008).

