एक Galectin-3 द्वारा Splicing गतिविधि के पूरक - मोती पर U1 snRNP परिसर
Summary
यह आलेख (ए) परमाणु अर्क से U1 snRNP की कमी के लिए प्रयोगात्मक प्रक्रियाओं का वर्णन करता है, splicing गतिविधि के सहवर्ती नुकसान के साथ; और (ख) गैलेक्टिन-3 द्वारा U1-समाप्त अर्क में splicing गतिविधि का पुनर्गठन – U1 snRNP कणों को मोतियों के लिए बाध्य सहसंयोजक रूप से विरोधी गैलेक्टिन -3 एंटीबॉडी के साथ युग्मित।
Abstract
क्लासिक कमी-पुनर्गठन प्रयोगों से संकेत मिलता है कि गैलेक्टिन -3 परमाणु अर्क में एक आवश्यक स्प्लिसिंग कारक है। स्प्लिसिंग मार्ग में गैलेक्टिन -3 को शामिल करने के तंत्र को इस पेपर में संबोधित किया गया है। 12% -32% ग्लिसरॉल ग्रेडिएंट पर हेला सेल परमाणु अर्क का अवसादन एक अंतर्जात ~ 10एस कण में समृद्ध अंशों को उत्पन्न करता है जिसमें गैलेक्टिन -3 और यू 1 एसएनआरएनएपी शामिल हैं। अब हम एक प्रोटोकॉल का वर्णन करने के लिए splicing गतिविधि के सहवर्ती नुकसान के साथ U1 snRNP के परमाणु अर्क को कम करने के लिए. U1-समाप्त निकालने में splicing गतिविधि गैलेक्टिन-3 द्वारा पुनर्गठित किया जा सकता है – U1 snRNP कण agarose मोतियों पर फंसे सहसंयोजक विरोधी गैलेक्टिन -3 एंटीबॉडी के साथ युग्मित. परिणामों से संकेत मिलता है कि गैलेक्टिन -3 – U1 snRNP – प्री-एमआरएनए टर्नरी कॉम्प्लेक्स एक कार्यात्मक ई कॉम्प्लेक्स है जो मध्यवर्ती और स्प्लिसिंग प्रतिक्रिया के उत्पादों के लिए अग्रणी है और गैलेक्टिन -3 यू 1 एसएनआरएनएपी के साथ अपने सहयोग के माध्यम से स्प्लिसिंग मार्ग में प्रवेश करता है। एक विशिष्ट स्प्लिसिंग कारक के समाप्त होने वाले अर्क में स्प्लिसिंग गतिविधि को पुनर्गठित करने के लिए मोतियों पर जटिल आत्मीयता- या इम्यूनो-चयनित का उपयोग करने की योजना आमतौर पर अन्य प्रणालियों पर लागू हो सकती है।
Introduction
अधिकांश यूकेरियोटिक मैसेंजर आरएनए (एमआरएनए) के उत्पादन में एक परमाणु प्रक्रिया में इंट्रोन को हटाना और एक्सोन के बंधाव को शामिल करना शामिल है जिसे प्री-एमआरएनए स्प्लिसिंग 1 कहा जाता है। आरएनए-प्रोटीन कॉम्प्लेक्स (आरएनपी) के दो वर्ग पूर्व-मैसेंजर आरएनए के प्रसंस्करण को स्प्लिसोसोमल कॉम्प्लेक्स के माध्यम से परिपक्व एमआरएनए में निर्देशित करते हैं। एक वर्ग, नवजात पूर्व-दूत आरएनपी, विषम परमाणु आरएनपी प्रोटीन और अन्य आरएनए-बाइंडिंग प्रोटीन के बंधन द्वारा सह-ट्रांसक्रिप्शनल रूप से बनाया जाता है, जिसमें एसआर परिवार के कुछ सदस्य शामिल हैं, जो एचएनआरएनपी कॉम्प्लेक्स 2 की उपज देते हैं। दूसरी श्रेणी, यूरासिल-समृद्ध छोटे परमाणु आरएनपी (U1, U2, U4, U5, और U6 snRNAs के साथ U snRNPs) यू-विशिष्ट और कोर प्रोटीन 3,4 के साथ जुड़ा हुआ है। यू snRNPs एक गतिशील remodeling मार्ग में पूर्व मैसेंजर RNPs के विशिष्ट क्षेत्रों के साथ एक आदेशित फैशन में बातचीत के रूप में introns excised रहे हैं और exons परिपक्व mRNPs5 का उत्पादन करने के लिए ligated रहे हैं. कई अतिरिक्त परमाणु प्रोटीन इन प्रसंस्करण घटनाओं में भाग लेते हैं6।
गैलेक्टिन -1 (Gal1) और गैलेक्टिन -3 (Gal3) दो प्रोटीन हैं जो स्प्लिसिंग मार्ग में आवश्यक कारक हैं जैसा कि कमी-पुनर्गठन अध्ययन7,8 द्वारा दिखाया गया है। सक्षम परमाणु अर्क (एनई) को स्प्लिसिंग से दोनों गैलेक्टिन को हटाने से स्प्लिसोसोम असेंबली और स्प्लिसिंग गतिविधि को शुरुआती चरण में समाप्त कर दिया जाता है। इस तरह के दोगुने समाप्त एनई में या तो गैलेक्टिन के अलावा दोनों गतिविधियों को बहाल करता है। Gal1 और Gal3 सक्रिय spliceosomes के घटक हैं जैसा कि पूर्व-mRNA के विशिष्ट immunoprecipitation, splicing intermediates, और परिपक्व mRNA द्वारा या तो Gal1 या Gal39 के लिए विशिष्ट एंटीसीरम द्वारा प्रमाणित है। महत्वपूर्ण रूप से, Gal3 अंतर्जात U snRNA के साथ सहयोग करता है जिसमें splicing pathway के बाहर NE में कण होते हैं, जैसा कि एंटी-Gal3 antisera10 द्वारा snRNPs की वर्षा द्वारा दिखाया गया है। अंत में, हेला कोशिकाओं में Gal3 की चुप्पी कई जीन11 के splicing पैटर्न को बदल देती है।
पूर्वनिर्मित spliceosomes12 को अलग करने के लिए एनई प्री-इनक्यूबेटेड में, snRNPs कई परिसरों में पाए जाते हैं जो 7S से 60S से अधिक तक ग्लिसरॉल ग्रेडिएंट में तलछट करते हैं। यद्यपि ग्लिसरॉल ग्रेडिएंट फ्रैक्शनेशन स्प्लिसोसोमल कॉम्प्लेक्स और घटकों के अलगाव के लिए एक आम तकनीक है (उदाहरण के लिए संदर्भ 13,14,15 देखें), हमने एंटीबॉडी इम्यूनोप्रिसिपेशन का उपयोग करके विशिष्ट अंशों की विशेषता करके इस विधि का विस्तार किया है। 10S पर एक snRNP तलछट में Gal3 के साथ केवल U1 snRNA होता है। Gal3 या U1 snRNP सह-अवक्षेप दोनों U1 और Gal3 के लिए विशिष्ट antisera के साथ 10S अंश के immunoprecipitation U1 और Gal3 दोनों को दर्शाता है कि U1 snRNP मोनोपार्टिकल्स में से कुछ Gal310 के लिए बाध्य हैं। U1 snRNP के रूप में पहला जटिल है कि spliceosomal assembly1,5 में पूर्व-mRNP के लिए बांधता है, यह कदम splicing मार्ग में Gal3 के लिए एक संभावित प्रवेश साइट का प्रतिनिधित्व करता है। इस आधार पर, हमने दिखाया कि 10S Gal3-U1 snRNP मोनोपार्टिकल्स एंटी-गैल 3 से बंधे हैं, जिसमें मोतियों ने U1 snRNP को समाप्त करने के लिए splicing गतिविधि को बहाल किया है, इस परिसर को एक तंत्र के रूप में स्थापित किया गया है जिसके द्वारा Gal3 को spliceosomal pathway16 में भर्ती किया जाता है। यह splicing प्रतिक्रिया में विशिष्ट चरणों में spliceosomes को अलग करने और संबंधित कारकों को सूचीबद्ध करने के प्रयासों के साथ विरोधाभास करता है17,18। इस तरह के अध्ययनों में, कुछ समय बिंदु पर कुछ कारकों की उपस्थिति का पता लगाया जाता है लेकिन उस तंत्र को नहीं जिसके द्वारा उन्हें लोड किया गया था।
हमने पहले एनई की तैयारी, स्प्लिसिंग सब्सट्रेट, स्प्लिसिंग प्रतिक्रिया मिश्रण की असेंबली, और प्री-एमआरएनए स्प्लिसिंग 19 में गैलेक्टिन की भूमिका के हमारे प्रलेखन में उत्पादों के विश्लेषण का विस्तार से वर्णन किया था। अब हम परमाणु अर्क के फ्रैक्शनेशन के लिए प्रयोगात्मक प्रक्रियाओं का वर्णन करने के लिए Gal3 – U1 snRNP परिसर में समृद्ध एक अंश प्राप्त करने के लिए और बाद के परिसर के immuno-चयन के लिए एक U1-depleted परमाणु निकालने में splicing गतिविधि का पुनर्गठन करने के लिए।
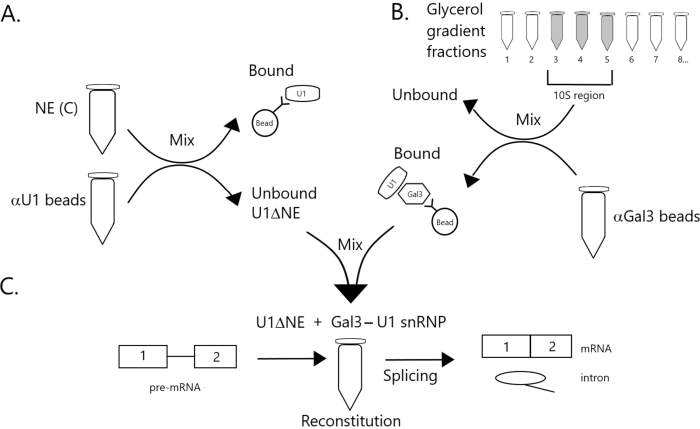
चित्र 1: परमाणु अर्क में स्प्लिसिंग गतिविधि के पूरक को दर्शाने वाला योजनाबद्ध आरेख मोती पर एक Gal3-U1 snRNP परिसर द्वारा U1 snRNP के समाप्त हो गए हैं। (A) बफर C (NE(C)) में NE को प्रोटीन A-Sepharose मोतियों के साथ सहसंयोजक रूप से एंटी-U1 snRNP (αU1 मोतियों) के साथ युग्मित किया गया है। अनबाउंड भिन्न U1 snRNP (U1ΠNE) से समाप्त हो गया है। (बी) बफर डी (एनई (डी)) में एनई को अल्ट्रासेंट्रीफ्यूजेशन द्वारा 12% -32% ग्लिसरॉल ग्रेडिएंट पर विभाजित किया जाता है। 10S क्षेत्र (अंश 3-5) के अनुरूप अंशों को संयुक्त किया जाता है और मोतियों के साथ मिश्रित किया जाता है जो एंटी-गैल 3 एंटीबॉडी (αGal3 मोतियों) के साथ सहसंयोजक रूप से युग्मित होते हैं। मोतियों के लिए बाध्य सामग्री एक Gal3-U1 snRNP मोनोपार्टिकल शामिल हैं। (ग) भाग (बी) से Gal3-U1 snRNP परिसर को भाग (ए) से U1ΠNE के साथ मिश्रित किया जाता है, जिसमें 32P-लेबल वाले MINX पूर्व-mRNA सब्सट्रेट का उपयोग किया जाता है और splicing प्रतिक्रिया के मध्यवर्ती और उत्पादों का विश्लेषण जेल वैद्युतकणसंचलन और ऑटोरेडियोग्राफी द्वारा किया जाता है। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहाँ क्लिक करें.
Protocol
Representative Results
Discussion
यह रिपोर्ट प्रयोगात्मक विवरण प्रदान करती है जो एक Gal3 – U1 snRNP परिसर को एंटी-गैल 3 लेपित मोतियों पर फंसा हुआ दस्तावेज करती है, पूर्व-mRNA सब्सट्रेट को बांध सकती है और यह टर्नरी कॉम्प्लेक्स एक U1 snRNP-depleted NE में स्प्लिस?…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
इस काम को राष्ट्रीय विज्ञान फाउंडेशन अनुदान MCB-0092919 और मिशिगन स्टेट यूनिवर्सिटी इंट्राम्यूरल रिसर्च ग्रांट 09-CDFP-2001 (RJP के लिए) और नेशनल इंस्टीट्यूट ऑफ हेल्थ ग्रांट जीएम -38740 और मिशिगन AgBioResearch Project MICL02455 (JLW के लिए) द्वारा समर्थित किया गया है।
MINX पूर्व mRNA सब्सट्रेट splicing assays में इस्तेमाल किया डॉ सुसान Berget (Baylor College of Medicine, ह्यूस्टन, TX, संयुक्त राज्य अमेरिका) से एक तरह का उपहार था।
Materials
| anti-U1 snRNP | The Binding Site | Hu ENA-RNP #33471 | human autoimmune serum specific for U1 snRNP |
| bottle top vacuum filter | Fisher Scientific | Corning 431153 (0.22 μm; PES 150 ml) | for filtering solutions containing Tris |
| centrifuge | International Equipment Company | IEC Model PR-6 | for pelletting Sepharose beads in immunoprecipitation |
| diethylpyrocarbonate (DEPC) | Sigma-Aldrich | 159220-5G | for treatment of water used in preparation of all solutions |
| dimethylpimelimidate (DMP) | Sigma-Aldrich | 80490-5G | for cross-linking antibody to Sepharose beads |
| electrophoresis cell | BioRad Laboratories, Inc | Mini-Protean II | for SDS-PAGE separation of proteins |
| ethanolamine | Sigma-Aldrich | 411000-100ml | for blocking after the cross-linking reaction |
| gel electrophoresis system | Hoefer, Inc | HSI SE 500 Series | for separating snRNAs by gel electrophoresis |
| gel slab dryer | BioRad | Model 224 | for drying gel slabs for autoradiography |
| Hybond ECL membrane | GE Healthcare | RPN3032D (0.2 μm; 30 cm x 3 m) | for immunoblotting of proteins on membrane |
| microdialyzer (12 x 100 μl sample capacity) | Pierce | Microdialyzer System 100 | for exchanging the buffer of nuclear extract |
| microdialyzer membranes (8K cutoff) | Pierce | 66310 | for exchanging the buffer of nuclear extract |
| non-fat dry milk | Spartan Stores | Spartan Instant Non-fat Dry Milk | |
| Protein A Sepharose CL-4B | Millipore-Sigma | GE 17-0780-01 | for coupling antibody to beads |
| Proteinase K | Millipore-Sigma | P2308-5mg | for stopping the splicing reaction to isolate the RNAs |
| RNasin | Promega | N2111 | for inhibiting ribonuclease activity |
| rocker/rotator | Lab Industries, Inc | Labquake Shaker 400-110 | for mixing protein solutions in coupling reactions and in immunoprecipitation |
| Safety-Solve | Research Products International Corp. | No. 111177 | scintillation counting cocktail for determination of radioactivity in splicing substrate |
| scintillation counter | Beckman Instruments | LS6000SC | scintillation counter for determination of radioactivity |
| speed vaccum concentrator | Savant | SVC 100H | for drying ethanol-precipitated RNA pellets |
| Transphor electrophoresis unit | Hoefer, Inc | Hoefer TE Series Transphor | for protein transfer from SDS-PAGE to blotting membrane |
Riferimenti
- Hoskins, A. A., Moore, M. J. The spliceosome: a flexible, reversible macromolecular machine. Trends In Biochemical Sciences. 37, 179-188 (2012).
- Choi, Y. D., Grabowski, P., Sharp, P. A., Dreyfuss, G. Heterogeneous nuclear ribonucleoproteins: role in RNA splicing. Science. 231, 1534-1539 (1986).
- Lerner, M., Steitz, J. A. Snurps and scyrps. Cell. 25, 298-300 (1981).
- Maniatis, T., Reed, R. The role of small nuclear ribonucleoprotein particles in pre-mRNA splicing. Nature. 325, 673-678 (1987).
- Hoskins, A. A., et al. Ordered and dynamic assembly of single spliceosomes. Science. 331, 1289-1295 (2011).
- Coppin, L., Leclerc, J., Vincent, A., Porchet, N., Pigny, P. Messenger RNA life-cycle in cancer: emerging role of conventional and non-conventional RNA-binding proteins. International Journal of Molecular Sciences. 19, 650-676 (2018).
- Dagher, S. F., Wang, J. L., Patterson, R. J. Identification of galectin-3 as a factor in pre-mRNA splicing. Proceedings of the National Academy of Sciences of the United States of America. 92, 1213-1217 (1995).
- Vyakarnam, A., Dagher, S. F., Wang, J. L., Patterson, R. J. Evidence for a role for galectin-1 in pre-mRNA splicing. Molecular and Cellular Biology. 17, 4730-4737 (1997).
- Wang, W., Park, J. W., Wang, J. L., Patterson, R. J. Immunoprecipitation of spliceosomal RNAs by antisera to galectin-1 and galectin-3. Nucleic Acids Research. 34, 5166-5174 (2006).
- Haudek, K. C., Voss, P. G., Locascio, L. E., Wang, J. L., Patterson, R. J. A mechanism for incorporation of galectin-3 into the spliceosome through its association with U1 snRNP. Biochimica. 48, 7705-7712 (2009).
- Fritsch, K., et al. Galectin-3 interacts with components of the nuclear ribonucleoprotein complex. BMC Cancer. 16, 502-511 (2016).
- Conway, G. C., Krainer, A. R., Spector, D. L., Roberts, R. J. Multiple splicing factors are released from endogenous complexes during in vitro pre-mRNA splicing. Molecular and Cellular Biology. 9, 5273-5280 (1989).
- Dery, K. J., Yean, S. L., Lin, R. J. Assembly and glycerol gradient isolation of yeast spliceosomes containing transcribed or synthetic U6 snRNA. Methods in Molecular Biology. 488, 41-63 (2008).
- Yoshimoto, R., Kataoka, N., Okawa, K., Ohno, M. Isolation and characterization of post-splicing lariat-intron complexes. Nucleic Acids Research. 37, 891-902 (2009).
- Malca, H., Shomron, N., Ast, G. The U1 snRNP base pairs with the 5′ splice site within a penta-snRNP complex. Molecular and Cellular Biology. 23, 3442-3455 (2003).
- Haudek, K. C., Voss, P. G., Wang, J. L., Patterson, R. J. A 10S galectin-3 – snRNP complex assembles into active spliceosomes. Nucleic Acids Research. 44, 6391-6397 (2016).
- Rappsilber, J., Ryder, U., Lamond, A. I., Mann, M. Large-scale proteomic analysis of the human spliceosome. Genome Research. 12, 1231-1245 (2002).
- Jurica, M. S., Moore, M. J. Capturing splicing complexes to study structure and mechanism. Methods. 28, 336-345 (2002).
- Patterson, R. J., Haudek, K. C., Voss, P. G., Wang, J. L. Examination of the role of galectins in pre-mRNA splicing. Methods in Molecular Biology. 1207, 431-449 (2015).
- Dignam, J. D., Lebovitz, R. M., Roeder, R. G. Accurate transcription initiation by RNA polymerase II in a soluble extract from isolated mammalian nuclei. Nucleic Acids Research. 11, 1475-1489 (1983).
- Agarwal, N., Sun, Q., Wang, S. Y., Wang, J. L. Carbohydrate-binding protein 35. I. Properties of the recombinant polypeptide and the individuality of the domains. Journal of Biological Chemistry. 268, 14932 (1993).
- Zillmann, M., Zapp, M. I., Berget, S. M. Gel electrophoretic isolation of splicing complexes containing U1 small nuclear ribonucleoprotein particles. Molecular and Cellular Biology. 8, 814-821 (1988).
- Barondes, S. H., et al. Galectins: a family of animal β-galactoside-binding proteins. Cell. 76, 597-598 (1994).
- Laing, J. G., Wang, J. L. Identification of carbohydrate binding protein 35 in heterogeneous nuclear ribonucleoprotein complex. Biochimica. 27, 5329-5334 (1988).
- Vyakarnam, A., Lenneman, A. J., Lakkides, K. M., Patterson, R. J., Wang, J. L. A comparative nuclear localization study of galectin-1 with other splicing components. Experimental Cell Research. 242, 419-428 (1998).
- Michaud, S., Reed, R. An ATP-independent complex commits pre-mRNA to the mammalian spliceosome assembly pathway. Genes & Development. 5, 2534-2546 (1991).
- Chiu, Y. -. F., et al. Cwc25 is a novel splicing factor required after Prp2 and Yju2 to facilitate the first catalytic reaction. Molecular and Cellular Biology. 29, 5671-5678 (2009).
- Krishnan, R., et al. Biased Brownian ratcheting leads to pre-mRNA remodeling and capture prior to first-step splicing. Nature Structural and Molecular Biology. 20, 1450-1457 (2013).
- Gray, R. M., et al. Distinct effects on splicing of two monoclonal antibodies directed against the amino-terminal domain of galectin-3. Archives of Biochemistry and Biophysics. 475, 100-108 (2008).

