Complementação da Atividade de Emenda por um Complexo Galectin-3 - U1 snRNP on Beads
Summary
Este artigo descreve os procedimentos experimentais para (a) esgotamento do U1 snRNP a partir de extratos nucleares, com perda concomitante de atividade de emenda; e (b) reconstituição da atividade de emenda no extrato empobrecido U1 por partículas de galectina-3 – U1 snRNP ligadas a contas covalentemente acopladas a anticorpos anti-galectina-3.
Abstract
Experimentos clássicos de esgotamento-reconstituição indicam que a galectina-3 é um fator de emenda necessário em extratos nucleares. O mecanismo de incorporação da galectina-3 na via de emenda é abordado neste artigo. A sedimentação de extratos nucleares de células HeLa em 12%-32% gradientes de glicerol produz frações enriquecidas em uma partícula endógena ~10S que contém galectina-3 e U1 snRNP. Descrevemos agora um protocolo para esgotar extratos nucleares do U1 snRNP com perda concomitante de atividade de emenda. A atividade de emenda no extrato empobrecido U1 pode ser reconstituída pela partícula de galectin-3 – U1 snRNP presa em contas de agarose covalentemente acopladas com anticorpos anti-galectina-3. Os resultados indicam que o complexo ternário pré-mRNA é um complexo E funcional que leva a intermediários e produtos da reação de emenda e que a galectina-3 entra no caminho de emenda através de sua associação com o U1 snRNP. O esquema de utilização de complexos de afinidade ou imuno-selecionados em contas para reconstituir a atividade de emenda em extratos esgotados de um fator de emenda específico pode ser geralmente aplicável a outros sistemas.
Introduction
A produção da maioria dos RNAs (mRNAs) do mensageiro eucariótico envolve a remoção de introns e a ligadura de exons em um processo nuclear chamado de emenda pré-mRNA1. Duas classes de complexos de proteína RNA (RNPs) direcionam o processamento do RNA pré-mensageiro em mRNA maduro através de complexos spliceossômicos. Uma classe, rnps pré-mensageiro nascente, é formada co-transcrição pela vinculação de proteínas heterogêneas nucleares RNP e outras proteínas de ligação de RNA, incluindo alguns membros da família SR, produzindo complexos hnRNP2. A segunda classe, rica em uracil, pequenas RNPs nucleares (U snRNPs com U1, U2, U4, U5 e U6 snRNAs) está associada com proteínas específicas e core u3,4. Os SNRNPs da U interagem de forma ordenada com regiões específicas de RNPs pré-mensageiros em um caminho de remodelação dinâmica à medida que os introns são extirpados e exons são ligados para produzir mRNPs5 maduros. Muitas proteínas nucleares adicionais participam desses eventos de processamento6.
Galectin-1 (Gal1) e galectin-3 (Gal3) são duas proteínas que são fatores necessários na via de emenda, como mostrado pelos estudos de esgotamento-reconstituição7,8. A remoção de ambas as galectinas de emendas extratos nucleares competentes (NE) abolia a montagem de emendas e a atividade de emenda em um passo inicial. A adição de uma galectina a um NE duplamente esgotado restaura ambas as atividades. Gal1 e Gal3 são componentes de emendas ativas, evidenciadas por imunoprecipitação específica de pré-mRNA, intermediários de emenda e mRNA maduro por antiserum específico para Gal1 ou Gal39. É importante ressaltar que a Gal3 associa-se ao snRNA u endógeno contendo partículas no NE fora da via de emenda, como mostrado pela precipitação de snRNPs por anti-Gal3 antisera10. Finalmente, o silenciamento de Gal3 em células HeLa altera padrões de emenda de numerosos genes11.
No NE pré-incubado para desmontar os spliceosomes pré-formados12, snRNPs são encontrados em múltiplos complexos sedimentando em gradientes de glicerol de 7S para maiores de 60S. Embora o fracionamento do gradiente de glicerol seja uma técnica comum para o isolamento de complexos e componentes spliceossômicos (ver referências13,14,15 por exemplo), ampliamos esse método caracterizando frações específicas usando imunoprecipitações de anticorpos. Um sedimento snRNP em 10S contém apenas u1 snRNA juntamente com Gal3. A imunoprecipitação da fração 10S com antisera específica para Gal3 ou U1 snRNP co-precipita tanto u1 quanto Gal3 indicando que algumas das mon partículas do U1 snRNP estão ligadas a Gal310. Como o U1 snRNP é o primeiro complexo que se liga ao pré-mRNP em conjuntos emendasomais1,5, esta etapa representa um potencial local de entrada para Gal3 na via de emenda. Com base nisso, mostramos que as monpartículas 10S Gal3-U1 snRNP ligadas à anti-Gal3 contendo contas restauradas para um NE esgotado U1 snRNP, estabelecendo este complexo como um mecanismo pelo qual Gal3 é recrutado para o caminho emendalomal16. Isso contrasta com as tentativas de isolar os emendas em estágios específicos na reação de emenda e catalogação dos fatores associados17,18. Em tais estudos, a presença de certos fatores em algum momento é apurada, mas não o mecanismo pelo qual foram carregados.
Havia descrito anteriormente em detalhes a preparação do NE, o substrato de emenda, a montagem da mistura de reação de emenda, e a análise de produtos em nossa documentação do papel das galectinas no splicing pré-mRNA19. Descrevemos agora os procedimentos experimentais para o fracionamento de extratos nucleares para obter uma fração enriquecida no complexo Gal3 – U1 snRNP e para a imuno-seleção deste último complexo para reconstituir a atividade de emenda em um extrato nuclear esgotado pelo U1.
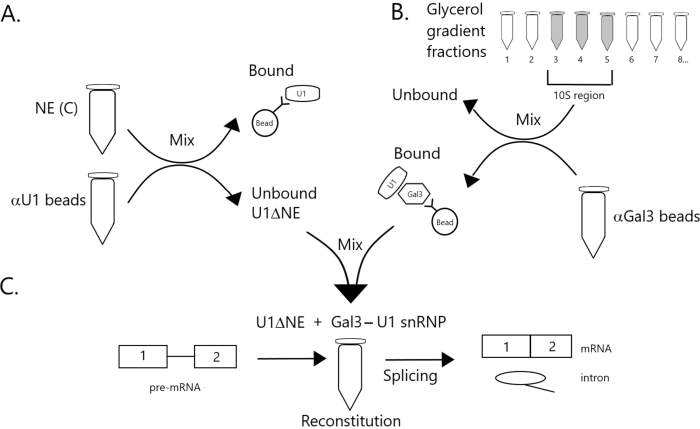
Figura 1: Diagrama esquemático ilustrando a complementação da atividade de emenda em extrato nuclear esgotado de U1 snRNP por um complexo de snRNP Gal3-U1 em contas. (A) NE em Buffer C (NE(C)) é incubado com contas de proteína A-Sepharose covalentemente acopladas com anti-U1 snRNP (contas αU1). A fração desvinculada está esgotada de U1 snRNP (U1ΔNE). (B) NE em Buffer D (NE(D)) é fracionado sobre um gradiente de glicerol de 12%-32% por ultracentrifugação. Frações correspondentes à região 10S (frações 3-5) são combinadas e misturadas com contas covalentemente acopladas com anticorpos anti-Gal3 (contas αGal3). O material ligado às contas contém uma monopartícula de SnRNP Gal3-U1. (C) O complexo de snRNP Gal3-U1 da Parte (B) é misturado com U1ΔNE da Parte (A) em um ensaio de emenda usando substrato minx pré-mRNA com 32P e os intermediários e produtos da reação de emenda são analisados por eletroforese gel e autoradiografia. Clique aqui para ver uma versão maior desta figura.
Protocol
Representative Results
Discussion
Este relatório fornece os detalhes experimentais que documentam um complexo Gal3 – U1 snRNP preso em contas revestidas anti-Gal3 pode se ligar ao substrato pré-mRNA e este complexo ternário pode restaurar a atividade de emenda a um NE er. . Os estudos de imunofluorescência precoce e fracionamento subcelular forneceram o indício inicial de uma associação de Gal3 com componentes da máquina de emenda: colocalização em manchas nucleares com polipeptídeos de núcleo Sm de snRNPs e serina e a…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Este trabalho foi apoiado pela National Science Foundation Grant MCB-0092919 e Michigan State University Intramural Research Grant 09-CDFP-2001 (para RJP) e pelos Institutos Nacionais de Saúde Grant GM-38740 e Michigan AgBioResearch Project MICL02455 (para JLW).
O substrato minx pré-mRNA usado nos ensaios de emenda foi um presente gentil da Dra.
Materials
| anti-U1 snRNP | The Binding Site | Hu ENA-RNP #33471 | human autoimmune serum specific for U1 snRNP |
| bottle top vacuum filter | Fisher Scientific | Corning 431153 (0.22 μm; PES 150 ml) | for filtering solutions containing Tris |
| centrifuge | International Equipment Company | IEC Model PR-6 | for pelletting Sepharose beads in immunoprecipitation |
| diethylpyrocarbonate (DEPC) | Sigma-Aldrich | 159220-5G | for treatment of water used in preparation of all solutions |
| dimethylpimelimidate (DMP) | Sigma-Aldrich | 80490-5G | for cross-linking antibody to Sepharose beads |
| electrophoresis cell | BioRad Laboratories, Inc | Mini-Protean II | for SDS-PAGE separation of proteins |
| ethanolamine | Sigma-Aldrich | 411000-100ml | for blocking after the cross-linking reaction |
| gel electrophoresis system | Hoefer, Inc | HSI SE 500 Series | for separating snRNAs by gel electrophoresis |
| gel slab dryer | BioRad | Model 224 | for drying gel slabs for autoradiography |
| Hybond ECL membrane | GE Healthcare | RPN3032D (0.2 μm; 30 cm x 3 m) | for immunoblotting of proteins on membrane |
| microdialyzer (12 x 100 μl sample capacity) | Pierce | Microdialyzer System 100 | for exchanging the buffer of nuclear extract |
| microdialyzer membranes (8K cutoff) | Pierce | 66310 | for exchanging the buffer of nuclear extract |
| non-fat dry milk | Spartan Stores | Spartan Instant Non-fat Dry Milk | |
| Protein A Sepharose CL-4B | Millipore-Sigma | GE 17-0780-01 | for coupling antibody to beads |
| Proteinase K | Millipore-Sigma | P2308-5mg | for stopping the splicing reaction to isolate the RNAs |
| RNasin | Promega | N2111 | for inhibiting ribonuclease activity |
| rocker/rotator | Lab Industries, Inc | Labquake Shaker 400-110 | for mixing protein solutions in coupling reactions and in immunoprecipitation |
| Safety-Solve | Research Products International Corp. | No. 111177 | scintillation counting cocktail for determination of radioactivity in splicing substrate |
| scintillation counter | Beckman Instruments | LS6000SC | scintillation counter for determination of radioactivity |
| speed vaccum concentrator | Savant | SVC 100H | for drying ethanol-precipitated RNA pellets |
| Transphor electrophoresis unit | Hoefer, Inc | Hoefer TE Series Transphor | for protein transfer from SDS-PAGE to blotting membrane |
Riferimenti
- Hoskins, A. A., Moore, M. J. The spliceosome: a flexible, reversible macromolecular machine. Trends In Biochemical Sciences. 37, 179-188 (2012).
- Choi, Y. D., Grabowski, P., Sharp, P. A., Dreyfuss, G. Heterogeneous nuclear ribonucleoproteins: role in RNA splicing. Science. 231, 1534-1539 (1986).
- Lerner, M., Steitz, J. A. Snurps and scyrps. Cell. 25, 298-300 (1981).
- Maniatis, T., Reed, R. The role of small nuclear ribonucleoprotein particles in pre-mRNA splicing. Nature. 325, 673-678 (1987).
- Hoskins, A. A., et al. Ordered and dynamic assembly of single spliceosomes. Science. 331, 1289-1295 (2011).
- Coppin, L., Leclerc, J., Vincent, A., Porchet, N., Pigny, P. Messenger RNA life-cycle in cancer: emerging role of conventional and non-conventional RNA-binding proteins. International Journal of Molecular Sciences. 19, 650-676 (2018).
- Dagher, S. F., Wang, J. L., Patterson, R. J. Identification of galectin-3 as a factor in pre-mRNA splicing. Proceedings of the National Academy of Sciences of the United States of America. 92, 1213-1217 (1995).
- Vyakarnam, A., Dagher, S. F., Wang, J. L., Patterson, R. J. Evidence for a role for galectin-1 in pre-mRNA splicing. Molecular and Cellular Biology. 17, 4730-4737 (1997).
- Wang, W., Park, J. W., Wang, J. L., Patterson, R. J. Immunoprecipitation of spliceosomal RNAs by antisera to galectin-1 and galectin-3. Nucleic Acids Research. 34, 5166-5174 (2006).
- Haudek, K. C., Voss, P. G., Locascio, L. E., Wang, J. L., Patterson, R. J. A mechanism for incorporation of galectin-3 into the spliceosome through its association with U1 snRNP. Biochimica. 48, 7705-7712 (2009).
- Fritsch, K., et al. Galectin-3 interacts with components of the nuclear ribonucleoprotein complex. BMC Cancer. 16, 502-511 (2016).
- Conway, G. C., Krainer, A. R., Spector, D. L., Roberts, R. J. Multiple splicing factors are released from endogenous complexes during in vitro pre-mRNA splicing. Molecular and Cellular Biology. 9, 5273-5280 (1989).
- Dery, K. J., Yean, S. L., Lin, R. J. Assembly and glycerol gradient isolation of yeast spliceosomes containing transcribed or synthetic U6 snRNA. Methods in Molecular Biology. 488, 41-63 (2008).
- Yoshimoto, R., Kataoka, N., Okawa, K., Ohno, M. Isolation and characterization of post-splicing lariat-intron complexes. Nucleic Acids Research. 37, 891-902 (2009).
- Malca, H., Shomron, N., Ast, G. The U1 snRNP base pairs with the 5′ splice site within a penta-snRNP complex. Molecular and Cellular Biology. 23, 3442-3455 (2003).
- Haudek, K. C., Voss, P. G., Wang, J. L., Patterson, R. J. A 10S galectin-3 – snRNP complex assembles into active spliceosomes. Nucleic Acids Research. 44, 6391-6397 (2016).
- Rappsilber, J., Ryder, U., Lamond, A. I., Mann, M. Large-scale proteomic analysis of the human spliceosome. Genome Research. 12, 1231-1245 (2002).
- Jurica, M. S., Moore, M. J. Capturing splicing complexes to study structure and mechanism. Methods. 28, 336-345 (2002).
- Patterson, R. J., Haudek, K. C., Voss, P. G., Wang, J. L. Examination of the role of galectins in pre-mRNA splicing. Methods in Molecular Biology. 1207, 431-449 (2015).
- Dignam, J. D., Lebovitz, R. M., Roeder, R. G. Accurate transcription initiation by RNA polymerase II in a soluble extract from isolated mammalian nuclei. Nucleic Acids Research. 11, 1475-1489 (1983).
- Agarwal, N., Sun, Q., Wang, S. Y., Wang, J. L. Carbohydrate-binding protein 35. I. Properties of the recombinant polypeptide and the individuality of the domains. Journal of Biological Chemistry. 268, 14932 (1993).
- Zillmann, M., Zapp, M. I., Berget, S. M. Gel electrophoretic isolation of splicing complexes containing U1 small nuclear ribonucleoprotein particles. Molecular and Cellular Biology. 8, 814-821 (1988).
- Barondes, S. H., et al. Galectins: a family of animal β-galactoside-binding proteins. Cell. 76, 597-598 (1994).
- Laing, J. G., Wang, J. L. Identification of carbohydrate binding protein 35 in heterogeneous nuclear ribonucleoprotein complex. Biochimica. 27, 5329-5334 (1988).
- Vyakarnam, A., Lenneman, A. J., Lakkides, K. M., Patterson, R. J., Wang, J. L. A comparative nuclear localization study of galectin-1 with other splicing components. Experimental Cell Research. 242, 419-428 (1998).
- Michaud, S., Reed, R. An ATP-independent complex commits pre-mRNA to the mammalian spliceosome assembly pathway. Genes & Development. 5, 2534-2546 (1991).
- Chiu, Y. -. F., et al. Cwc25 is a novel splicing factor required after Prp2 and Yju2 to facilitate the first catalytic reaction. Molecular and Cellular Biology. 29, 5671-5678 (2009).
- Krishnan, R., et al. Biased Brownian ratcheting leads to pre-mRNA remodeling and capture prior to first-step splicing. Nature Structural and Molecular Biology. 20, 1450-1457 (2013).
- Gray, R. M., et al. Distinct effects on splicing of two monoclonal antibodies directed against the amino-terminal domain of galectin-3. Archives of Biochemistry and Biophysics. 475, 100-108 (2008).

