Комплементация сплайсинговой активности комплексом Галектин-3 - U1 snRNP на бусинах
Summary
В данной статье описаны экспериментальные процедуры для (а) истощения U1 snRNP из ядерных экстрактов с сопутствующей потерей сплайсинговой активности; и (b) восстановление сплайсинговой активности в U1-обедненном экстракте частицами snRNP галектина-3-U1, связанными с ковалентно связанными с анти-галектин-3 антителами.
Abstract
Классические эксперименты по истощению-восстановлению показывают, что галектин-3 является обязательным фактором сплайсинга в ядерных экстрактах. Механизм включения галектина-3 в сплайсинговый путь рассматривается в данной работе. Осаждение клеточных ядерных экстрактов HeLa на 12%-32% градиентах глицерина дает фракции, обогащенные эндогенной частицей ~10S, содержащей галектин-3 и U1 snRNP. Теперь мы описываем протокол для истощения ядерных экстрактов U1 snRNP с сопутствующей потерей сплайсинговой активности. Сплайсинговая активность в U1-истощенном экстракте может быть восстановлена частицей snRNP галектина-3 – U1, захваченной на бусинах агарозы, ковалентно связанной с антителами против галектина-3. Результаты показывают, что троичный комплекс галектин-3 – U1 snRNP – пре-мРНК является функциональным комплексом Е, приводящим к промежуточным продуктам и продуктам реакции сплайсинга, и что галектин-3 попадает в путь сплайсинга через его ассоциацию с U1 snRNP. Схема использования комплексов аффинно- или иммуно-, выбранных на шариках, для восстановления сплайсинговой активности в экстрактах, обедненных специфическим фактором сплайсинга, может быть в целом применима к другим системам.
Introduction
Производство большинства эукариотических мессенджерных РНК (мРНК) включает удаление интронов и лигирование экзонов в ядерном процессе, называемом сплайсингом премрнК1. Два класса РНК-белковых комплексов (RNP) направляют обработку пре-мессенджерной РНК в зрелую мРНК через сплайсеосомальные комплексы. Один класс, зарождающиеся предмаршированные RNP, образуется котранскрипционно путем связывания гетерогенных ядерных белков RNP и других РНК-связывающих белков, включая некоторых членов семейства SR, давая комплексы hnRNP2. Второй класс, богатый урацилом малые ядерные RNP (U snRNP с U1, U2, U4, U5 и U6 snRNAs) связан с U-специфическими и основными белками3,4. U snRNP взаимодействуют упорядоченным образом с определенными областями предмарш-мессенджерных RNP в динамическом пути ремоделирования, поскольку интроны иссекаются, а экзоны лигируются для получения зрелых mRNPs5. Многие дополнительные ядерные белки участвуют в этих событиях обработки6.
Галектин-1 (Gal1) и галектин-3 (Gal3) являются двумя белками, которые являются необходимыми факторами в пути сплайсинга, как показано исследованиями истощения-восстановления7,8. Удаление обоих галектинов из сращивания компетентных ядерных экстрактов (NE) отменяет сплайсеосомную сборку и сплайсинговую деятельность на ранней стадии. Добавление любого галектина к такому вдвойне истощенному NE восстанавливает обе активности. Gal1 и Gal3 являются компонентами активных сплайсеосом, о чем свидетельствует специфическая иммунопреципитация пре-мРНК, сплайсинговых промежуточных продуктов и зрелой мРНК антисывороткой, специфичной для Gal1 или Gal39. Важно отметить, что Gal3 ассоциируется с эндогенной U snRNA, содержащей частицы в NE вне пути сплайсинга, как показано осаждением snRNP анти-Gal3 antisera10. Наконец, глушение Gal3 в клетках HeLa изменяет паттерны сплайсинга многочисленных генов11.
В NE, предварительно инкубированном для разборки предварительно сформированных сплайсеосом12, snRNP обнаруживаются в нескольких комплексах, осажденных в градиентах глицерина от 7S до более 60S. Хотя фракционирование градиента глицерина является распространенным методом выделения сплайсеосомальных комплексов и компонентов (см., например, ссылки13,14,15), мы расширили этот метод, охарактеризовав специфические фракции с использованием иммунопреципитаций антител. SnRNP осадок при 10S содержит только U1 snRNA вместе с Gal3. Иммунопреципитация фракции 10S с антисыворотки, специфичной для Gal3 или U1 snRNP, осаждает как U1, так и Gal3, указывая на то, что некоторые из моночастиц U1 snRNP связаны с Gal310. Поскольку U1 snRNP является первым комплексом, который связывается с pre-mRNP в сплайсеосомальной сборке1,5, этот шаг представляет собой потенциальный входной участок для Gal3 в путь сплайсинга. На этой основе мы показали, что моночастицы 10S Gal3-U1 snRNP, связанные с бусинами, содержащими анти-Gal3, восстанавливают активность сплайсинга к U1 snRNP истощенной NE, устанавливая этот комплекс как один механизм, с помощью которого Gal3 набирается в сплайсеосомальный путь16. Это контрастирует с попытками выделения сплайсеосом на определенных этапах реакции сплайсинга и каталогизации связанных с ними факторов17,18. В таких исследованиях констатируется наличие определенных факторов в какой-то момент времени, но не механизм, с помощью которого они были загружены.
Ранее мы подробно описали подготовку NE, сплайсинговую подложку, сборку реакционной смеси сплайсинга и анализ продуктов в нашей документации о роли галектинов в сплайсинге премрнК19. Теперь описаны экспериментальные процедуры фракционирования ядерных экстрактов для получения фракции, обогащенной комплексом SnRNP Gal3 – U1, и иммуноотбора последнего комплекса для восстановления сплайсинговой активности в ядерном экстракте, обедненном U1.
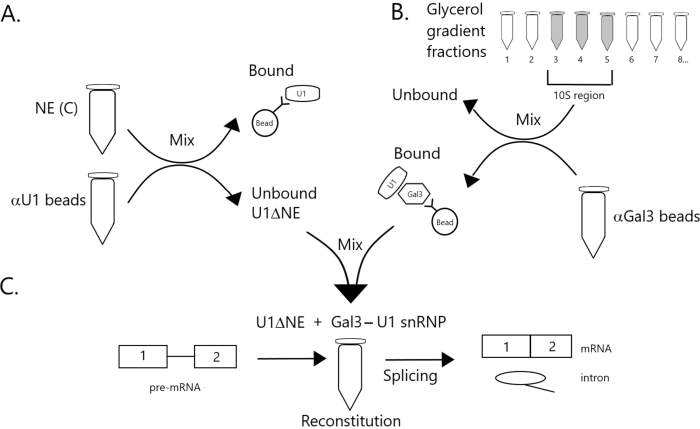
Рисунок 1: Принципиальная диаграмма, иллюстрирующая дополнение сплайсинговой активности в ядерном экстракте, обедненном U1 snRNP комплексом Gal3-U1 snRNP на шариках. (A) NE в буфере C (NE(C)) инкубируют с белком A-Sepharose бусинами, ковалентно связанными с анти-U1 snRNP (бусины αU1). Несвязанная фракция обедняется U1 snRNP (U1ΔNE). (B) NE в буфере D (NE(D)) фракционируют по 12%-32% градиенту глицерина путем ультрацентрифугирования. Фракции, соответствующие области 10S (фракции 3-5), объединяют и смешивают с бусинами, ковалентно связанными с антителами против Gal3 (шарики αGal3). Материал, связанный с шариками, содержит моночастицу Gal3-U1 snRNP. (C) Комплекс SnRNP Gal3-U1 из части (B) смешивают с U1ΔNE из части (A) в сплайсинговом анализе с использованием 32P-меченого субстрата MINX pre-mRNA, а промежуточные продукты и продукты реакции сплайсинга анализируют с помощью гелевого электрофореза и ауторадиографии. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Protocol
Representative Results
Discussion
В этом отчете представлены экспериментальные детали, которые документируют комплекс Gal3 – U1 snRNP, захваченный на бусинах, покрытых анти-Gal3, может связываться с субстратом пре-мРНК, и этот троичный комплекс может восстанавливать активность сплайсинга к обедненному U1 snRNP NE. Gal3 является одни?…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Эта работа была поддержана грантом Национального научного фонда MCB-0092919 и грантом Мичиганского государственного университета 09-CDFP-2001 (для RJP) и грантом Национальных институтов здравоохранения GM-38740 и Мичиганским agBioResearch Project MICL02455 (для JLW).
Субстрат MINX пре-мРНК, используемый в анализах сплайсинга, был добрым подарком от доктора Сьюзан Бергет (Медицинский колледж Бейлора, Хьюстон, Техас, США).
Materials
| anti-U1 snRNP | The Binding Site | Hu ENA-RNP #33471 | human autoimmune serum specific for U1 snRNP |
| bottle top vacuum filter | Fisher Scientific | Corning 431153 (0.22 μm; PES 150 ml) | for filtering solutions containing Tris |
| centrifuge | International Equipment Company | IEC Model PR-6 | for pelletting Sepharose beads in immunoprecipitation |
| diethylpyrocarbonate (DEPC) | Sigma-Aldrich | 159220-5G | for treatment of water used in preparation of all solutions |
| dimethylpimelimidate (DMP) | Sigma-Aldrich | 80490-5G | for cross-linking antibody to Sepharose beads |
| electrophoresis cell | BioRad Laboratories, Inc | Mini-Protean II | for SDS-PAGE separation of proteins |
| ethanolamine | Sigma-Aldrich | 411000-100ml | for blocking after the cross-linking reaction |
| gel electrophoresis system | Hoefer, Inc | HSI SE 500 Series | for separating snRNAs by gel electrophoresis |
| gel slab dryer | BioRad | Model 224 | for drying gel slabs for autoradiography |
| Hybond ECL membrane | GE Healthcare | RPN3032D (0.2 μm; 30 cm x 3 m) | for immunoblotting of proteins on membrane |
| microdialyzer (12 x 100 μl sample capacity) | Pierce | Microdialyzer System 100 | for exchanging the buffer of nuclear extract |
| microdialyzer membranes (8K cutoff) | Pierce | 66310 | for exchanging the buffer of nuclear extract |
| non-fat dry milk | Spartan Stores | Spartan Instant Non-fat Dry Milk | |
| Protein A Sepharose CL-4B | Millipore-Sigma | GE 17-0780-01 | for coupling antibody to beads |
| Proteinase K | Millipore-Sigma | P2308-5mg | for stopping the splicing reaction to isolate the RNAs |
| RNasin | Promega | N2111 | for inhibiting ribonuclease activity |
| rocker/rotator | Lab Industries, Inc | Labquake Shaker 400-110 | for mixing protein solutions in coupling reactions and in immunoprecipitation |
| Safety-Solve | Research Products International Corp. | No. 111177 | scintillation counting cocktail for determination of radioactivity in splicing substrate |
| scintillation counter | Beckman Instruments | LS6000SC | scintillation counter for determination of radioactivity |
| speed vaccum concentrator | Savant | SVC 100H | for drying ethanol-precipitated RNA pellets |
| Transphor electrophoresis unit | Hoefer, Inc | Hoefer TE Series Transphor | for protein transfer from SDS-PAGE to blotting membrane |
Riferimenti
- Hoskins, A. A., Moore, M. J. The spliceosome: a flexible, reversible macromolecular machine. Trends In Biochemical Sciences. 37, 179-188 (2012).
- Choi, Y. D., Grabowski, P., Sharp, P. A., Dreyfuss, G. Heterogeneous nuclear ribonucleoproteins: role in RNA splicing. Science. 231, 1534-1539 (1986).
- Lerner, M., Steitz, J. A. Snurps and scyrps. Cell. 25, 298-300 (1981).
- Maniatis, T., Reed, R. The role of small nuclear ribonucleoprotein particles in pre-mRNA splicing. Nature. 325, 673-678 (1987).
- Hoskins, A. A., et al. Ordered and dynamic assembly of single spliceosomes. Science. 331, 1289-1295 (2011).
- Coppin, L., Leclerc, J., Vincent, A., Porchet, N., Pigny, P. Messenger RNA life-cycle in cancer: emerging role of conventional and non-conventional RNA-binding proteins. International Journal of Molecular Sciences. 19, 650-676 (2018).
- Dagher, S. F., Wang, J. L., Patterson, R. J. Identification of galectin-3 as a factor in pre-mRNA splicing. Proceedings of the National Academy of Sciences of the United States of America. 92, 1213-1217 (1995).
- Vyakarnam, A., Dagher, S. F., Wang, J. L., Patterson, R. J. Evidence for a role for galectin-1 in pre-mRNA splicing. Molecular and Cellular Biology. 17, 4730-4737 (1997).
- Wang, W., Park, J. W., Wang, J. L., Patterson, R. J. Immunoprecipitation of spliceosomal RNAs by antisera to galectin-1 and galectin-3. Nucleic Acids Research. 34, 5166-5174 (2006).
- Haudek, K. C., Voss, P. G., Locascio, L. E., Wang, J. L., Patterson, R. J. A mechanism for incorporation of galectin-3 into the spliceosome through its association with U1 snRNP. Biochimica. 48, 7705-7712 (2009).
- Fritsch, K., et al. Galectin-3 interacts with components of the nuclear ribonucleoprotein complex. BMC Cancer. 16, 502-511 (2016).
- Conway, G. C., Krainer, A. R., Spector, D. L., Roberts, R. J. Multiple splicing factors are released from endogenous complexes during in vitro pre-mRNA splicing. Molecular and Cellular Biology. 9, 5273-5280 (1989).
- Dery, K. J., Yean, S. L., Lin, R. J. Assembly and glycerol gradient isolation of yeast spliceosomes containing transcribed or synthetic U6 snRNA. Methods in Molecular Biology. 488, 41-63 (2008).
- Yoshimoto, R., Kataoka, N., Okawa, K., Ohno, M. Isolation and characterization of post-splicing lariat-intron complexes. Nucleic Acids Research. 37, 891-902 (2009).
- Malca, H., Shomron, N., Ast, G. The U1 snRNP base pairs with the 5′ splice site within a penta-snRNP complex. Molecular and Cellular Biology. 23, 3442-3455 (2003).
- Haudek, K. C., Voss, P. G., Wang, J. L., Patterson, R. J. A 10S galectin-3 – snRNP complex assembles into active spliceosomes. Nucleic Acids Research. 44, 6391-6397 (2016).
- Rappsilber, J., Ryder, U., Lamond, A. I., Mann, M. Large-scale proteomic analysis of the human spliceosome. Genome Research. 12, 1231-1245 (2002).
- Jurica, M. S., Moore, M. J. Capturing splicing complexes to study structure and mechanism. Methods. 28, 336-345 (2002).
- Patterson, R. J., Haudek, K. C., Voss, P. G., Wang, J. L. Examination of the role of galectins in pre-mRNA splicing. Methods in Molecular Biology. 1207, 431-449 (2015).
- Dignam, J. D., Lebovitz, R. M., Roeder, R. G. Accurate transcription initiation by RNA polymerase II in a soluble extract from isolated mammalian nuclei. Nucleic Acids Research. 11, 1475-1489 (1983).
- Agarwal, N., Sun, Q., Wang, S. Y., Wang, J. L. Carbohydrate-binding protein 35. I. Properties of the recombinant polypeptide and the individuality of the domains. Journal of Biological Chemistry. 268, 14932 (1993).
- Zillmann, M., Zapp, M. I., Berget, S. M. Gel electrophoretic isolation of splicing complexes containing U1 small nuclear ribonucleoprotein particles. Molecular and Cellular Biology. 8, 814-821 (1988).
- Barondes, S. H., et al. Galectins: a family of animal β-galactoside-binding proteins. Cell. 76, 597-598 (1994).
- Laing, J. G., Wang, J. L. Identification of carbohydrate binding protein 35 in heterogeneous nuclear ribonucleoprotein complex. Biochimica. 27, 5329-5334 (1988).
- Vyakarnam, A., Lenneman, A. J., Lakkides, K. M., Patterson, R. J., Wang, J. L. A comparative nuclear localization study of galectin-1 with other splicing components. Experimental Cell Research. 242, 419-428 (1998).
- Michaud, S., Reed, R. An ATP-independent complex commits pre-mRNA to the mammalian spliceosome assembly pathway. Genes & Development. 5, 2534-2546 (1991).
- Chiu, Y. -. F., et al. Cwc25 is a novel splicing factor required after Prp2 and Yju2 to facilitate the first catalytic reaction. Molecular and Cellular Biology. 29, 5671-5678 (2009).
- Krishnan, R., et al. Biased Brownian ratcheting leads to pre-mRNA remodeling and capture prior to first-step splicing. Nature Structural and Molecular Biology. 20, 1450-1457 (2013).
- Gray, R. M., et al. Distinct effects on splicing of two monoclonal antibodies directed against the amino-terminal domain of galectin-3. Archives of Biochemistry and Biophysics. 475, 100-108 (2008).

