Komplementering av splitsningsaktivitet av ett Galectin-3 - U1 snRNP-komplex på pärlor
Summary
Denna artikel beskriver de experimentella förfarandena för a) utarmning av U1 snRNP från kärnextrakt, med samtidig förlust av splitsning aktivitet; och b) rekonstitution av splitsning aktivitet i U1-utarmat extrakt av galectin-3 – U1 snRNP partiklar bundna till pärlor kovalent i kombination med anti-galectin-3 antikroppar.
Abstract
Klassiska utarmning-rekonstitution experiment visar att galectin-3 är en obligatorisk splitsning faktor i kärnämne extrakt. Mekanismen för införlivande av galectin-3 i skarvningsvägen behandlas i detta dokument. Sedimentering av HeLa-cellskärnextrakt på 12-32% glycerol gradienter ger fraktioner berikade i en endogen ~ 10S partikel som innehåller galectin-3 och U1 snRNP. Vi beskriver nu ett protokoll för att tömma kärnämne extrakt av U1 snRNP med samtidig förlust av splitsning verksamhet. Splitsning aktivitet i U1-utarmat extrakt kan rekonstitueras av galectin-3 – U1 snRNP partikel fångas på agarose pärlor kovalent i kombination med anti-galectin-3 antikroppar. Resultaten indikerar att galectin-3 – U1 snRNP – pre-mRNA ternary komplex är ett funktionellt E-komplex som leder till intermediärer och produkter av splitsningsreaktionen och att galectin-3 går in i skarvningsvägen genom sin associering med U1 snRNP. Systemet för användning av komplex affinitet- eller immunvalt på pärlor för rekonstituerande splitsning aktivitet i extrakt uttömda av en specifik splitsningsfaktor kan vara allmänt tillämpliga på andra system.
Introduction
Produktion av mest eukaryotic budbärareRNA (mRNAs) innebär borttagning av introner och ligation av exons i en kärn- processaa som benämns pre-mRNA-skarvning1. Två klasser av RNA-proteinkomplex (RNPs) styr bearbetningen av pre-messenger RNA till mogna mRNA via spliceosomal komplex. En klass, gryende pre-messenger RNPs, bildas co-transcriptionally genom bindning av heterogena nukleära RNP-proteiner och andra RNA-bindande proteiner, inklusive vissa medlemmar av SR-familjen, vilket ger hnRNP-komplex2. Den andra klassen, uracil-rika små nukleära RNPs (U snRNPs med U1, U2, U4, U5 och U6 snRNAs) är associerad med U-specifika och kärnproteiner3,4. U snRNPs interagerar på ett ordnat sätt med specifika regioner av pre-messenger RNPs i en dynamisk ombyggnadsväg som introner är strukna och exons är ligated för att producera mogna MRNPs5. Många ytterligare nukleära proteiner deltar i dessa bearbetningsevenemang6.
Galectin-1 (Gal1) och galectin-3 (Gal3) är två proteiner som krävs faktorer i skarvningsvägen som framgår av utarmnings-rekonstitutionsstudier7,8. Avlägsnande av båda galectins från skarvning kompetent kärnämne extrakt (NE) avskaffar skareosome montering och splitsning verksamhet i ett tidigt steg. Tillägg av antingen galectin till ett så dubbelt utarmat NE återställer båda aktiviteterna. Gal1 och Gal3 är komponenter i aktiva splitseomer, vilket framgår av specifik immunprecipitation av pre-mRNA, skarvning av intermediärer och mogna mRNA med antiserumspecifikt för antingen Gal1 eller Gal39. Viktigt är att Gal3 associerar med endogent U snRNA som innehåller partiklar i NE utanför skarvningsvägen som framgår av utfällning av snRNPs av anti-Gal3 antisera10. Slutligen, ljuddämpning av Gal3 i HeLa celler förändrar skarvning mönster av många gener11.
I NE pre-incubated för att demontera förformade spliteosomer12, snRNPs finns i flera komplex sedimentering i glycerol gradienter från 7S till mer än 60S. Även om glycerol gradient fraktionering är en vanlig teknik för isolering av spliceosomal komplex och komponenter (se referenser13,14,15 till exempel), har vi utvidgat denna metod genom att karakterisera specifika fraktioner med hjälp av antikroppar immunprecipitations. En snRNP sedimentering vid 10S innehåller endast U1 snRNA tillsammans med Gal3. Immunoprecipitation av 10S-fraktionen med antisera specifik för Gal3 eller U1 snRNP co-fäller ut både U1 och Gal3 som indikerar att några av U1 snRNP-monopartiklarna är bundna till Gal310. Eftersom U1 snRNP är det första komplexet som binder till pre-mRNP i spliteosomal sammansättning1,5, representerar detta steg en potentiell ingångsplats för Gal3 i skarvningsvägen. På denna grund visade vi att 10S Gal3-U1 snRNP monopartiklar bundna till anti-Gal3 som innehåller pärlor återställde splitsning aktivitet till en U1 snRNP utarmade NE, upprätta detta komplex som en mekanism genom vilken Gal3 rekryteras till den spliteosomala vägen16. Detta står i kontrast till försök att isolera splitsosomer i specifika stadier i skarvningsreaktionen och katalogisering av de associerade faktorerna17,18. I sådana studier fastställs förekomsten av vissa faktorer vid någon tidpunkt men inte den mekanism genom vilken de laddades.
Vi hade tidigare beskrivit i detalj beredningen av NE, skarvning substratet, monteringen av skarvning reaktion blandning och analys av produkter i vår dokumentation av galectins roll i pre-mRNA splitsning19. Vi beskriver nu de experimentella förfarandena för fraktionering av kärnextrakt för att erhålla en bråkdel berikad i Gal3 – U1 snRNP komplex och för immuno-urval av det senare komplexet att rekonstituerande splitsning verksamhet i en U1-utarmade kärnämne extrakt.
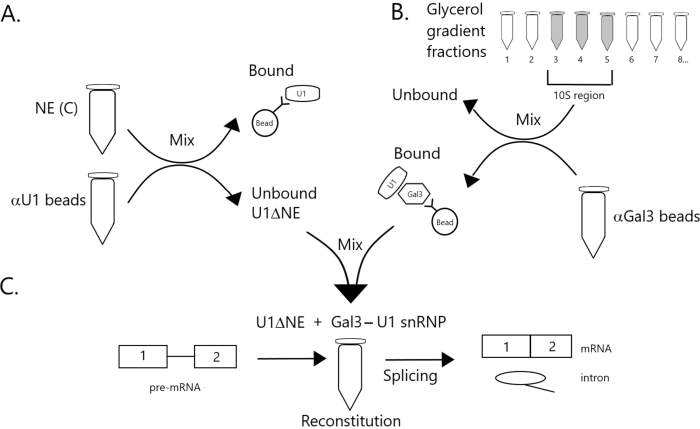
Figur 1: Schematiskt diagram som illustrerar komplementering av skarvningsaktivitet i kärnextrakt som uttömts av U1 snRNP av ett Gal3-U1 snRNP-komplex på pärlor. Den obundna fraktionen är uttömd av U1 snRNP (U1ΔNE). (B) NE i buffert D (NE(D)) är fraktionerad över en 12-32% glycerol gradient genom ultracentrifugation. Fraktioner som motsvarar 10S-regionen (fraktioner 3-5) kombineras och blandas med pärlor kovalent i kombination med anti-Gal3-antikroppar (αGal3 pärlor). Materialet som är bundet till pärlorna innehåller en Gal3-U1 snRNP monopartikel. C) Gal3-U1 snRNP-komplexet från del B blandas med U1ΔNE från del A i en skarvningsanalys med 32P-märkt MINX pre-mRNA-substrat och intermediärerna och produkterna i skarvningsreaktionen analyseras med gelelektrofores och autoradiografi. Klicka här för att se en större version av den här figuren.
Protocol
Representative Results
Discussion
Denna rapport ger de experimentella detaljerna som dokumenterar ett Gal3 – U1 snRNP-komplex fångat på anti-Gal3-belagda pärlor kan binda till pre-mRNA-substrat och detta ternarykomplex kan återställa splitsningsaktiviteten till en U1 snRNP-utarmad NE. Gal3 är en medlem av en familj av proteiner som ursprungligen isolerades på grundval av dess galaktosspecifika kolhydratbindningsaktivitet23 . Tidiga immunofluorescens- och subcellulära fraktioneringsstudier gav den första antydan till en as…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Detta arbete har stöttats av National Science Foundation Grant MCB-0092919 och Michigan State University Intramural Research Grant 09-CDFP-2001 (till RJP) och av National Institutes of Health Grant GM-38740 och Michigan AgBioResearch Project MICL02455 (till JLW).
MINX pre-mRNA-substrat som användes i skarvningsanalyserna var en snäll gåva från Dr. Susan Berget (Baylor College of Medicine, Houston, TX, USA).
Materials
| anti-U1 snRNP | The Binding Site | Hu ENA-RNP #33471 | human autoimmune serum specific for U1 snRNP |
| bottle top vacuum filter | Fisher Scientific | Corning 431153 (0.22 μm; PES 150 ml) | for filtering solutions containing Tris |
| centrifuge | International Equipment Company | IEC Model PR-6 | for pelletting Sepharose beads in immunoprecipitation |
| diethylpyrocarbonate (DEPC) | Sigma-Aldrich | 159220-5G | for treatment of water used in preparation of all solutions |
| dimethylpimelimidate (DMP) | Sigma-Aldrich | 80490-5G | for cross-linking antibody to Sepharose beads |
| electrophoresis cell | BioRad Laboratories, Inc | Mini-Protean II | for SDS-PAGE separation of proteins |
| ethanolamine | Sigma-Aldrich | 411000-100ml | for blocking after the cross-linking reaction |
| gel electrophoresis system | Hoefer, Inc | HSI SE 500 Series | for separating snRNAs by gel electrophoresis |
| gel slab dryer | BioRad | Model 224 | for drying gel slabs for autoradiography |
| Hybond ECL membrane | GE Healthcare | RPN3032D (0.2 μm; 30 cm x 3 m) | for immunoblotting of proteins on membrane |
| microdialyzer (12 x 100 μl sample capacity) | Pierce | Microdialyzer System 100 | for exchanging the buffer of nuclear extract |
| microdialyzer membranes (8K cutoff) | Pierce | 66310 | for exchanging the buffer of nuclear extract |
| non-fat dry milk | Spartan Stores | Spartan Instant Non-fat Dry Milk | |
| Protein A Sepharose CL-4B | Millipore-Sigma | GE 17-0780-01 | for coupling antibody to beads |
| Proteinase K | Millipore-Sigma | P2308-5mg | for stopping the splicing reaction to isolate the RNAs |
| RNasin | Promega | N2111 | for inhibiting ribonuclease activity |
| rocker/rotator | Lab Industries, Inc | Labquake Shaker 400-110 | for mixing protein solutions in coupling reactions and in immunoprecipitation |
| Safety-Solve | Research Products International Corp. | No. 111177 | scintillation counting cocktail for determination of radioactivity in splicing substrate |
| scintillation counter | Beckman Instruments | LS6000SC | scintillation counter for determination of radioactivity |
| speed vaccum concentrator | Savant | SVC 100H | for drying ethanol-precipitated RNA pellets |
| Transphor electrophoresis unit | Hoefer, Inc | Hoefer TE Series Transphor | for protein transfer from SDS-PAGE to blotting membrane |
Riferimenti
- Hoskins, A. A., Moore, M. J. The spliceosome: a flexible, reversible macromolecular machine. Trends In Biochemical Sciences. 37, 179-188 (2012).
- Choi, Y. D., Grabowski, P., Sharp, P. A., Dreyfuss, G. Heterogeneous nuclear ribonucleoproteins: role in RNA splicing. Science. 231, 1534-1539 (1986).
- Lerner, M., Steitz, J. A. Snurps and scyrps. Cell. 25, 298-300 (1981).
- Maniatis, T., Reed, R. The role of small nuclear ribonucleoprotein particles in pre-mRNA splicing. Nature. 325, 673-678 (1987).
- Hoskins, A. A., et al. Ordered and dynamic assembly of single spliceosomes. Science. 331, 1289-1295 (2011).
- Coppin, L., Leclerc, J., Vincent, A., Porchet, N., Pigny, P. Messenger RNA life-cycle in cancer: emerging role of conventional and non-conventional RNA-binding proteins. International Journal of Molecular Sciences. 19, 650-676 (2018).
- Dagher, S. F., Wang, J. L., Patterson, R. J. Identification of galectin-3 as a factor in pre-mRNA splicing. Proceedings of the National Academy of Sciences of the United States of America. 92, 1213-1217 (1995).
- Vyakarnam, A., Dagher, S. F., Wang, J. L., Patterson, R. J. Evidence for a role for galectin-1 in pre-mRNA splicing. Molecular and Cellular Biology. 17, 4730-4737 (1997).
- Wang, W., Park, J. W., Wang, J. L., Patterson, R. J. Immunoprecipitation of spliceosomal RNAs by antisera to galectin-1 and galectin-3. Nucleic Acids Research. 34, 5166-5174 (2006).
- Haudek, K. C., Voss, P. G., Locascio, L. E., Wang, J. L., Patterson, R. J. A mechanism for incorporation of galectin-3 into the spliceosome through its association with U1 snRNP. Biochimica. 48, 7705-7712 (2009).
- Fritsch, K., et al. Galectin-3 interacts with components of the nuclear ribonucleoprotein complex. BMC Cancer. 16, 502-511 (2016).
- Conway, G. C., Krainer, A. R., Spector, D. L., Roberts, R. J. Multiple splicing factors are released from endogenous complexes during in vitro pre-mRNA splicing. Molecular and Cellular Biology. 9, 5273-5280 (1989).
- Dery, K. J., Yean, S. L., Lin, R. J. Assembly and glycerol gradient isolation of yeast spliceosomes containing transcribed or synthetic U6 snRNA. Methods in Molecular Biology. 488, 41-63 (2008).
- Yoshimoto, R., Kataoka, N., Okawa, K., Ohno, M. Isolation and characterization of post-splicing lariat-intron complexes. Nucleic Acids Research. 37, 891-902 (2009).
- Malca, H., Shomron, N., Ast, G. The U1 snRNP base pairs with the 5′ splice site within a penta-snRNP complex. Molecular and Cellular Biology. 23, 3442-3455 (2003).
- Haudek, K. C., Voss, P. G., Wang, J. L., Patterson, R. J. A 10S galectin-3 – snRNP complex assembles into active spliceosomes. Nucleic Acids Research. 44, 6391-6397 (2016).
- Rappsilber, J., Ryder, U., Lamond, A. I., Mann, M. Large-scale proteomic analysis of the human spliceosome. Genome Research. 12, 1231-1245 (2002).
- Jurica, M. S., Moore, M. J. Capturing splicing complexes to study structure and mechanism. Methods. 28, 336-345 (2002).
- Patterson, R. J., Haudek, K. C., Voss, P. G., Wang, J. L. Examination of the role of galectins in pre-mRNA splicing. Methods in Molecular Biology. 1207, 431-449 (2015).
- Dignam, J. D., Lebovitz, R. M., Roeder, R. G. Accurate transcription initiation by RNA polymerase II in a soluble extract from isolated mammalian nuclei. Nucleic Acids Research. 11, 1475-1489 (1983).
- Agarwal, N., Sun, Q., Wang, S. Y., Wang, J. L. Carbohydrate-binding protein 35. I. Properties of the recombinant polypeptide and the individuality of the domains. Journal of Biological Chemistry. 268, 14932 (1993).
- Zillmann, M., Zapp, M. I., Berget, S. M. Gel electrophoretic isolation of splicing complexes containing U1 small nuclear ribonucleoprotein particles. Molecular and Cellular Biology. 8, 814-821 (1988).
- Barondes, S. H., et al. Galectins: a family of animal β-galactoside-binding proteins. Cell. 76, 597-598 (1994).
- Laing, J. G., Wang, J. L. Identification of carbohydrate binding protein 35 in heterogeneous nuclear ribonucleoprotein complex. Biochimica. 27, 5329-5334 (1988).
- Vyakarnam, A., Lenneman, A. J., Lakkides, K. M., Patterson, R. J., Wang, J. L. A comparative nuclear localization study of galectin-1 with other splicing components. Experimental Cell Research. 242, 419-428 (1998).
- Michaud, S., Reed, R. An ATP-independent complex commits pre-mRNA to the mammalian spliceosome assembly pathway. Genes & Development. 5, 2534-2546 (1991).
- Chiu, Y. -. F., et al. Cwc25 is a novel splicing factor required after Prp2 and Yju2 to facilitate the first catalytic reaction. Molecular and Cellular Biology. 29, 5671-5678 (2009).
- Krishnan, R., et al. Biased Brownian ratcheting leads to pre-mRNA remodeling and capture prior to first-step splicing. Nature Structural and Molecular Biology. 20, 1450-1457 (2013).
- Gray, R. M., et al. Distinct effects on splicing of two monoclonal antibodies directed against the amino-terminal domain of galectin-3. Archives of Biochemistry and Biophysics. 475, 100-108 (2008).

