Komplementering af splejsning aktivitet af en Galectin-3 - U1 snRNP Kompleks på perler
Summary
I denne artikel beskrives forsøgsprocedurerne for a) udtømning af U1 snRNP fra nukleare ekstrakter med deraf følgende tab af splejsning; og b) rekonstitution af splejsning aktivitet i U1-udtømte ekstrakt af galectin-3 – U1 snRNP partikler bundet til perler kovalent kombineret med anti-galectin-3 antistoffer.
Abstract
Klassiske udtømning-rekonstitution eksperimenter tyder på, at galectin-3 er en nødvendig splejsning faktor i nukleare ekstrakter. Mekanismen for inkorporering af galectin-3 i splejsning vej er behandlet i dette papir. Sedimentering af HeLa celle nukleare ekstrakter på 12%-32% glycerol gradienter giver fraktioner beriget i en endogen ~ 10S partikel, der indeholder galectin-3 og U1 snRNP. Vi beskriver nu en protokol om at nedbryde nukleare ekstrakter af U1 snRNP med samtidig tab af splejsning aktivitet. Splejsning aktivitet i U1-udtømte ekstrakt kan rekonstitueres af galectin-3 – U1 snRNP partikel fanget på agarose perler kovalent kombineret med anti-galectin-3 antistoffer. Resultaterne tyder på, at galectin-3 – U1 snRNP – præ-mRNA ternary kompleks er et funktionelt E-kompleks, der fører til mellemprodukter og produkter af splejsning reaktion, og at galectin-3 kommer ind i splejsning vej gennem sin tilknytning til U1 snRNP. Ordningen med at anvende komplekser affinitet- eller immun-udvalgt på perler til at rekonstruere splejsning aktivitet i ekstrakter udtømt af en bestemt splejsning faktor kan være generelt gældende for andre systemer.
Introduction
Produktion af de fleste eukaryote messenger RNA’er (mRNAs) indebærer fjernelse af introner og ligation af exons i en nuklear proces, der kaldes præ-mRNA splejsning1. To klasser af RNA-proteinkomplekser (RRNPs) leder behandlingen af RNA før budbringeren ind i moden mRNA via splejsede komplekser. En klasse, spirende pre-messenger RNPs, er dannet co-transcriptionally ved binding af heterogene nukleare RNP proteiner og andre RNA-bindende proteiner, herunder nogle medlemmer af SR familien, der giver hnRNP komplekser2. Den anden klasse, uracil-rige små nukleare RNPs (U snRNPs med U1, U2, U4, U5, og U6 snRNAs) er forbundet med U-specifikke og kerneproteiner3,4. U snRNPs interagerer på en ordnet måde med specifikke regioner af præ-messenger RNPs i en dynamisk remodeling vej som introns er fjernet og exons er ligated til at producere modne mRNPs5. Mange yderligere nukleare proteiner deltager i disse behandlingshændelser6.
Galectin-1 (Gal1) og galectin-3 (Gal3) er to proteiner, der er nødvendige faktorer i splejsning vej som vist ved udtømning-rekonstitution undersøgelser7,8. Fjernelse af begge galectiner fra splejsning af kompetente nukleare ekstrakter (NE) afskaffer splejsning og splejsning aktivitet på et tidligt trin. Tilføjelse af enten galectin til en sådan dobbelt udtømt NE genopretter begge aktiviteter. Gal1 og Gal3 er komponenter i aktive splekviomer, som det fremgår af specifik immunprecipitation af præ-mRNA, splejsning mellemprodukter, og modne mRNA ved antiserum specifikke for enten Gal1 eller Gal39. Det er vigtigt, gal3 forbinder med endogene U snRNA indeholdende partikler i NE uden for splejsning vej som det fremgår af nedbør af snRNPs af anti-Gal3 antisera10. Endelig ændrer lyddæmpning af Gal3 i HeLa-celler splejsningsmønstre af mange gener11.
I NE præ-inkuberet til at adskille præformede spleknoomer12, snRNPs findes i flere komplekser sedimentering i glycerol gradienter fra 7S til større end 60S. Selvom glycerolgradientfraktionering er en almindelig teknik til isolering af splejsede komplekser og komponenter (se f.eks. referencer13,14,15), har vi udvidet denne metode ved at karakterisere specifikke fraktioner ved hjælp af antistofimmunpitationer. En snRNP sedimentering ved 10S indeholder kun U1 snRNA sammen med Gal3. Immunprecipitation af 10S-fraktionen med antisera, der er specifik for Gal3 eller U1 snRNP, medudfælder både U1 og Gal3, hvilket indikerer, at nogle af U1 snRNP monopartiklerne er bundet til Gal310. Da U1 snRNP er det første kompleks, der binder sig til præ-mRNP i splejset samling1,5, repræsenterer dette trin et potentielt indgangssted for Gal3 i splejsningsvejen. På dette grundlag viste vi, at 10S Gal3-U1 snRNP monopartikler bundet til anti-Gal3 indeholder perler restaureret splejsning aktivitet til en U1 snRNP udtømt NE, oprettelse af dette kompleks som en mekanisme, hvorved Gal3 er rekrutteret i splejset vej16. Dette står i kontrast til forsøg på at isolere splejsomer på bestemte stadier i splejsning reaktion og katalogisering de tilhørende faktorer17,18. I sådanne undersøgelser fastslås tilstedeværelsen af visse faktorer på et tidspunkt, men ikke den mekanisme, hvormed de blev indlæst.
Vi havde tidligere beskrevet i detaljer forberedelsen af NE, splejsningsubstratet, samlingen af splejsning reaktion blanding, og analysen af produkter i vores dokumentation af galectins rolle i præ-mRNA splejsning19. Vi beskriver nu forsøgsprocedurerne for fraktionering af nukleare ekstrakter for at opnå en brøkdel beriget i Gal3 – U1 snRNP-kompleks og for immunvalg af sidstnævnte kompleks for at rekonstruere splejsningsaktivitet i et U1-udtømt nukleart ekstrakt.
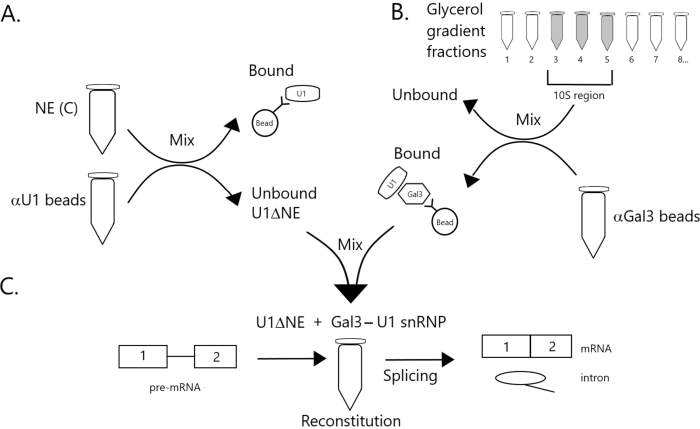
Figur 1: Skemat diagram, der illustrerer komplementering af splejsning i nukleart ekstrakt udtømt af U1 snRNP af et Gal3-U1 snRNP-kompleks på perler. (A) NE i Buffer C (NE(C)) inkuberes med Protein A-Sepharose perler kovalent kombineret med anti-U1 snRNP (αU1 perler). Den ubundne brøk er udtømt af U1 snRNP (U1ΔNE). (B) NE i Buffer D (NE(D)) er fraktioneret over en 12%-32% glycerol gradient ved ultracentrifugering. Fraktioner svarende til 10S-regionen (fraktioner 3-5) kombineres og blandes med perler kovalent kombineret med anti-Gal3 antistoffer (αGal3 perler). Materialet bundet til perlerne indeholder en Gal3-U1 snRNP monopartikler. (C) Gal3-U1 snRNP-komplekset fra del (B) blandes med U1ΔNE fra del (A) i en splejsning ved hjælp af 32P-mærket MINX pre-mRNA-substrat, og mellemprodukter og produkter fra splejsningreaktionen analyseres ved gelelektroforse og autoradiografi. Klik her for at se en større version af dette tal.
Protocol
Representative Results
Discussion
Denne rapport giver de eksperimentelle detaljer, der dokumenterer en Gal3 – U1 snRNP kompleks fanget på anti-Gal3 belagt perler kan binde sig til præ-mRNA substrat og dette ternary kompleks kan genoprette splejsning aktivitet til en U1 snRNP-udtømt NE. Gal3 er et medlem af en familie af proteiner oprindeligt isoleret på grundlag af sin galactose-specifikke kulhydrat-bindende aktivitet23 . Tidlige immunfluorescens- og subcellulære fraktioneringsundersøgelser gav den første antydning af en sa…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Dette arbejde er blevet støttet af National Science Foundation Grant MCB-0092919 og Michigan State University Intramural Research Grant 09-CDFP-2001 (til RJP) og af National Institutes of Health Grant GM-38740 og Michigan AgBioResearch Project MICL02455 (til JLW).
MINX pre-mRNA substrat, der anvendes i splejsning assays var en venlig gave fra Dr. Susan Berget (Baylor College of Medicine, Houston, TX, USA).
Materials
| anti-U1 snRNP | The Binding Site | Hu ENA-RNP #33471 | human autoimmune serum specific for U1 snRNP |
| bottle top vacuum filter | Fisher Scientific | Corning 431153 (0.22 μm; PES 150 ml) | for filtering solutions containing Tris |
| centrifuge | International Equipment Company | IEC Model PR-6 | for pelletting Sepharose beads in immunoprecipitation |
| diethylpyrocarbonate (DEPC) | Sigma-Aldrich | 159220-5G | for treatment of water used in preparation of all solutions |
| dimethylpimelimidate (DMP) | Sigma-Aldrich | 80490-5G | for cross-linking antibody to Sepharose beads |
| electrophoresis cell | BioRad Laboratories, Inc | Mini-Protean II | for SDS-PAGE separation of proteins |
| ethanolamine | Sigma-Aldrich | 411000-100ml | for blocking after the cross-linking reaction |
| gel electrophoresis system | Hoefer, Inc | HSI SE 500 Series | for separating snRNAs by gel electrophoresis |
| gel slab dryer | BioRad | Model 224 | for drying gel slabs for autoradiography |
| Hybond ECL membrane | GE Healthcare | RPN3032D (0.2 μm; 30 cm x 3 m) | for immunoblotting of proteins on membrane |
| microdialyzer (12 x 100 μl sample capacity) | Pierce | Microdialyzer System 100 | for exchanging the buffer of nuclear extract |
| microdialyzer membranes (8K cutoff) | Pierce | 66310 | for exchanging the buffer of nuclear extract |
| non-fat dry milk | Spartan Stores | Spartan Instant Non-fat Dry Milk | |
| Protein A Sepharose CL-4B | Millipore-Sigma | GE 17-0780-01 | for coupling antibody to beads |
| Proteinase K | Millipore-Sigma | P2308-5mg | for stopping the splicing reaction to isolate the RNAs |
| RNasin | Promega | N2111 | for inhibiting ribonuclease activity |
| rocker/rotator | Lab Industries, Inc | Labquake Shaker 400-110 | for mixing protein solutions in coupling reactions and in immunoprecipitation |
| Safety-Solve | Research Products International Corp. | No. 111177 | scintillation counting cocktail for determination of radioactivity in splicing substrate |
| scintillation counter | Beckman Instruments | LS6000SC | scintillation counter for determination of radioactivity |
| speed vaccum concentrator | Savant | SVC 100H | for drying ethanol-precipitated RNA pellets |
| Transphor electrophoresis unit | Hoefer, Inc | Hoefer TE Series Transphor | for protein transfer from SDS-PAGE to blotting membrane |
Riferimenti
- Hoskins, A. A., Moore, M. J. The spliceosome: a flexible, reversible macromolecular machine. Trends In Biochemical Sciences. 37, 179-188 (2012).
- Choi, Y. D., Grabowski, P., Sharp, P. A., Dreyfuss, G. Heterogeneous nuclear ribonucleoproteins: role in RNA splicing. Science. 231, 1534-1539 (1986).
- Lerner, M., Steitz, J. A. Snurps and scyrps. Cell. 25, 298-300 (1981).
- Maniatis, T., Reed, R. The role of small nuclear ribonucleoprotein particles in pre-mRNA splicing. Nature. 325, 673-678 (1987).
- Hoskins, A. A., et al. Ordered and dynamic assembly of single spliceosomes. Science. 331, 1289-1295 (2011).
- Coppin, L., Leclerc, J., Vincent, A., Porchet, N., Pigny, P. Messenger RNA life-cycle in cancer: emerging role of conventional and non-conventional RNA-binding proteins. International Journal of Molecular Sciences. 19, 650-676 (2018).
- Dagher, S. F., Wang, J. L., Patterson, R. J. Identification of galectin-3 as a factor in pre-mRNA splicing. Proceedings of the National Academy of Sciences of the United States of America. 92, 1213-1217 (1995).
- Vyakarnam, A., Dagher, S. F., Wang, J. L., Patterson, R. J. Evidence for a role for galectin-1 in pre-mRNA splicing. Molecular and Cellular Biology. 17, 4730-4737 (1997).
- Wang, W., Park, J. W., Wang, J. L., Patterson, R. J. Immunoprecipitation of spliceosomal RNAs by antisera to galectin-1 and galectin-3. Nucleic Acids Research. 34, 5166-5174 (2006).
- Haudek, K. C., Voss, P. G., Locascio, L. E., Wang, J. L., Patterson, R. J. A mechanism for incorporation of galectin-3 into the spliceosome through its association with U1 snRNP. Biochimica. 48, 7705-7712 (2009).
- Fritsch, K., et al. Galectin-3 interacts with components of the nuclear ribonucleoprotein complex. BMC Cancer. 16, 502-511 (2016).
- Conway, G. C., Krainer, A. R., Spector, D. L., Roberts, R. J. Multiple splicing factors are released from endogenous complexes during in vitro pre-mRNA splicing. Molecular and Cellular Biology. 9, 5273-5280 (1989).
- Dery, K. J., Yean, S. L., Lin, R. J. Assembly and glycerol gradient isolation of yeast spliceosomes containing transcribed or synthetic U6 snRNA. Methods in Molecular Biology. 488, 41-63 (2008).
- Yoshimoto, R., Kataoka, N., Okawa, K., Ohno, M. Isolation and characterization of post-splicing lariat-intron complexes. Nucleic Acids Research. 37, 891-902 (2009).
- Malca, H., Shomron, N., Ast, G. The U1 snRNP base pairs with the 5′ splice site within a penta-snRNP complex. Molecular and Cellular Biology. 23, 3442-3455 (2003).
- Haudek, K. C., Voss, P. G., Wang, J. L., Patterson, R. J. A 10S galectin-3 – snRNP complex assembles into active spliceosomes. Nucleic Acids Research. 44, 6391-6397 (2016).
- Rappsilber, J., Ryder, U., Lamond, A. I., Mann, M. Large-scale proteomic analysis of the human spliceosome. Genome Research. 12, 1231-1245 (2002).
- Jurica, M. S., Moore, M. J. Capturing splicing complexes to study structure and mechanism. Methods. 28, 336-345 (2002).
- Patterson, R. J., Haudek, K. C., Voss, P. G., Wang, J. L. Examination of the role of galectins in pre-mRNA splicing. Methods in Molecular Biology. 1207, 431-449 (2015).
- Dignam, J. D., Lebovitz, R. M., Roeder, R. G. Accurate transcription initiation by RNA polymerase II in a soluble extract from isolated mammalian nuclei. Nucleic Acids Research. 11, 1475-1489 (1983).
- Agarwal, N., Sun, Q., Wang, S. Y., Wang, J. L. Carbohydrate-binding protein 35. I. Properties of the recombinant polypeptide and the individuality of the domains. Journal of Biological Chemistry. 268, 14932 (1993).
- Zillmann, M., Zapp, M. I., Berget, S. M. Gel electrophoretic isolation of splicing complexes containing U1 small nuclear ribonucleoprotein particles. Molecular and Cellular Biology. 8, 814-821 (1988).
- Barondes, S. H., et al. Galectins: a family of animal β-galactoside-binding proteins. Cell. 76, 597-598 (1994).
- Laing, J. G., Wang, J. L. Identification of carbohydrate binding protein 35 in heterogeneous nuclear ribonucleoprotein complex. Biochimica. 27, 5329-5334 (1988).
- Vyakarnam, A., Lenneman, A. J., Lakkides, K. M., Patterson, R. J., Wang, J. L. A comparative nuclear localization study of galectin-1 with other splicing components. Experimental Cell Research. 242, 419-428 (1998).
- Michaud, S., Reed, R. An ATP-independent complex commits pre-mRNA to the mammalian spliceosome assembly pathway. Genes & Development. 5, 2534-2546 (1991).
- Chiu, Y. -. F., et al. Cwc25 is a novel splicing factor required after Prp2 and Yju2 to facilitate the first catalytic reaction. Molecular and Cellular Biology. 29, 5671-5678 (2009).
- Krishnan, R., et al. Biased Brownian ratcheting leads to pre-mRNA remodeling and capture prior to first-step splicing. Nature Structural and Molecular Biology. 20, 1450-1457 (2013).
- Gray, R. M., et al. Distinct effects on splicing of two monoclonal antibodies directed against the amino-terminal domain of galectin-3. Archives of Biochemistry and Biophysics. 475, 100-108 (2008).

