विकास और जीन अभिव्यक्ति में विषमता को मापने के लिए माइक्रोकॉलोनियों की उच्च-थ्रूपुट लाइव इमेजिंग
Summary
खमीर विकास फेनोटाइप को माइक्रोकॉलनी में बढ़ने वाली स्थिर कोशिकाओं के अत्यधिक समानांतर समय-चूक इमेजिंग के माध्यम से ठीक मापा जाता है। इसके साथ ही, तनाव सहिष्णुता, प्रोटीन अभिव्यक्ति, और प्रोटीन स्थानीयकरण की निगरानी की जा सकती है, एकीकृत डेटासेट पैदा करने के लिए कैसे पर्यावरण और आनुवंशिक मतभेदों का अध्ययन करने के लिए, साथ ही जीन अभिव्यक्ति विषमता आइसोजेनिक कोशिकाओं के बीच, विकास मिलाना ।
Abstract
माइक्रोबियल विकास दर में बीच और भीतर तनाव विषमता के सटीक माप तनाव सहिष्णुता, रोगजनकता, और फिटनेस के अन्य प्रमुख घटकों में आनुवंशिक और पर्यावरणीय आदानों को समझने के लिए आवश्यक हैं। यह पांडुलिपि एक माइक्रोस्कोप-आधारित परख का वर्णन करती है जो प्रति प्रयोग लगभग 105 सैकरोमाइसेस सेरेविसिया माइक्रोकॉलनी को ट्रैक करती है। एक मल्टीवेल प्लेट में स्थिर खमीर के स्वचालित समय-चूक इमेजिंग के बाद, माइक्रोकॉलोनी विकास दर आसानी से कस्टम छवि विश्लेषण सॉफ्टवेयर के साथ विश्लेषण कर रहे हैं । प्रत्येक माइक्रोकॉलोनी के लिए, फ्लोरोसेंट प्रोटीन की अभिव्यक्ति और स्थानीयकरण और तीव्र तनाव के अस्तित्व पर भी नजर रखी जा सकती है। यह परख उपभेदों की औसत वृद्धि दर के सटीक अनुमान की अनुमति देता है, साथ ही क्लोनल आबादी के भीतर विकास, जीन अभिव्यक्ति और तनाव सहिष्णुता में विषमता का व्यापक मापन करता है।
Introduction
विकास फेनोटाइप खमीर फिटनेस के लिए गंभीर योगदान करते हैं। प्राकृतिक चयन प्रभावी जनसंख्या आकार के विपरीत से भिन्न विकास दर के साथ वंश के बीच कुशलता से अंतर कर सकता है, जो 108 व्यक्तियों से अधिक हो सकता है1। इसके अलावा, जनसंख्या के भीतर व्यक्तियों के बीच विकास दर की परिवर्तनशीलता एक विकासवादी रूप से प्रासंगिक पैरामीटर है, क्योंकि यह शर्त हेजिंग2,3,4,5,6 जैसी अस्तित्व रणनीतियों के लिए आधार के रूप में काम करसकताहै। इसलिए, परख है कि विकास फेनोटाइप के अत्यधिक सटीक माप के लिए अनुमति देते हैं और उनके वितरण सूक्ष्मजीवों के अध्ययन के लिए निर्णायक हैं। यहां वर्णित माइक्रोकॉलोनी विकास परख प्रति प्रयोग ~ 105 माइक्रोकॉलन के लिए व्यक्तिगत विकास दर माप उत्पन्न कर सकती है। इस परख इसलिए खमीर विकासवादी आनुवंशिकी और जीनोमिक्स का अध्ययन करने के लिए एक शक्तिशाली प्रोटोकॉल प्रदान करता है। यह अपने आप को विशेष रूप से अच्छी तरह से परीक्षण करने के लिए उधार देता है कि आनुवंशिक रूप से समान एकल कोशिकाओं की आबादी के भीतर परिवर्तनशीलता कैसे उत्पन्न होती है, बनाए रखा जाता है, और जनसंख्याफिटनेस7, 8,9,10में योगदान देता है।
यहां वर्णित विधि(चित्रा 1)माइक्रोकॉलन में विकास को ट्रैक करने के लिए तरल मीडिया में 96-या 384-अच्छी तरह से ग्लास-बॉटम प्लेट पर बढ़ने वाली कोशिकाओं की समय-समय पर कैप्चर की गई, कम आवर्धन ब्राइटफील्ड छवियों का उपयोग करती है। कोशिकाएं लेक्टिन कॉनकानवेलियन ए का पालन करती हैं, जो माइक्रोस्कोप प्लेट के नीचे कोट करती हैं, और दो आयामी उपनिवेश बनाती हैं। क्योंकि माइक्रोकॉलोनी मोनोलेयर में बढ़ती है, माइक्रोकॉलोनी क्षेत्र सेल संख्या7के साथ अत्यधिक सहसंबद्ध है। इसलिए, माइक्रोकॉलोनी विकास दर और अंतराल समय के सटीक अनुमान कस्टम छवि-विश्लेषण सॉफ्टवेयर के साथ उत्पन्न किए जा सकते हैं जो प्रत्येक माइक्रोकॉलोनी के क्षेत्र के परिवर्तन की दर को ट्रैक करता है। इसके अलावा, प्रायोगिक सेटअप इन माइक्रोकॉलनियों में व्यक्त फ्लोरोसेंटली लेबल प्रोटीन के बहुतायत और यहां तक कि उपकोशिकीय स्थानीयकरण की निगरानी कर सकता है। इस माइक्रोकॉलोनी विकास परख से डेटा के डाउनस्ट्रीम प्रसंस्करण कस्टम विश्लेषण द्वारा या मौजूदा छवि विश्लेषण सॉफ्टवेयर द्वारा प्राप्त किया जा सकता है, जैसे कि प्रोसेसिंग इमेजेज आसानी से (पाई)11,मजबूत कॉलोनी क्षेत्र मान्यता के लिए एक एल्गोरिदम और कम आवर्धन, ब्राइटफील्ड छवियों से उच्च-थ्रूपुट विकास विश्लेषण, जो गिटहब12के माध्यम से उपलब्ध है।
क्योंकि माइक्रोकॉलोनी-विकास परख से प्राप्त विकास दर अनुमान बड़ी संख्या में एकल-कॉलोनी मापों से उत्पन्न होते हैं, वे बेहद सटीक होते हैं, मानक त्रुटियों के साथ एक यथोचित आकार के प्रयोग के लिए अनुमानों की तुलना में परिमाण के कई आदेश छोटे होते हैं। इसलिए, विभिन्न जीनोटाइप, उपचार या पर्यावरणीय स्थितियों के बीच विकास दर के अंतर का पता लगाने के लिए परख की शक्ति अधिक है। मल्टीवेल-प्लेट प्रारूप कई अलग-अलग वातावरण और जीनोटाइप संयोजनों को एक ही प्रयोग में तुलना करने की अनुमति देता है। यदि उपभेदों का गठन विभिन्न फ्लोरोसेंट मार्कर व्यक्त करते हैं, तो उन्हें एक ही कुएं में मिलाया जा सकता है और बाद में छवि विश्लेषण द्वारा प्रतिष्ठित किया जा सकता है, जो अच्छी तरह से डेटा सामान्यीकरण की अनुमति देकर बिजली को और बढ़ा सकता है।
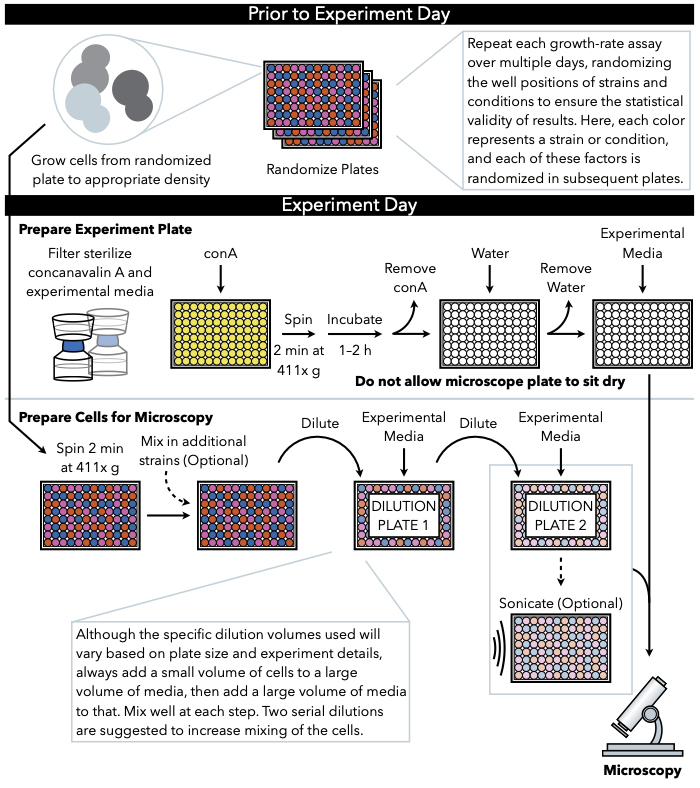
चित्रा 1:प्रोटोकॉल का योजनाबद्ध प्रतिनिधित्व। यह प्रोटोकॉल दो मुख्य चरणों का पालन करता है, जो प्रयोगात्मक प्लेट की तैयारी और कोशिकाओं को छवि के लिए तैयार कर रहे हैं। प्लेटों का यादृच्छिकीकरण और कोशिकाओं का विकास प्रयोग दिवस तक पहले और अग्रणी होना चाहिए। कमजोर पड़ने के दौरान प्रत्येक चरण में कोशिकाओं का बार-बार मिश्रण चढ़ाना तक चरणों में जरूरी है, और इसलिए प्रयोगात्मक प्लेट को पहले तैयार करने की सिफारिश की जाती है ताकि यह कोशिका कमजोर पड़ने के पूरा होने पर तुरंत चढ़ाना के लिए तैयार हो। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।
Protocol
Representative Results
Discussion
यहां वर्णित प्रोटोकॉल एक बहुमुखी परख है जो सेल विकास और जीन अभिव्यक्ति को व्यक्तिगत माइक्रोकॉलनी के स्तर पर एक साथ निगरानी करने की अनुमति देता है। इन दोनों तौर-तरीकों के संयोजन से अद्वितीय जैविक अंतर?…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
हम नाओमी जिव, साशा लेवी और शुआंग ली को इस प्रोटोकॉल को विकसित करने में उनके योगदान के लिए धन्यवाद देते हैं, साझा उपकरणों के लिए डेविड ग्रेशम, और वीडियो उत्पादन में मदद के लिए मारिसा टीला। इस काम को राष्ट्रीय स्वास्थ्य अनुदान संस्थानों R35GM118170 द्वारा समर्थित किया गया था।
Materials
| General Materials | |||
| 500 mL Bottletop Filter .22 µm PES Sterilizing, Low Protein Binding, w/45mm Neck | Fisher | CLS431154 | used to filter the media |
| BD Falcon*Tissue Culture Plates, microtest u-bottom | Fisher | 08-772-54 | 96-well culture tubes used to freeze cells, pre-grow cells, and dilutions |
| BD Syringes without Needle, 50 mL | Fisher | 13-689-8 | Used to filter the Concanavalin A |
| Costar Sterile Disposable Reagent Reservoirs | Fisher | 07-200-127 | reagent reservoirs used to pipette solutions with multichannel pipette |
| Costar Thermowell Aluminum Sealing Tape | Fisher | 07-200-684 | 96-well plate seal for pre-growth and freezing |
| lint and static free Kimwipes | Fisher | 06-666A | lint and static free wipes to keep microscope plate bottom free of debris and scratches |
| Nalgene Syringe Filters | ThermoFisher Scientific | 199-2020 | 0.2 μm pore size, 25 mm diameter; used to filter concanavalin A solution |
| Media Components | |||
| Minimal chemically defined media (MD; 2% glucose) | alternative microscopy media used for yeast pre-growth and growth during microscopy | ||
| Synthetic Complete Media (SC; 2% glucose) | microscopy media used for yeast pre-growth and growth during microscopy | ||
| Yeast extract-peptone-dextrose (YEPD; 2% glucose) medium | cell growth prior to freezing down randomized plates | ||
| Microscopy Materials | |||
| Breathe-Easy sealing membrane | Millipore Sigma | Z380059-1PAK | breathable membranes used to seal plate during microscopy experiment. At this stage breathable membranes are reccomended because they prevent condensation in the wells and allow for better microscopy images |
| Brooks 96-well flat clear glass bottom microscope plate | Dot Scientific | MGB096-1-2-LG-L | microscope plate |
| Concanavalin A from canavalia ensiformis (Jack Bean), lyophilized powder | Millipore Sigma | 45-C2010-1G | Make 5x concanavalin A solution and freeze 5ml of 5x concanavalin A in 50 mL conical tubes at -80 °C |
| Strains Used | |||
| MAH.5, MAH.96, MAH.52, MAH.66, MAH.11, MAH.58, MAH.135, MAH.15, MAH.44, MAH.132 | Haploid mutation accumulation strains in a laboratory background, described in Hall and Joseph 2010 | ||
| EP026.2A-2C | Progeny of the ancestral Hall and Joseph 2010 mutation accumulation strain, transformed with YFR054cΔ::Scw11P::GFP | ||
| Equipment | |||
| Misonix Sonicator S-4000 with 96-pin attachment | Sonicator https://www.labx.com/item/misonix-inc-s-4000-sonicator/4771281 | ||
| Nikon Eclipse Ti-E with Perfect Focus System | Inverted microscope with automated stage and autofocus system |
Riferimenti
- Geiler-Samerotte, K. A., Hashimoto, T., Dion, M. F., Budnik, B. A., Airoldi, E. M., Drummond, D. A. Quantifying condition-dependent intracellular protein levels enables high-precision fitness estimates. PloS one. 8 (9), 75320 (2013).
- Kussell, E., Leibler, S. Phenotypic diversity, population growth, and information in fluctuating environments. Science. 309 (5743), 2075-2078 (2005).
- Thattai, M., van Oudenaarden, A. Stochastic gene expression in fluctuating environments. Genetica. 167 (1), 523-530 (2004).
- King, O. D., Masel, J. The evolution of bet-hedging adaptations to rare scenarios. Theoretical population biology. 72 (4), 560-575 (2007).
- Acar, M., Mettetal, J. T., van Oudenaarden, A. Stochastic switching as a survival strategy in fluctuating environments. Nature genetics. 40 (4), 471-475 (2008).
- Avery, S. V. Microbial cell individuality and the underlying sources of heterogeneity. Nature reviews. Microbiology. 4 (8), 577-587 (2006).
- Levy, S. F., Ziv, N., Siegal, M. L. Bet hedging in yeast by heterogeneous, age-correlated expression of a stress protectant. PLoS biology. 10 (5), 1001325 (2012).
- van Dijk, D., et al. Slow-growing cells within isogenic populations have increased RNA polymerase error rates and DNA damage. Nature communications. 6, 7972 (2015).
- Ziv, N., Shuster, B. M., Siegal, M. L., Gresham, D. Resolving the Complex Genetic Basis of Phenotypic Variation and Variability of Cellular Growth. Genetica. 206 (3), 1645-1657 (2017).
- Li, S., Giardina, D. M., Siegal, M. L. Control of nongenetic heterogeneity in growth rate and stress tolerance of Saccharomyces cerevisiae by cyclic AMP-regulated transcription factors. PLoS genetics. 14 (11), 1007744 (2018).
- Plavskin, Y., Li, S., Ziv, N., Levy, S. F., Siegal, M. L. Robust colony recognition for high-throughput growth analysis from suboptimal low-magnification brightfield micrographs. bioRxiv. , (2018).
- Ziv, N., Siegal, M. L., Gresham, D. Genetic and nongenetic determinants of cell growth variation assessed by high-throughput microscopy. Molecular biology and evolution. 30 (12), 2568-2578 (2013).
- Hall, D. W., Joseph, S. B. A high frequency of beneficial mutations across multiple fitness components in Saccharomyces cerevisiae. Genetica. 185 (4), 1397-1409 (2010).
- Saleemuddin, M., Husain, Q. Concanavalin A: a useful ligand for glycoenzyme immobilization–a review. Enzyme and microbial technology. 13 (4), 290-295 (1991).
- Geiler-Samerotte, K. A., Bauer, C. R., Li, S., Ziv, N., Gresham, D., Siegal, M. L. The details in the distributions: why and how to study phenotypic variability. Current opinion in biotechnology. 24 (4), 752-759 (2013).
- Nakagawa, S., Schielzeth, H. Repeatability for Gaussian and non-Gaussian data: a practical guide for biologists. Biological reviews of the Cambridge Philosophical Society. 85 (4), 935-956 (2010).
- Bolker, J. A. Exemplary and surrogate models: two modes of representation in biology. Perspectives in biology and medicine. 52 (4), 485-499 (2009).

