Ligand Tahlilinin Diferansiyel Radyal Kılcal Etkisi ile Küçük Ligandların Bağlayıcı Proteinlerinin Tanımlanması (DRaCALA)
Summary
Ligand Assay’ın Diferansiyel Radyal Kılcal Etkisi (DRaCALA), orfeome kütüphanesi kullanarak bir organizmanın küçük ligand bağlayıcı proteinlerini tanımlamak için kullanılabilir.
Abstract
Son on yılda bakteriyel fizyolojide küçük sinyal moleküllerinin anlaşılmasında muazzam bir ilerleme kaydedildi. Özellikle, nükleotid türevi ikincil habercilerin (NSM) hedef proteinleri sistematik olarak tanımlanmış ve model organizmalarda incelenmiştir. Bu başarılar esas olarak, bu küçük moleküllerin hedef proteinlerini sistematik olarak tanımlamak için kullanılan ligand tahlilinin (DRaCALA) yakalama bileşik tekniği ve diferansiyel radyal kılcal etkisi de dahil olmak üzere birkaç yeni tekniğin geliştirilmesinden kaynaklanmaktadır. Bu makalede, DRaCALA tekniğinin bir örneği ve video gösterimi olarak NSM’lerin, guanosine penta ve tetrafosfatların (p)ppGpp kullanımı açıklanmaktadır. DRaCALA kullanılarak, bu tahlilin gücünü gösteren Escherichia coli K-12 model organizmasında bilinen 20’den 9’u ve (p)ppGpp’nin 12 yeni hedef proteini tanımlanmıştır. Prensip olarak, DRaCALA radyoaktif izotoplar veya floresan boyalar ile etiketlenebilen küçük ligandların incelenmesi için kullanılabilir. Bu tekniğin daha fazla uygulanması için DRaCALA’nın kritik adımları, artıları ve eksileri burada tartışılmaktadır.
Introduction
Bakteriler sürekli değişen ortamlara uyum sağlamak için birkaç küçük sinyal molekülü kullanır1,2. Örneğin, otoindüserler, N-acylhomoserine lactones ve modifiye oligopeptidleri, popülasyon davranışını koordine etmek için bakteriler arasındaki hücreler arası iletişime aracılık eder, çekirdek algılama olarak bilinen bir fenomen2. Bir diğer küçük sinyal molekülü grubu, yaygın olarak çalışılan döngüsel adenozin monofosfat (cAMP), döngüsel di-AMP, döngüsel di-guanosine monofosfat (döngüsel di-GMP) ve guanosine penta ve tetra fosfatları (p)ppGpp1dahil olmak üzere NSM’lerdir. Bakteriler bu NSM’leri çeşitli stres koşullarına yanıt olarak üretirler. Üretildikten sonra, bu moleküller hedef proteinlerine bağlanır ve karşılaşılan streslerle başa çıkmak ve bakteriyel sağkalımını artırmak için birkaç farklı fizyolojik ve metabolik yolu düzenler. Bu nedenle, hedef proteinlerin tanımlanması, bu küçük moleküllerin moleküler fonksiyonlarını çözmek için kaçınılmaz bir ön koşuldur.
Son on yıl, esas olarak bu küçük moleküllerin hedef proteinlerini ortaya çıkaran çeşitli teknik yenilikler nedeniyle, bu küçük sinyal molekülleri hakkında bir bilgi patlamasına tanık oldu. Bunlar arasında yakalama bileşik tekniği3,4,5ve ligand tahlil (DRaCALA)6’nın diferansiyel radyal kılcal etkisi bu makalede tartışılacaktır.
Vincent Lee ve iş arkadaşları tarafından 20116’daicat edilen DRaCALA, nitroselüloz membran yeteneğini farklı olarak sequester serbest ve proteine bağlı ligandlara dağıtır. Proteinler gibi moleküller nitroselüloz bir zar üzerinde yayılamazken, NSM’ler gibi küçük ligandlar yayılabilir. NSM’yi(örneğin,ppGpp) test edilecek proteinle karıştırarak ve membran üzerinde tespit ederek, iki senaryo beklenebilir (Şekil 1): Eğer (p)ppGpp proteine bağlanırsa, radyolabeled (p)ppGpp protein tarafından noktanın merkezinde tutulur ve dışarı doğru dağılmasın, yoğun bir küçük nokta (yani. güçlü radyoaktif sinyal) bir fosforimager altında. Bununla birlikte, (p)ppGpp proteine bağlanmazsa, tekdüze arka plan radyoaktif sinyali olan büyük bir nokta üretmek için serbestçe dışa doğru dağılacaktır.
Ayrıca, DRaCALA, protein yeterli miktarda mevcutsa, tüm hücre lisatında küçük bir molekül ile amaçlanmamış bir protein arasındaki etkileşimi tespit edebilir. Bu basitlik, bir ORFeome ifade kitaplığı kullanarak protein hedeflerini hızla tanımlamada DRaCALA’nın kullanılmasına izin verir. Nitekim, cAMP7, siklik di-AMP8, döngüsel di-GMP9,10ve (p)ppGpp 11,12,13hedef proteinleri DRaCALA kullanılarak sistematik olarak tanımlanmıştır. Bu video makalesi, başarılı bir DRaCALA taraması gerçekleştirmedeki kritik adımları ve dikkat edilmesi gerekenleri göstermek ve açıklamak için örnek olarak (p)ppGpp kullanır. Not olarak, DRaCALA14’ün daha kapsamlı bir açıklamasının, DRaCALA’yı gerçekleştirmeden önce bu makaleyle birlikte okunması şiddetle tavsiye edilir.
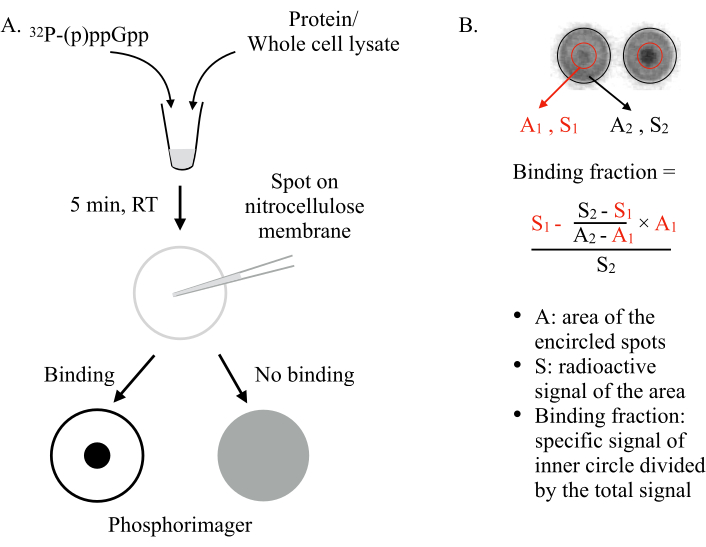
Şekil 1: DRaCALA prensibi. (A) DRaCALA tahlilinin şeması. Ayrıntılar için metne bakın. (B) Bağlama fraksiyonunun nicelleştirilmesi ve hesaplanması. Ayrıntılar için metne bakın. Kısaca, DRaCALA lekeleri, tüm noktayı ve iç koyu noktayı(yani,test edilen proteinin bağlanması nedeniyle tutulan (p)ppGpp) çevreleyen iki daire çizilerek analiz edilecektir. Spesifik bağlama sinyali, spesifik olmayan arka plan sinyalini çıkardıktan sonra iç dairenin (S1) radyoaktif sinyalidir (A1 × ((S2-S1)/(A 2 -A1))) tarafından hesaplanır). Bağlama fraksiyonu, toplam radyoaktif sinyale (S2) bölünen belirli bağlama sinyalidir. Kısaltmalar: DRaCALA = Ligand Tahlilinin Diferansiyel Radyal Kılcal Hareketi; (p)ppGpp = guanosine penta ve tetrafosfatlar; RT = oda sıcaklığı. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Protocol
Representative Results
Discussion
DRaCALA taramasının yapılmasında kritik adımlardan biri iyi tüm hücre lysates elde etmektir. İlk olarak, test edilen proteinler büyük miktarlarda ve çözünür formlarda üretilmelidir. İkincisi, hücrelerin lizisi eksiksiz olmalı ve lizat viskozitesi minimum olmalıdır. Lysozyme’ın dahil edilmesi ve üç donma-çözülme döngüsünün kullanılması genellikle hücreleri tamamen iyse etmek için yeterlidir. Bununla birlikte, serbest bırakılan kromozomal DNA lizozu viskoz yapar ve yüksek arka plan bağ…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Çalışma, YEZ’e bir NNF Proje Hibesi (NNF19OC0058331) ve MlS’e Marie Skłodowska-Curie hibe anlaşması (Nº 801199) kapsamında Avrupa Birliği’nin Horizon 2020 araştırma ve yenilik programı tarafından desteklenmektedir.

Materials
| 32P-α-GTP | Perkinelmer | BLU006X250UC | |
| 96 x pin tool | V&P Scientific | VP 404 | 96 Bolt Replicator, on 9 mm centers, 4.2 mm Bolt Diameter, 24 mm long |
| 96-well V-bottom microtiter plate | Sterilin | MIC9004 | Sterilin Microplate V Well 611V96 |
| Agar | OXOID – Thermo Fisher | LP0011 | Agar no. 1 |
| ASKA collection strain | NBRP, SHIGEN, JAPAN | Ref: DNA Research, Volume 12, Issue 5, 2005, Pages 291–299. https://doi.org/10.1093/dnares/dsi012 | |
| Benzonase | SIGMA | E1014-25KU | genetically engineered endonuclease from Serratia marcescens |
| Bradford Protein Assay Dye | Bio-Rad | 5000006 | Reagent Concentrate |
| DMSO | SIGMA | D8418 | ≥99.9% |
| DNase 1 | SIGMA | DN25-1G | |
| gel filtration10x300 column | GE Healthcare | 28990944 | contains 20% ethanol as preservative |
| Glycerol | PanReac AppliChem | 122329.1214 | Glycerol 87% for analysis |
| Hypercassette | Amersham | RPN 11647 | 20 x 40 cm |
| Imidazole | SIGMA | 56750 | puriss. p.a., ≥ 99.5% (GC) |
| IP Storage Phosphor Screen | FUJIFILM | 28956474 | BAS-MS 2040 20x 40 cm |
| Isopropyl β-d-1-thiogalactopyranoside (IPTG) | SIGMA | I6758 | Isopropyl β-D-thiogalactoside |
| Lysogeny Broth (LB) | Invitrogen – Thermo Fisher | 12795027 | Miller's LB Broth Base |
| Lysozyme | SIGMA | L4949 | from chicken egg white; BioUltra, lyophilized powder, ≥98% |
| MgCl2 (Magnesium chloride) | SIGMA | 208337 | |
| MilliQ water | ultrapure water | ||
| multichannel pipette | Thermo Scientific | 4661110 | F1 – Clip Tip; 1-10 ul, 8 x channels |
| NaCl | VWR Chemicals | 27810 | AnalaR NORMAPUR, ACS, Reag. Ph. Eur. |
| Ni-NTA Agarose | Qiagen | 30230 | |
| Nitrocellulose Blotting Membrane | Amersham Protran | 10600003 | Premium 0.45 um 300 mm x 4 m |
| PBS | OXOID – Thermo Fisher | BR0014G | Phosphate buffered saline (Dulbecco A), Tablets |
| PEG3350 (Polyethylene glycol 3350) | SIGMA | 202444 | |
| phenylmethylsulfonyl fluoride (PMSF) | SIGMA | 93482 | Phenylmethanesulfonyl fluoride solution – 0.1 M in ethanol (T) |
| Phosphor-imager | GE Healthcare | 28955809 | Typhoon FLA-7000 Phosphor-imager |
| Pipette Tips, filtered | Thermo Scientific | 94410040 | ClipTip 12.5 μl nonsterile |
| Poly-Prep Chromatography column | Bio-Rad | 7311550 | polypropylene chromatography column |
| Protease inhibitor Mini | Pierce | A32955 | Tablets, EDTA-free |
| screw cap tube | Thermo Scientific | 3488 | Microcentifuge Tubes, 2.0 ml with screw cap, nonsterile |
| SLS 96-deep Well plates | Greiner | 780285 | MASTERBLOCK, 2 ML, PP, V-Bottom, Natural |
| spin column | Millipore | UFC500396 | Amicon Ultra -0.5 ml Centrifugal Filters |
| Thermomixer | Eppendorf | 5382000015 | Thermomixer C |
| TLC plate (PEI-cellulose F TLC plates) | Merck Millipore | 105579 | DC PEI-cellulose F (20 x 20 cm) |
| Tris | SIGMA | BP152 | Tris Base for Molecular Biology |
| Tween 20 | SIGMA | P1379 | viscous non-ionic detergent |
| β-mercaptoethanol | SIGMA | M3148 | 99% (GC/titration) |
Riferimenti
- Kalia, D., et al. Nucleotide, c-di-GMP, c-di-AMP, cGMP, cAMP, (p)ppGpp signaling in bacteria and implications in pathogenesis. Chemical Society Reviews. 42 (1), 305-341 (2013).
- Camilli, A., Bassler, B. L. Bacterial small-molecule signaling pathways. Science. 311 (5764), 1113-1116 (2006).
- Luo, Y., et al. The cAMP capture compound mass spectrometry as a novel tool for targeting cAMP-binding proteins: from protein kinase A to potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channels. Molecular & Cellular Proteomics. 8 (12), 2843-2856 (2009).
- Nesper, J., Reinders, A., Glatter, T., Schmidt, A., Jenal, U. A novel capture compound for the identification and analysis of cyclic di-GMP binding proteins. Journal of Proteomics. 75 (15), 4874-4878 (2012).
- Laventie, B. J., et al. Capture compound mass spectrometry–a powerful tool to identify novel c-di-GMP effector proteins. Journal of Visual Experiments. (97), e51404 (2015).
- Roelofs, K. G., Wang, J., Sintim, H. O., Lee, V. T. Differential radial capillary action of ligand assay for high-throughput detection of protein-metabolite interactions. Proceedings of the National Academy of Sciences of the United States of America. 108 (37), 15528-15533 (2011).
- Zhang, Y., et al. Evolutionary adaptation of the essential tRNA methyltransferase TrmD to the signaling molecule 3 ‘,5 ‘-cAMP in bacteria. Journal of Biological Chemistry. 292 (1), 313-327 (2017).
- Corrigan, R. M., et al. Systematic identification of conserved bacterial c-di-AMP receptor proteins. Proceedings of the National Academy of Sciences of the United States of America. 110 (22), 9084-9089 (2013).
- Roelofs, K. G., et al. Systematic identification of cyclic-di-GMP binding proteins in Vibrio cholerae reveals a novel class of cyclic-di-GMP-binding ATPases associated with type II secretion systems. PLoS Pathogen. 11 (10), 1005232 (2015).
- Fang, X., et al. GIL, a new c-di-GMP-binding protein domain involved in regulation of cellulose synthesis in enterobacteria. Molecular Microbiology. 93 (3), 439-452 (2014).
- Corrigan, R. M., Bellows, L. E., Wood, A., Grundling, A. ppGpp negatively impacts ribosome assembly affecting growth and antimicrobial tolerance in Gram-positive bacteria. Proceedings of the National Academy of Sciences of the United States of America. 113 (12), 1710-1719 (2016).
- Zhang, Y., Zbornikova, E., Rejman, D., Gerdes, K. Novel (p)ppGpp binding and metabolizing proteins of Escherichia coli. Mbio. 9 (2), 02188 (2018).
- Yang, J., et al. The nucleotide pGpp acts as a third alarmone in Bacillus, with functions distinct from those of (p) ppGpp. Nature Communications. 11 (1), 5388 (2020).
- Orr, M. W., Lee, V. T. Differential radial capillary action of ligand assay (DRaCALA) for high-throughput detection of protein-metabolite interactions in bacteria. Methods in Molecular Biology. 1535, 25-41 (2017).
- Kitagawa, M., et al. Complete set of ORF clones of Escherichia coli ASKA library (A complete Set of E. coli K-12 ORF archive): Unique resources for biological research. DNA Research. 12 (5), 291-299 (2005).
- Hochstadt-Ozer, J., Cashel, M. The regulation of purine utilization in bacteria. V. Inhibition of purine phosphoribosyltransferase activities and purine uptake in isolated membrane vesicles by guanosine tetraphosphate. Journal of Biological Chemistry. 247 (21), 7067-7072 (1972).
- Zhang, Y. E., et al. p)ppGpp regulates a bacterial nucleosidase by an allosteric two-domain switch. Molecular Cell. 74 (6), 1239-1249 (2019).
- Wang, B., et al. Affinity-based capture and identification of protein effectors of the growth regulator ppGpp. Nature Chemical Biology. 15 (2), 141-150 (2019).

