생체멤브레인 포스 프로브 분광법에 의한 분자 스프링 상수 분석
Summary
생체멤브레인 힘 프로브(BFP)는 시투 동적 힘 분광법(DFS) 기술이다. BFP는 살아있는 세포에 분자 상호 작용의 봄 상수를 측정하기 위하여 이용될 수 있습니다. 이 프로토콜은 BFP에 의해 검출된 분자 결합에 대한 스프링 상수 분석을 제시합니다.
Abstract
생체멤브레인 힘 프로브(BFP)는 최근 단일 분자 결합 운동학을 측정하고, 리간드 수용체 상호 작용의 기계적 특성을 평가하고, 단백질 동적 형성 변화를 시각화하고, 보다 흥미롭게도 해명 수용체 매개 세포 메카노센싱 메커니즘을 측정할 수 있는 토종 세포 표면 또는 시투 동적 힘 분광법(DFS) 나노툴로 부상했습니다. 최근에는 BFP가 분자 결합의 봄 상수를 측정하는 데 사용되었습니다. 이 프로토콜은 분자 스프링 상수 DFS 분석을 수행하기 위한 단계별 절차를 설명합니다. 특히 두 개의 BFP 작동 모드, 즉 비드 셀 및 비드 비드 모드에 대해 설명합니다. 이 프로토콜은 DFS 원시 데이터로부터 분자 결합 및 세포의 스프링 상수를 도출하는 데 중점을 둡니다.
Introduction
살아있는 세포 DFS 기술로, BFP는 인간 적혈구 (RBC)를 엔지니어; 그림 1) 0.1-3 pN/nm1,2,3의호환 스프링 상수 범위의 초감도 및 튜닝 가능한 힘 트랜스듀서로. 리간드 수용체 상호 작용을 조사하기 위해 BFP는 DFS 측정을 ~1 pN(10-12 N), ~3nm(10-9m), ~0.5ms(10-3s)로, 유효및 측두해상도 4,5에서가능하게 한다. 실험 구성은 프로브와 대상이라는 두 개의 반대 마이크로파이프로 구성됩니다. 프로브 마이크로피펫은 RBC를 흡인하고 비드는 비오틴 스트렙타비딘 상호 작용을 통해 정점에 붙어 있습니다. 비드는 관심의 리간드로 코팅된다(그림 1A). 대상 마이크로피펫은 비드셀(도1B)과비드비드(도1C)모드에 대응하는 관심 수용체를 베어링하는 셀 또는 비드(dad)를흡인한다.
BFP 시공, 조립 및 DFS 실험 프로토콜은 이전에1,6을상세히 기술하였다. 간단히, BFP 터치 주기는 5 단계로 구성되어 있습니다 : 접근, 임프, 접촉, 철회 및 해리(그림 1D). 수평 RBC 정점 위치는 ΔxRBC로표시됩니다. 처음에, 미스트레스(제로포스) RBC 변형 ΔxRBC는 0(표1)이다. 대상은 프로브 비드(그림 1D)에서충돌하고 철회하는 압조 번역기에 의해 구동됩니다. RBC 프로브는 먼저 음수 RBC 변형 ΔxRBC < 0으로 대상에 의해 압축된다. 본드 이벤트에서, 철회 단계는 압축에서 인장 상으로 전환되며, 양성 RBC 변형 ΔxRBC > 0(도2C 및 D). Hooke의 법칙에 따르면, BFP 베어링 힘은 F = kRBC × ΔxRBC로측정할 수있으며, 여기서 kRBC(표 1)는BFP의 RBC 스프링 상수이다. 본드 파열 및 원터치 사이클이 완료되면 프로브 비드는 ΔxRBC = 0(도1D)으로제로 포스 위치로 돌아갑니다.
kRBC를결정하기 위해, 우리는 RBC와 프로브 비드(도1A)사이의프로브 마이크로피펫 내부 오리피스(Rp),RBC(R0)및 원형 접촉영역(Rc)의반을 측정하고 기록한다. 그런 다음 kRBC는 에반의 모델(Eq. 1)7,8에 따라 BP(도S1A)8,9를작동시키는 가상 계측기(VI)역할을 하는 LabVIEW 프로그램을 이용하여 계산된다.
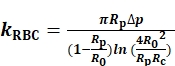 (Eq. 1)
(Eq. 1)
BFP가 확립되고 DFS 원시 데이터가 얻어지므로, 이에 따라 리간드 수용체 쌍 또는 세포의 스프링 상수를 분석하는 방법을 제시한다. DFS 원시 데이터는 글리코처단백질 Thy-1 및 K562 세포 베어링 인테그린 α5 β1(thy-1-α5β1)의상호작용에 관한 것입니다. 도 3A 및 3B)10 및 피브리노겐 및 비드 코팅 인테그린 αIIbβ3 (FGN-αIIbβ3; 도 3C) 11,12는 각각 비드 셀 및 비드-비드 분석 모드를 시연하는 데 사용되어 왔다.
BFP 실험 준비
BFP 실험 준비 및 계측에 대한 자세한 내용은 이전에 게시된 프로토콜3을참조하십시오. 간단히 말해서, 인간 RBC는 탄소/중탄산염 완충제에서 비오틴-PEG3500-NHS를 사용하여 바이오티니징되었습니다. 관심있는 단백질은 인산염 완충제에서 MAL-PEG3500-NHS를 사용하여 보로실리케이트 유리 구슬에 공동으로 결합되었습니다. 생체작 RBC에 부착하기 위해, 프로브 비드는 또한 MAL-SA를 사용하여 스트렙타비딘(SA)으로 코팅된다. 재료 및 표 2의 테이블을 참조하십시오.
BFP(도1, 왼쪽)를조립하기 위해, ‘도우미’라고 불리는 세 번째 마이크로피펫은 프로브 비드를 전달하고 RBC의 정점1,3에접착제를 붙이는 데 사용됩니다. SA 코팅 프로브 비드와 바이오티니저드 RBC 사이의 공유 상호 작용은 리간드 수용체 의 결합보다 훨씬 강하다. 따라서, 해리시아트 단계는 RBC로부터 프로브 비드의 분리보다는 리간드 수용체 결합 파열로 해석될 수 있다.
Protocol
Representative Results
Discussion
요약하자면, 우리는 DFS 원시 데이터를 전처리하고 BFP 비드 비드 및 비드 셀 분석 모드에서 분자 스프링 상수를 도출하기 위한 상세한 데이터 분석 프로토콜을 제공했습니다. 분자 및 세포 스프링 상수를 결정하는 데 필요한 생체 역학 모델 및 방정식이 제시됩니다. 상이한 통합이 연구되고 있기는 하지만, 비드-비드 모드와 비드셀 모드에 의해 측정된 k몰은 상당한 범위 차이를 가지…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
도움이 되는 토론을 위한 기요메 트로아데크, 하드웨어 컨설팅을 위한 지하오 왕, 시드니 제조 허브, 그레그 수안닝, 사이먼 링거에게 실험실 스타트업을 지원해 주셔서 감사합니다. 이 작품은 호주 연구 위원회 디스커버리 프로젝트 (DP2000101970 – L.A.J.), NSW 심장 혈관 용량 구축 프로그램 (초기 중간 경력 연구원 그랜트 – L.A.J.), 시드니 연구 가속기 상 (SOAR – L.A.J.), 라마시오에 의해 지원되었다 티 재단 건강 투자 보조금 (2020HIG76 – L.A.J.), 국립 보건 및 의학 연구 위원회 아이디어 보조금 (APP2003904 – L.A.J.), 엔지니어링 시작 기금 및 주요 장비 계획의 시드니 학부 (L.A.J.). 라이닝 아놀드 주 호주 연구 위원회 DECRA 동료입니다 (DE190100609).
Materials
| 3-Mercaptopropyltrimethoxysilane (MPTMS) | Uct, Specialties, llc | 4420-74-0 | Glass bead functionalization |
| Anhy. Sodium Phosphate Dibasic (Na2HPO4) | Sigma-Aldrich | S7907 | Phosphate buffer preparation |
| BFP data acquisition VI | LabVIEW | BFP control and parameter setting | |
| BFP data analysis VI | LabVIEW | BFP raw data analysis | |
| Biotin-PEG3500-NHS | JenKem | A5026-1 | RBC biotinylation |
| Borosilicate Glass beads | Distrilab Particle Technology, Netherlands | 9002 | Glass bead functionalization |
| Bovine serum albumin | Sigma-Aldrich | A0336 | Ligand functionalization |
| Camera VI | LabVIEW | BFP monitoring | |
| D-glucose | Sigma-Aldrich | G7021 | Tyrode’s buffer preparation |
| Hepes | Sigma-Aldrich | H3375 | Tyrode’s buffer preparation |
| MAL-PEG3500-NHS | JenKem | A5002-1 | Glass bead functionalization |
| Potassium Chloride (KCl) | Sigma-Aldrich | P9541 | Tyrode’s buffer preparation |
| Sodium Bicarbonate (NaHCO3) | Sigma-Aldrich | S5761 | Carbonate/bicarbonate buffer preparation; Tyrode’s buffer preparation |
| Sodium Carbonate (Na2CO3) | Sigma-Aldrich | S2127 | Carbonate/bicarbonate buffer preparation |
| Sodium Chloride (NaCl) | Sigma-Aldrich | S7653 | Tyrode’s buffer preparation |
| Sodium Phosphate Monobasic Monohydrate (NaH2PO4•H2O) | Sigma-Aldrich | S9638 | Phosphate buffer preparation |
| Streptavidin-Maleimide | Sigma-Aldrich | S9415 | Glass bead functionalization |
Riferimenti
- Chen, Y., et al. Fluorescence Biomembrane Force Probe: Concurrent Quantitation of Receptor-ligand Kinetics and Binding-induced Intracellular Signaling on a Single Cell. The Journal of Visualized Experiments. (102), e52975 (2015).
- Su, Q. P., Ju, L. A. Biophysical nanotools for single-molecule dynamics. Biophysics Reviews. 10 (5), 1349-1357 (2018).
- Ju, L. Dynamic Force Spectroscopy Analysis on the Redox States of Protein Disulphide Bonds. Methods in Molecular Biology. 1967, 115-131 (2019).
- An, C., et al. Ultra-stable Biomembrane Force Probe for Accurately Determining Slow Dissociation Kinetics of PD-1 Blockade Antibodies on Single Living Cells. Nano Letters. 20 (7), 5133-5140 (2020).
- Chen, Y., Ju, L., Rushdi, M., Ge, C., Zhu, C. Receptor-mediated cell mechanosensing. Molecular Biology of the Cell. 28 (23), 3134-3155 (2017).
- Ju, L., Chen, Y., Rushdi, M. N., Chen, W., Zhu, C. Two-Dimensional Analysis of Cross-Junctional Molecular Interaction by Force Probes. Methods in Molecular Biology. 1584, 231-258 (2017).
- Evans, E., Ritchie, K., Merkel, R. Sensitive force technique to probe molecular adhesion and structural linkages at biological interfaces. Biophysical Journal. 68 (6), 2580-2587 (1995).
- Ju, L., Zhu, C. Benchmarks of Biomembrane Force Probe Spring Constant Models. Biophysical Journal. 113 (12), 2842-2845 (2017).
- Evans, E., Ritchie, K., Merkel, R. Sensitive Force Technique to Probe Molecular Adhesion and Structural Linkages at Biological Interfaces. Biophysical Journal. 68, 2580 (1995).
- Fiore, V. F., Ju, L., Chen, Y., Zhu, C., Barker, T. H. Dynamic catch of a Thy-1-alpha5beta1+syndecan-4 trimolecular complex. Nature Communications. 5, 4886 (2014).
- Passam, F., et al. Mechano-redox control of integrin de-adhesion. Elife. 7, (2018).
- Chen, Y., et al. An integrin alphaIIbbeta3 intermediate affinity state mediates biomechanical platelet aggregation. Nature Materials. 18 (7), 760-769 (2019).
- Chen, Y., Lee, H., Tong, H., Schwartz, M., Zhu, C. Force regulated conformational change of integrin αVβ3. Matrix Biology. 60, 70-85 (2017).
- Liu, B., Chen, W., Zhu, C. Molecular force spectroscopy on cells. Annual Review of Physical Chemistry. 66, 427-451 (2015).
- Piper, J. W., Swerlick, R. A., Zhu, C. Determining force dependence of two-dimensional receptor-ligand binding affinity by centrifugation. Biophysical Journal. 74 (1), 492-513 (1998).
- Ju, L., Dong, J. -. f., Cruz, M. A., Zhu, C. The N-terminal flanking region of the A1 domain regulates the force-dependent binding of von Willebrand factor to platelet glycoprotein Ibα. Journal of Biological Chemistry. 288 (45), 32289-32301 (2013).
- Ju, L., Chen, Y., Xue, L., Du, X., Zhu, C. Cooperative unfolding of distinctive mechanoreceptor domains transduces force into signals. Elife. 5, 15447 (2016).

