Análisis constante de resorte molecular mediante espectroscopia de sonda de fuerza de biomembrana
Summary
Una sonda de fuerza de biomembrana (BFP) es una técnica de espectroscopia de fuerza dinámica in situ (DFS). BFP se puede utilizar para medir la constante de resorte de las interacciones moleculares en las células vivas. Este protocolo presenta análisis constantes de resorte para enlaces moleculares detectados por BFP.
Abstract
Una sonda de fuerza de biomembrana (BFP) ha surgido recientemente como una nanoherramienta de espectroscopia de fuerza dinámica (DFS) de superficie celular nativa o in situ que puede medir la cinética de unión molecular única, evaluar las propiedades mecánicas de las interacciones ligando-receptor, visualizar los cambios conformacionales dinámicos de proteínas y dilucidar de manera más emocionante los mecanismos de menosensión celular mediados por el receptor. Más recientemente, BFP se ha utilizado para medir la constante de resorte de los enlaces moleculares. Este protocolo describe el procedimiento paso a paso para realizar el análisis DFS constante de resorte molecular. Específicamente, se discuten dos modos de operación BFP, a saber, los modos Bead-Cell y Bead-Bead. Este protocolo se centra en derivar constantes de resorte del enlace molecular y la célula a partir de datos brutos de DFS.
Introduction
Como una técnica de DFS de células vivas, BFP diseña un glóbulo rojo humano (RBC; Figura 1) en un transductor de fuerza ultrasensible y sintonizable con un rango constante de resorte compatible a 0.1-3 pN/nm1,2,3. Para sondear la interacción ligando-receptor, BFP permite mediciones de DFS a ~1 pN(10 -12 N), ~3 nm(10 -9 m) y ~0.5 ms (10-3 s) en resolución de fuerza, espacial y temporal4,5. Su configuración experimental consiste en dos micropipetas opuestas, a saber, la sonda y el objetivo. La micropipeta de la sonda aspira un glóbulo rojo y una cuenta se pega en su ápice a través de una interacción biotina-estreptavidina. La cuenta está recubierta con el ligando de interés(Figura 1A). La micropipeta objetivo aspira una célula o una cuenta que lleva el receptor de interés, correspondiente a los modos Bead-Cell (Figura 1B) y Bead-Bead (Figura 1C), respectivamente5.
La construcción de BFP, el montaje y los protocolos experimentales DFS se describieron en detalle anteriormente1,6. Brevemente, un ciclo táctil BFP consta de 5 etapas: Aproximación, Incidente, Contacto, Retracción y Disociación(Figura 1D). La posición horizontal del ápice de RBC se denota como ΔxRBC. Al principio, la deformación de RBC no astresada (fuerza cero) ΔxRBC es 0 (Tabla 1). El objetivo es entonces impulsado por un piezotraduclador para inmisionar y retraerse de la cuenta de sonda (Figura 1D). La sonda RBC es comprimida primero por el Target con deformación negativa de RBC ΔxRBC < 0. En un evento de Bond, la etapa de retracción pasa de una fase compresiva a una fase de tracción con deformación positiva deRBC ΔxRBC > 0 ( Figura2C y D). De acuerdo con la ley de Hooke, la fuerza de soporte BFP puede medirse como F = kRBC × ΔxRBC, donde kRBC (Tabla 1)es la constante de resorte RBC del BFP. Tras la ruptura del enlace y la finalización de un ciclo de toque, la cuenta de sonda vuelve a la posición de fuerza cero con ΔxRBC = 0 (Figura 1D).
Para determinar el kRBC,medimos y registramos los radios del orificio interno de la micropipeta de la sonda (Rp), el RBC (R0) y el área de contacto circular (Rc) entre el RBC y la cuenta de la sonda ( Figura1A). Luego se calcula kRBC de acuerdo con el modelo de Evan (Eq. 1)7,8 utilizando un programa LabVIEW que actúa como un instrumento virtual (VI) para operar el BFP(Figura S1A)8,9.
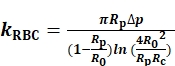 (Eq. 1)
(Eq. 1)
Con un BFP establecido y datos brutos DFS obtenidos, a continuación presentamos cómo analizar la constante de resorte del par ligando-receptor o células. Los datos brutos de DFS sobre la interacción de la proteína glicosilada Thy-1 y la integrina portadora de células K562 α5β1 (Thy-1-α5β1; Figuras 3A y 3B)10 y la de la integrina recubierta de fibrinógeno y cuentas αIIbβ3 (FGN-αIIbβ3; Figura 3C) 11,12 se han utilizado para demostrar los modos de análisis Bead-Cell y Bead-Bead, respectivamente.
Preparación experimental BFP
Para obtener detalles sobre la preparación experimental y la instrumentación de BFP, consulte los protocolos publicados anteriormente3. En resumen, los glóbulos rojos humanos se han biotinilado utilizando la biotina-PEG3500-NHS en el tampón de carbono / bicarbonato. Las proteínas de interés se han acoplado covalentemente a las perlas de vidrio de borosilicato utilizando MAL-PEG3500-NHS en el tampón de fosfato. Para adherirse al RBC biotinilado, la perla de la sonda también está recubierta con estreptavidina (SA) utilizando el MAL-SA. Consulte la Tabla de materiales y la Tabla 2.
Para ensamblar el BFP(Figura 1, izquierda),se utilizará la tercera micropipeta denominada ‘Ayudante’ para entregar la cuenta de sonda y pegarla al vértice1,3del RBC. La interacción covalente entre la cuenta de sonda recubierta de SA y los glóbulos rojos biotinilados es mucho más fuerte que el enlace ligando-receptor de interés. Por lo tanto, la etapa disociada puede interpretarse como la ruptura del enlace ligando-receptor en lugar del desprendimiento de la cuenta de sonda del RBC.
Protocol
Representative Results
Discussion
En resumen, hemos proporcionado un protocolo detallado de análisis de datos para preprocesar los datos brutos de DFS y derivar constantes de resorte molecular en los modos de análisis BFP Bead-Bead y Bead-Cell. Se presentan los modelos biomecánicos y las ecuaciones necesarias para determinar las constantes de resorte moleculares y celulares. Aunque se estudian diferentes integrinas, el kmol medido por el modo Bead-Bead y el modo Bead-Cell posee diferencias de rango significativas<strong class="xfig…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Agradecemos a Guillaume Troadec por la útil discusión, a Zihao Wang por la consulta de hardware y a Sydney Manufacturing Hub, Gregg Suaning y Simon Ringer por el apoyo a nuestra startup de laboratorio. Este trabajo fue apoyado por el Proyecto de Descubrimiento del Consejo australiano de Investigación (DP200101970 – L.A.J.), el Programa de Desarrollo de Capacidades Cardiovasculares de NSW (Early-Mid Career Researcher Grant – L.A.J.), el premio Sydney Research Accelerator (SOAR – L.A.J.), Ramaciotti Foundations Health Investment Grant (2020HIG76 – L.A.J.), National Health and Medical Research Council Ideas Grant (APP2003904 – L.A.J.), y The University of Sydney Faculty of Engineering Startup Fund and Major Equipment Scheme (L.A.J.). Lining Arnold Ju es miembro del Consejo Australiano de Investigación DECRA (DE190100609).
Materials
| 3-Mercaptopropyltrimethoxysilane (MPTMS) | Uct, Specialties, llc | 4420-74-0 | Glass bead functionalization |
| Anhy. Sodium Phosphate Dibasic (Na2HPO4) | Sigma-Aldrich | S7907 | Phosphate buffer preparation |
| BFP data acquisition VI | LabVIEW | BFP control and parameter setting | |
| BFP data analysis VI | LabVIEW | BFP raw data analysis | |
| Biotin-PEG3500-NHS | JenKem | A5026-1 | RBC biotinylation |
| Borosilicate Glass beads | Distrilab Particle Technology, Netherlands | 9002 | Glass bead functionalization |
| Bovine serum albumin | Sigma-Aldrich | A0336 | Ligand functionalization |
| Camera VI | LabVIEW | BFP monitoring | |
| D-glucose | Sigma-Aldrich | G7021 | Tyrode’s buffer preparation |
| Hepes | Sigma-Aldrich | H3375 | Tyrode’s buffer preparation |
| MAL-PEG3500-NHS | JenKem | A5002-1 | Glass bead functionalization |
| Potassium Chloride (KCl) | Sigma-Aldrich | P9541 | Tyrode’s buffer preparation |
| Sodium Bicarbonate (NaHCO3) | Sigma-Aldrich | S5761 | Carbonate/bicarbonate buffer preparation; Tyrode’s buffer preparation |
| Sodium Carbonate (Na2CO3) | Sigma-Aldrich | S2127 | Carbonate/bicarbonate buffer preparation |
| Sodium Chloride (NaCl) | Sigma-Aldrich | S7653 | Tyrode’s buffer preparation |
| Sodium Phosphate Monobasic Monohydrate (NaH2PO4•H2O) | Sigma-Aldrich | S9638 | Phosphate buffer preparation |
| Streptavidin-Maleimide | Sigma-Aldrich | S9415 | Glass bead functionalization |
Riferimenti
- Chen, Y., et al. Fluorescence Biomembrane Force Probe: Concurrent Quantitation of Receptor-ligand Kinetics and Binding-induced Intracellular Signaling on a Single Cell. The Journal of Visualized Experiments. (102), e52975 (2015).
- Su, Q. P., Ju, L. A. Biophysical nanotools for single-molecule dynamics. Biophysics Reviews. 10 (5), 1349-1357 (2018).
- Ju, L. Dynamic Force Spectroscopy Analysis on the Redox States of Protein Disulphide Bonds. Methods in Molecular Biology. 1967, 115-131 (2019).
- An, C., et al. Ultra-stable Biomembrane Force Probe for Accurately Determining Slow Dissociation Kinetics of PD-1 Blockade Antibodies on Single Living Cells. Nano Letters. 20 (7), 5133-5140 (2020).
- Chen, Y., Ju, L., Rushdi, M., Ge, C., Zhu, C. Receptor-mediated cell mechanosensing. Molecular Biology of the Cell. 28 (23), 3134-3155 (2017).
- Ju, L., Chen, Y., Rushdi, M. N., Chen, W., Zhu, C. Two-Dimensional Analysis of Cross-Junctional Molecular Interaction by Force Probes. Methods in Molecular Biology. 1584, 231-258 (2017).
- Evans, E., Ritchie, K., Merkel, R. Sensitive force technique to probe molecular adhesion and structural linkages at biological interfaces. Biophysical Journal. 68 (6), 2580-2587 (1995).
- Ju, L., Zhu, C. Benchmarks of Biomembrane Force Probe Spring Constant Models. Biophysical Journal. 113 (12), 2842-2845 (2017).
- Evans, E., Ritchie, K., Merkel, R. Sensitive Force Technique to Probe Molecular Adhesion and Structural Linkages at Biological Interfaces. Biophysical Journal. 68, 2580 (1995).
- Fiore, V. F., Ju, L., Chen, Y., Zhu, C., Barker, T. H. Dynamic catch of a Thy-1-alpha5beta1+syndecan-4 trimolecular complex. Nature Communications. 5, 4886 (2014).
- Passam, F., et al. Mechano-redox control of integrin de-adhesion. Elife. 7, (2018).
- Chen, Y., et al. An integrin alphaIIbbeta3 intermediate affinity state mediates biomechanical platelet aggregation. Nature Materials. 18 (7), 760-769 (2019).
- Chen, Y., Lee, H., Tong, H., Schwartz, M., Zhu, C. Force regulated conformational change of integrin αVβ3. Matrix Biology. 60, 70-85 (2017).
- Liu, B., Chen, W., Zhu, C. Molecular force spectroscopy on cells. Annual Review of Physical Chemistry. 66, 427-451 (2015).
- Piper, J. W., Swerlick, R. A., Zhu, C. Determining force dependence of two-dimensional receptor-ligand binding affinity by centrifugation. Biophysical Journal. 74 (1), 492-513 (1998).
- Ju, L., Dong, J. -. f., Cruz, M. A., Zhu, C. The N-terminal flanking region of the A1 domain regulates the force-dependent binding of von Willebrand factor to platelet glycoprotein Ibα. Journal of Biological Chemistry. 288 (45), 32289-32301 (2013).
- Ju, L., Chen, Y., Xue, L., Du, X., Zhu, C. Cooperative unfolding of distinctive mechanoreceptor domains transduces force into signals. Elife. 5, 15447 (2016).

