Identificación global de redes de interacción co-traslacional mediante perfiles selectivos de ribosomas
Summary
Las interacciones co-traslacionales juegan un papel crucial en las modificaciones de la cadena naciente, la orientación, el plegado y las vías de ensamblaje. Aquí, describimos el Perfil Selectivo de Ribosomas, un método para el análisis directo in vivo de estas interacciones en el modelo eucariota Saccharomyces cerevisiae.
Abstract
En los últimos años, se ha hecho evidente que los ribosomas no solo decodifican nuestro ARNm, sino que también guían la aparición de la cadena polipeptídica en el entorno celular abarrotado. Los ribosomas proporcionan la plataforma para la unión controlada espacial y cinéticamente de factores dirigidos a la membrana, enzimas modificadoras y chaperonas plegables. Incluso el ensamblaje en complejos oligoméricos de alto orden, así como los pasos de formación de la red proteína-proteína, se descubrieron recientemente que estaban coordinados con la síntesis.
Aquí, describimos el Perfil Selectivo de Ribosomas, un método desarrollado para capturar interacciones co-traslacionales in vivo. Detallaremos los diversos pasos de purificación de afinidad necesarios para capturar complejos de cadena ribosómica-naciente junto con interactores co-traduccionales, así como la extracción de ARNm, la exclusión de tamaño, la transcripción inversa, la secuenciación profunda y los pasos de análisis de big data, necesarios para descifrar las interacciones co-traduccionales en resolución cercana al codón.
Introduction
Elrofiling Ribosome P(SeRP) es el único método, hasta la fecha, que captura y caracteriza las interacciones co-traslacionales, in vivo, de manera directa 1,2,3,4,5,6. SeRP permite el perfil global de las interacciones de cualquier factor con la traducción de ribosomas en resolución de codón cercano 2,7.
El método se basa en la congelación instantánea de las células en crecimiento y la preservación de la traducción activa. Los lisados celulares se tratan con RNasa I para digerir todo el ARNm en la célula, excepto los fragmentos de ARNm protegidos por ribosomas denominados “huellas de ribosomas”. La muestra se divide entonces en dos partes; una parte se utiliza directamente para el aislamiento de todas las huellas ribosómicas celulares, que representan toda la traducción en curso en la célula. La segunda parte se utiliza para la afinidad-purificación del subconjunto específico de ribosomas asociados con un factor de interés, por ejemplo: enzimas modificadoras, factores de translocación, chaperonas plegables e interacciones complejo-ensamblaje. Las huellas ribosómicas purificadas por afinidad se denominan colectivamente el interactoma. Luego, los ARNm protegidos por ribosomas se extraen y se utilizan para la generación de bibliotecas de ADNc, seguidos de una secuenciación profunda.
El análisis comparativo de las muestras totales de translatoma e interactoma permite la identificación de todos los orfs que se asocian con el factor de interés, así como la caracterización de cada perfil de interacción orf. Este perfil informa las secuencias precisas de inicio y terminación del compromiso a partir de las cuales se pueden inferir los codones decodificados y los residuos respectivos de la cadena polipeptídica emergente, así como sobre las variaciones de velocidad del ribosoma durante la interacción 7,8. La figura 1 muestra el protocolo como un esquema.
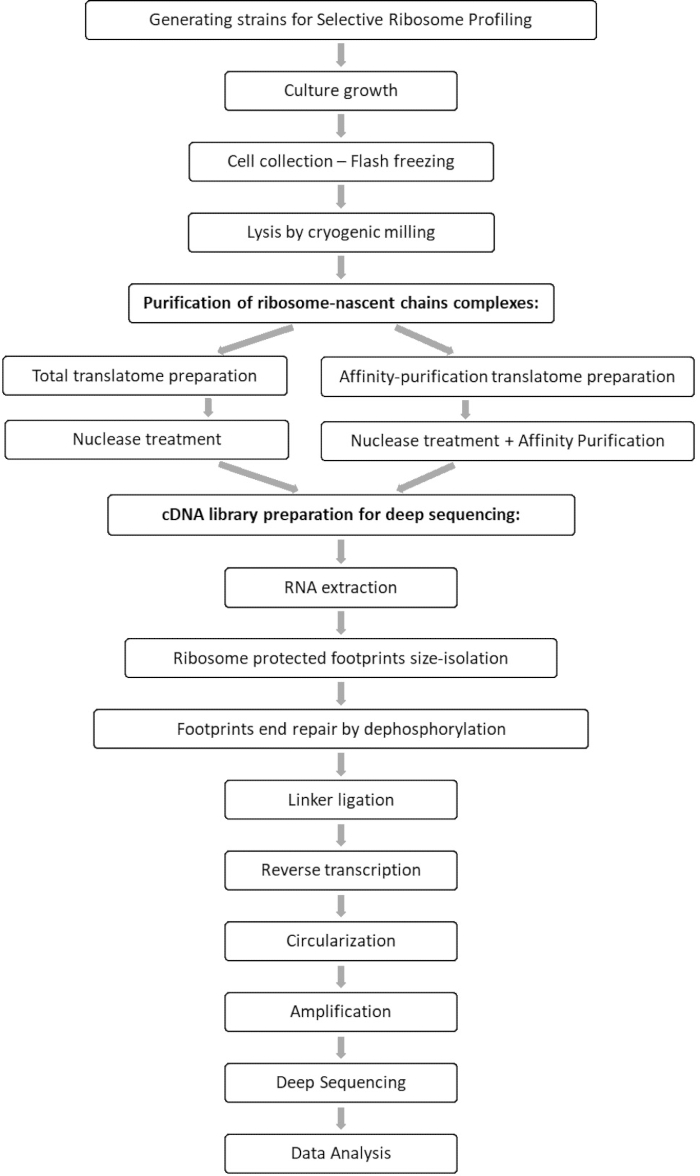
Figura 1: Una visión general del protocolo SeRP. Este protocolo se puede realizar en su totalidad en un plazo de 7-10 días. Haga clic aquí para ver una versión más grande de esta figura.
Protocol
Representative Results
Discussion
Aquí, el protocolo detalla el enfoque de perfil selectivo de ribosomas para capturar interacciones co-traduccionales en resolución cercana al codón. A medida que el ribosoma se eleva como un centro para coordinar la aparición de la cadena naciente en el citoplasma abarrotado, este es un método crucial para identificar y caracterizar las diversas interacciones co-traduccionales necesarias para garantizar un proteoma funcional, así como para estudiar diversas enfermedades. Hasta la fecha, SeRP es el único método q…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Nos gustaría agradecer a todos los miembros del laboratorio por las fructíferas discusiones y a Muhammad Makhzumy por la lectura crítica del manuscrito. Este trabajo fue financiado por la subvención 2106/20 de la ISF (Fundación Israelí para la Ciencia).
Materials
| 3'-Phosphorylated 28 nt RNA control oligonucleotide | IDT | custom order | RNase free HPLC purification; 5'-AUGUAGUCGGAGUCGAGGCGC GACGCGA/3Phos/-3' |
| Absolute ethanol | VWR | 20821 | |
| Acid phenol–chloroform | Ambion | AM9722 | |
| Antibody: mouse monclonal anti-HA | Merck | 11583816001 | 12CA5 |
| Aprotinin | Roth | A162.3 | |
| ATP* | NEB | P0756S | 10 mM |
| Bacto agar | BD | 214030 | |
| Bacto peptone | BD | 211820 | |
| Bacto tryptone | BD | 211699 | |
| Bacto yeast extract | BD | 212720 | |
| Bestatin hydrochloride | Roth | 2937.2 | |
| Chloroform | Merck | 102445 | |
| CircLigase II ssDNA Ligase* | Epicentre | CL9025K | 100 U/μL |
| Colloidal Coomassie staining solution | Roth | 4829 | |
| cOmplete, EDTA-free protease inhibitor cocktail tablets | Roche Diagnostics | 29384100 | |
| Cycloheximide | Biological Industries | A0879 | |
| DEPC treated and sterile filtered water* | Sigma | 95284 | |
| D-Glucose anhydrous | Merck | G5767-500G | |
| Diethylpyrocarbonate | Roth | K028 | |
| Dimethylsulfoxide* | Sigma-Aldrich | 276855 | |
| DNA ladder, 10 bp O'RangeRuler* | Thermo Fisher Scientific | SM1313 | |
| DNA loading dye* | Thermo Fisher Scientific | R0631 | 6× |
| DNase I, recombinant | Roche | 4716728001 | RNAse free |
| dNTP solution set* | NEB | N0446S | |
| EDTA* | Roth | 8043 | |
| Glycerol | VWR | 24388.260. | |
| Glycine solution | Sigma-Aldrich | 67419-1ML-F | 1 M |
| GlycoBlue | Ambion | AM9516 | 15 mg/mL |
| HEPES | Roth | HN78.3 | |
| HF Phusion polymerase* | NEB | M0530L | |
| HK from S. cerevisiae | Sigma-Aldrich | H6380-1.5KU | |
| Hydrochloric acid | AppliChem | A1305 | |
| Isopropanol | Sigma-Aldrich | 33539 | |
| Isopropyl β-D-1-thiogalactopyranoside | Roth | CN08 | |
| Kanamycin | Roth | T832.4 | |
| KCl | Roth | 6781.1 | |
| KH2PO4 | Roth | 3904.1 | |
| Leupeptin | Roth | CN33.4 | |
| Linker L(rt) | IDT | custom order | |
| Liquid nitrogen | |||
| MgCl2 | Roth | KK36.3 | |
| Na2HPO4 | Roth | P030.2 | |
| Na2HPO4·2H2O | Roth | T879.3 | |
| NaCl* | Invitrogen | AM97606 | 5 M |
| NaH2PO4·H2O | Roth | K300.2 | |
| NHS-activated Sepharose 4 fast-flow beads | GE Life Sciences | 17090601 | |
| Nonidet P 40 substitute | Sigma | 74385 | |
| Pepstatin A | Roth | 2936.2 | |
| Phenylmethyl sulfonyl fluoride | Roth | 6367 | |
| Precast gels | Bio-Rad | 5671034 | 10% and 12% |
| RNase I | Ambion | AM2294 | |
| SDS, 20% | Ambion | AM9820 | RNase free |
| Sodium acetate* | Ambion | AM9740 | 3 M, pH 5.5 |
| Sodium azide | Merck | S8032-100G | |
| Sodium chloride | Roth | 9265 | |
| Sodium hydroxide* | Sigma | S2770 | 1 N |
| Sucrose | Sigma-Aldrich | 16104 | |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Superscript III Reverse Transciptase* | Invitrogen | 18080-044 | |
| SYBR Gold* | Invitrogen | S11494 | |
| T4 polynucleotide kinase* | NEB | M0201L | |
| T4 RNA ligase 2* | NEB | M0242L | |
| TBE polyacrylamide gel* | Novex | EC6215BOX | 8% |
| TBE–urea polyacrylamide gel* | Novex | EC68752BOX | 10% |
| TBE–urea polyacrylamide gel* | Novex | EC6885BOX | 15% |
| TBE–urea sample buffer* | Novex | LC6876 | 2× |
| Tris | Roth | 4855 | |
| Tris* | Ambion | AM9851 | 1 M, pH 7.0 |
| Tris* | Ambion | AM9856 | 1 M, pH 8.0 |
| UltraPure 10× TBE buffer* | Invitrogen | 15581-044 | |
| * – for library preparation | |||
| gasket and spring clamp , 90 mm, | Millipore | XX1009020 | |
| ground joint flask 1 L , | Millipore | XX1504705 |
Riferimenti
- Oh, E., et al. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Shiber, A., et al. Cotranslational assembly of protein complexes in eukaryotes revealed by ribosome profiling. Nature. 561 (7722), 268-272 (2018).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature Protocols. 8 (11), 2212-2239 (2013).
- Galmozzi, C. V., Merker, D., Friedrich, U. A., Döring, K., Kramer, G. Selective ribosome profiling to study interactions of translating ribosomes in yeast. Nature Protocols. , (2019).
- Knorr, A. G., et al. Ribosome-NatA architecture reveals that rRNA expansion segments coordinate N-terminal acetylation. Nature Structural and Molecular Biology. 26 (1), 35-39 (2019).
- Matsuo, Y., Inada, T. The ribosome collision sensor Hel2 functions as preventive quality control in the secretory pathway. Cell Reports. 34 (12), (2021).
- Döring, K., et al. Profiling Ssb-Nascent chain interactions reveals principles of Hsp70-assisted folding. Cell. , (2017).
- Chartron, J. W., Hunt, K. C. L., Frydman, J. Cotranslational signal-independent SRP preloading during membrane targeting. Nature. 536 (7615), 224-228 (2016).
- Janke, C., et al. A versatile toolbox for PCR-based tagging of yeast genes: New fluorescent proteins, more markers and promoter substitution cassettes. Yeast. 21 (11), 947-962 (2004).
- Levi, O., Arava, Y. Expanding the CRISPR/Cas9 Toolbox for Gene Engineering in S. cerevisiae. Current Microbiology. 77 (3), 468-478 (2020).
- Giannoukos, G., et al. Efficient and robust RNA-seq process for cultured bacteria and complex community transcriptomes. Genome Biology. 13 (3), 23 (2012).
- . Illumina Index Adapters – Pooling Guide Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/experiment-design/index-adapters-pooling-guide-1000000041074-05.pdf (2019)
- Kanagawa, T. Bias and artifacts in multitemplate polymerase chain reactions (PCR). Journal of Bioscience and Bioengineering. 96 (4), 317-323 (2003).
- Bertolini, M., et al. Interactions between nascent proteins translated by adjacent ribosomes drive homomer assembly. Science. 371 (6524), (2021).
- Kramer, G., Shiber, A., Bukau, B. Mechanisms of cotranslational maturation of newly synthesized proteins. Annual Review of Biochemistry. 88, 337-364 (2019).
- Joazeiro, C. A. P. Mechanisms and functions of ribosome-associated protein quality control. Nature Reviews Molecular Cell Biology. 20 (6), 368-383 (2019).
- Beaupere, C., Chen, R. B., Pelosi, W., Labunskyy, V. M. Genome-wide quantification of translation in budding yeast by ribosome profiling. Journal of Visualized Experiments: JoVE. (130), e56820 (2017).

