Identificazione globale delle reti di interazione co-traslazionale mediante profilazione selettiva dei ribosomi
Summary
Le interazioni co-traslazionali svolgono un ruolo cruciale nelle modifiche della catena nascente, nel targeting, nella piegatura e nei percorsi di assemblaggio. Qui, descriviamo il Selective Ribosome Profiling, un metodo per l’analisi diretta in vivo di queste interazioni nel modello eucariota Saccharomyces cerevisiae.
Abstract
Negli ultimi anni, è diventato evidente che i ribosomi non solo decodificano il nostro mRNA, ma guidano anche l’emergere della catena polipeptidica nell’ambiente cellulare affollato. I ribosomi forniscono la piattaforma per il legame controllato spazialmente e cineticamente di fattori di targeting della membrana, enzimi modificanti e chaperoni pieghevoli. Anche l’assemblaggio in complessi oligomerici di alto ordine, così come le fasi di formazione della rete proteina-proteina, sono stati recentemente scoperti per essere coordinati con la sintesi.
Qui, descriviamo il Selective Ribosome Profiling, un metodo sviluppato per catturare le interazioni co-traduzionali in vivo. Descriveremo in dettaglio le varie fasi di purificazione dell’affinità necessarie per catturare complessi ribosomi-catena nascente insieme agli interattori co-traduzionali, nonché l’estrazione dell’mRNA, l’esclusione delle dimensioni, la trascrizione inversa, il sequenziamento profondo e le fasi di analisi dei big data, necessarie per decifrare le interazioni co-traduzionali nella risoluzione quasi codone.
Introduction
Selective Ribosome Profiling (SeRP) è l’unico metodo, ad oggi, che cattura e caratterizza le interazioni co-traslazionali, in vivo, in modo diretto 1,2,3,4,5,6. SeRP consente la profilazione globale delle interazioni di qualsiasi fattore con la traduzione dei ribosomi nella risoluzione vicino al codone 2,7.
Il metodo si basa sul congelamento flash delle cellule in crescita e sulla conservazione della traduzione attiva. I lisati cellulari vengono quindi trattati con RNasi I per digerire tutto l’mRNA nella cellula ad eccezione dei frammenti di mRNA protetti dai ribosomi chiamati “impronte di ribosomi”. Il campione viene quindi diviso in due parti; una parte viene utilizzata direttamente per l’isolamento di tutte le impronte ribosomiali cellulari, rappresentando tutta la traduzione in corso nella cellula. La seconda parte viene utilizzata per l’affinità-purificazione del sottoinsieme specifico di ribosomi associati a un fattore di interesse, ad esempio: modifica di enzimi, fattori di traslocazione, chaperoni di piegatura e interazioni complesso-assemblaggio. Le impronte ribosomiali purificate dall’affinità sono collettivamente chiamate interattoma. Quindi, gli mRNA protetti dai ribosomi vengono estratti e utilizzati per la generazione di librerie di cDNA, seguiti dal sequenziamento profondo.
L’analisi comparativa dei campioni totali di translatome e interactome consente l’identificazione di tutti gli orfs che si associano al fattore di interesse, nonché la caratterizzazione di ciascun profilo di interazione orf. Questo profilo riporta le precise sequenze di insorgenza e terminazione dell’incarico da cui si possono dedurre i codoni decodificati e i rispettivi residui della catena polipeptidica emergente, nonché sulle variazioni di velocità del ribosoma durante l’interazione 7,8. La Figura 1 illustra il protocollo come schematico.
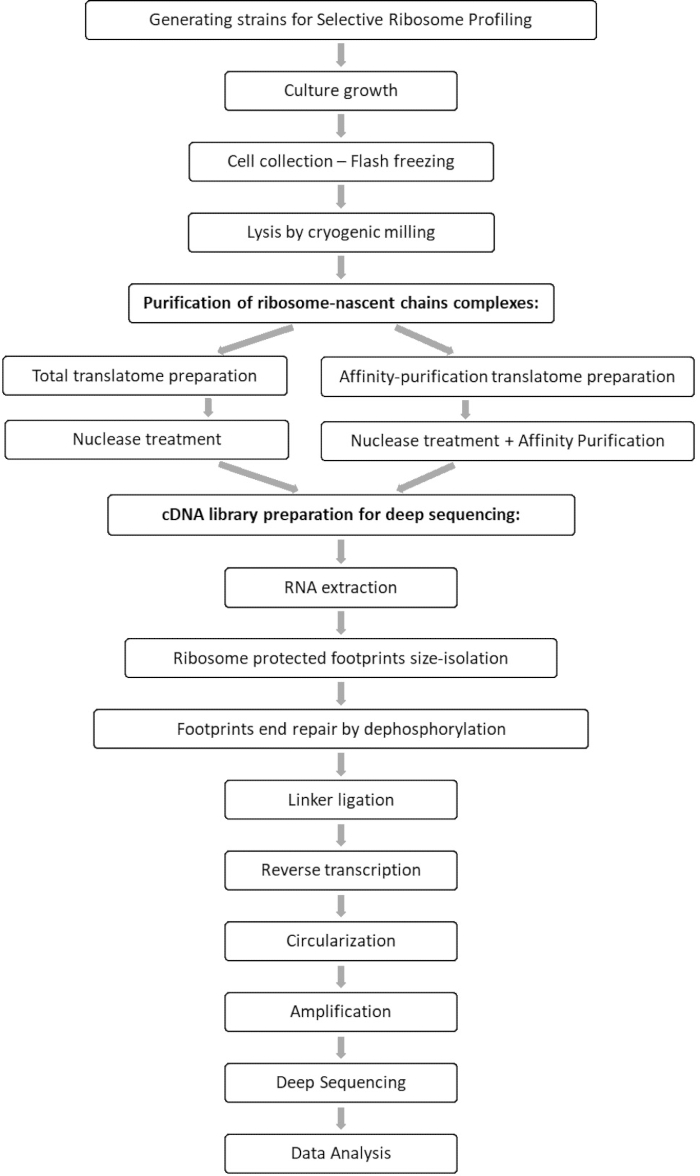
Figura 1: Panoramica del protocollo SeRP. Questo protocollo può essere eseguito nella sua interezza entro 7-10 giorni. Fare clic qui per visualizzare una versione più grande di questa figura.
Protocol
Representative Results
Discussion
Qui, il protocollo descrive in dettaglio l’approccio Selective Ribosome Profiling per l’acquisizione di interazioni co-traslazionali in risoluzione quasi codone. Mentre il ribosoma sale come hub per coordinare l’emergenza della catena nascente nel citoplasma affollato, questo è un metodo cruciale per identificare e caratterizzare le varie interazioni co-traduzionali necessarie per garantire un proteoma funzionale, nonché per studiare varie malattie. Ad oggi, la SeRP è l’unico metodo in grado di catturare e caratteriz…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Vorremmo ringraziare tutti i membri del laboratorio per le fruttuose discussioni e Muhammad Makhzumy per la lettura critica del manoscritto. Questo lavoro è stato finanziato dalla sovvenzione ISF (Israeli Science Foundation) 2106/20.
Materials
| 3'-Phosphorylated 28 nt RNA control oligonucleotide | IDT | custom order | RNase free HPLC purification; 5'-AUGUAGUCGGAGUCGAGGCGC GACGCGA/3Phos/-3' |
| Absolute ethanol | VWR | 20821 | |
| Acid phenol–chloroform | Ambion | AM9722 | |
| Antibody: mouse monclonal anti-HA | Merck | 11583816001 | 12CA5 |
| Aprotinin | Roth | A162.3 | |
| ATP* | NEB | P0756S | 10 mM |
| Bacto agar | BD | 214030 | |
| Bacto peptone | BD | 211820 | |
| Bacto tryptone | BD | 211699 | |
| Bacto yeast extract | BD | 212720 | |
| Bestatin hydrochloride | Roth | 2937.2 | |
| Chloroform | Merck | 102445 | |
| CircLigase II ssDNA Ligase* | Epicentre | CL9025K | 100 U/μL |
| Colloidal Coomassie staining solution | Roth | 4829 | |
| cOmplete, EDTA-free protease inhibitor cocktail tablets | Roche Diagnostics | 29384100 | |
| Cycloheximide | Biological Industries | A0879 | |
| DEPC treated and sterile filtered water* | Sigma | 95284 | |
| D-Glucose anhydrous | Merck | G5767-500G | |
| Diethylpyrocarbonate | Roth | K028 | |
| Dimethylsulfoxide* | Sigma-Aldrich | 276855 | |
| DNA ladder, 10 bp O'RangeRuler* | Thermo Fisher Scientific | SM1313 | |
| DNA loading dye* | Thermo Fisher Scientific | R0631 | 6× |
| DNase I, recombinant | Roche | 4716728001 | RNAse free |
| dNTP solution set* | NEB | N0446S | |
| EDTA* | Roth | 8043 | |
| Glycerol | VWR | 24388.260. | |
| Glycine solution | Sigma-Aldrich | 67419-1ML-F | 1 M |
| GlycoBlue | Ambion | AM9516 | 15 mg/mL |
| HEPES | Roth | HN78.3 | |
| HF Phusion polymerase* | NEB | M0530L | |
| HK from S. cerevisiae | Sigma-Aldrich | H6380-1.5KU | |
| Hydrochloric acid | AppliChem | A1305 | |
| Isopropanol | Sigma-Aldrich | 33539 | |
| Isopropyl β-D-1-thiogalactopyranoside | Roth | CN08 | |
| Kanamycin | Roth | T832.4 | |
| KCl | Roth | 6781.1 | |
| KH2PO4 | Roth | 3904.1 | |
| Leupeptin | Roth | CN33.4 | |
| Linker L(rt) | IDT | custom order | |
| Liquid nitrogen | |||
| MgCl2 | Roth | KK36.3 | |
| Na2HPO4 | Roth | P030.2 | |
| Na2HPO4·2H2O | Roth | T879.3 | |
| NaCl* | Invitrogen | AM97606 | 5 M |
| NaH2PO4·H2O | Roth | K300.2 | |
| NHS-activated Sepharose 4 fast-flow beads | GE Life Sciences | 17090601 | |
| Nonidet P 40 substitute | Sigma | 74385 | |
| Pepstatin A | Roth | 2936.2 | |
| Phenylmethyl sulfonyl fluoride | Roth | 6367 | |
| Precast gels | Bio-Rad | 5671034 | 10% and 12% |
| RNase I | Ambion | AM2294 | |
| SDS, 20% | Ambion | AM9820 | RNase free |
| Sodium acetate* | Ambion | AM9740 | 3 M, pH 5.5 |
| Sodium azide | Merck | S8032-100G | |
| Sodium chloride | Roth | 9265 | |
| Sodium hydroxide* | Sigma | S2770 | 1 N |
| Sucrose | Sigma-Aldrich | 16104 | |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Superscript III Reverse Transciptase* | Invitrogen | 18080-044 | |
| SYBR Gold* | Invitrogen | S11494 | |
| T4 polynucleotide kinase* | NEB | M0201L | |
| T4 RNA ligase 2* | NEB | M0242L | |
| TBE polyacrylamide gel* | Novex | EC6215BOX | 8% |
| TBE–urea polyacrylamide gel* | Novex | EC68752BOX | 10% |
| TBE–urea polyacrylamide gel* | Novex | EC6885BOX | 15% |
| TBE–urea sample buffer* | Novex | LC6876 | 2× |
| Tris | Roth | 4855 | |
| Tris* | Ambion | AM9851 | 1 M, pH 7.0 |
| Tris* | Ambion | AM9856 | 1 M, pH 8.0 |
| UltraPure 10× TBE buffer* | Invitrogen | 15581-044 | |
| * – for library preparation | |||
| gasket and spring clamp , 90 mm, | Millipore | XX1009020 | |
| ground joint flask 1 L , | Millipore | XX1504705 |
Riferimenti
- Oh, E., et al. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Shiber, A., et al. Cotranslational assembly of protein complexes in eukaryotes revealed by ribosome profiling. Nature. 561 (7722), 268-272 (2018).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature Protocols. 8 (11), 2212-2239 (2013).
- Galmozzi, C. V., Merker, D., Friedrich, U. A., Döring, K., Kramer, G. Selective ribosome profiling to study interactions of translating ribosomes in yeast. Nature Protocols. , (2019).
- Knorr, A. G., et al. Ribosome-NatA architecture reveals that rRNA expansion segments coordinate N-terminal acetylation. Nature Structural and Molecular Biology. 26 (1), 35-39 (2019).
- Matsuo, Y., Inada, T. The ribosome collision sensor Hel2 functions as preventive quality control in the secretory pathway. Cell Reports. 34 (12), (2021).
- Döring, K., et al. Profiling Ssb-Nascent chain interactions reveals principles of Hsp70-assisted folding. Cell. , (2017).
- Chartron, J. W., Hunt, K. C. L., Frydman, J. Cotranslational signal-independent SRP preloading during membrane targeting. Nature. 536 (7615), 224-228 (2016).
- Janke, C., et al. A versatile toolbox for PCR-based tagging of yeast genes: New fluorescent proteins, more markers and promoter substitution cassettes. Yeast. 21 (11), 947-962 (2004).
- Levi, O., Arava, Y. Expanding the CRISPR/Cas9 Toolbox for Gene Engineering in S. cerevisiae. Current Microbiology. 77 (3), 468-478 (2020).
- Giannoukos, G., et al. Efficient and robust RNA-seq process for cultured bacteria and complex community transcriptomes. Genome Biology. 13 (3), 23 (2012).
- . Illumina Index Adapters – Pooling Guide Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/experiment-design/index-adapters-pooling-guide-1000000041074-05.pdf (2019)
- Kanagawa, T. Bias and artifacts in multitemplate polymerase chain reactions (PCR). Journal of Bioscience and Bioengineering. 96 (4), 317-323 (2003).
- Bertolini, M., et al. Interactions between nascent proteins translated by adjacent ribosomes drive homomer assembly. Science. 371 (6524), (2021).
- Kramer, G., Shiber, A., Bukau, B. Mechanisms of cotranslational maturation of newly synthesized proteins. Annual Review of Biochemistry. 88, 337-364 (2019).
- Joazeiro, C. A. P. Mechanisms and functions of ribosome-associated protein quality control. Nature Reviews Molecular Cell Biology. 20 (6), 368-383 (2019).
- Beaupere, C., Chen, R. B., Pelosi, W., Labunskyy, V. M. Genome-wide quantification of translation in budding yeast by ribosome profiling. Journal of Visualized Experiments: JoVE. (130), e56820 (2017).

