Autofluoreszenzbildgebung zur Beurteilung des Zellstoffwechsels
Summary
Dieses Protokoll beschreibt die Fluoreszenzbildgebung und Analyse der endogenen metabolischen Coenzyme, des reduzierten Nicotinamidadenins (Phosphat) -Dinukleotids (NAD (P) H) und des oxidierten Flavinadenindinukleotids (FAD). Die Autofluoreszenzbildgebung von NAD(P)H und FAD bietet eine markierungsfreie, zerstörungsfreie Methode zur Beurteilung des Zellstoffwechsels.
Abstract
Der Zellstoffwechsel ist der Prozess, durch den Zellen Energie erzeugen, und viele Krankheiten, einschließlich Krebs, sind durch einen abnormalen Stoffwechsel gekennzeichnet. Reduziertes Nicotinamidadenin (Phosphat) Dinukleotid (NAD(P)H) und oxidiertes Flavinadenindinukleotid (FAD) sind Coenzyme von Stoffwechselreaktionen. NAD(P)H und FAD weisen Autofluoreszenz auf und können spektral durch Anregung und Emissionswellenlängen isoliert werden. Beide Coenzyme, NAD (P) H und FAD, können entweder in einer freien oder proteingebundenen Konfiguration vorliegen, von denen jedes eine ausgeprägte Fluoreszenzlebensdauer hat – die Zeit, für die das Fluorophor im angeregten Zustand verbleibt. Die Fluoreszenz-Lebensdauerbildgebung (FLIM) ermöglicht die Quantifizierung der Fluoreszenzintensität und Lebensdauer von NAD(P)H und FAD für eine markierungsfreie Analyse des Zellstoffwechsels. Fluoreszenzintensitäts- und Lebensdauermikroskope können für die Bildgebung von NAD(P)H und FAD optimiert werden, indem die geeigneten Anregungs- und Emissionswellenlängen ausgewählt werden. Metabolische Störungen durch Cyanid verifizieren Autofluoreszenz-Bildgebungsprotokolle, um metabolische Veränderungen in Zellen zu erkennen. Dieser Artikel demonstriert die Technik der Autofluoreszenzbildgebung von NAD(P)H und FAD zur Messung des Zellstoffwechsels.
Introduction
Stoffwechsel ist der zelluläre Prozess der Energieerzeugung. Der Zellstoffwechsel umfasst mehrere Wege, einschließlich Glykolyse, oxidative Phosphorylierung und Glutaminolyse. Gesunde Zellen nutzen diese Stoffwechselwege, um Energie für die Proliferation und Funktion zu erzeugen, wie zum Beispiel die Produktion von Zytokinen durch Immunzellen. Viele Krankheiten, einschließlich Stoffwechselstörungen, Krebs und Neurodegeneration, sind durch einen veränderten Zellstoffwechsel gekennzeichnet1. Zum Beispiel haben einige Krebszelltypen erhöhte Glykolyseraten, selbst in Gegenwart von Sauerstoff, um Moleküle für die Synthese von Nukleinsäuren, Proteinen und Lipiden zu erzeugen2,3. Dieses Phänomen, das als Warburg-Effekt bekannt ist, ist ein Kennzeichen vieler Krebsarten, einschließlich Brustkrebs, Lungenkrebs und Glioblastome4. Aufgrund der Veränderungen des Zellstoffwechsels, die mit dem Fortschreiten der Krebserkrankung verbunden sind, kann der Zellstoffwechsel ein Surrogat-Biomarker für die Arzneimittelreaktion sein5,6. Darüber hinaus ist das Verständnis der Arzneimittelwirksamkeit auf zellulärer Ebene von entscheidender Bedeutung, da die Zellheterogenität bei Individuen zu unterschiedlichen Arzneimittelreaktionen führen kann7,8.
Technologien, die Veränderungen im Zellstoffwechsel identifizieren und quantifizieren, sind für Studien zu Krebs und Medikamentenreaktion unerlässlich. Chemische und Proteinanalysen werden verwendet, um den Stoffwechsel von Zellen oder Geweben zu bewerten, aber es fehlen Einzelzellauflösungen und räumliche Informationen. Metabolische Plattenleser-basierte Assays können den pH- und Sauerstoffverbrauch in der Probe im Laufe der Zeit und die anschließende metabolische Störung durch Chemikalien messen. Über den pH-Wert kann die extrazelluläre Versauerungsrate (ECAR) berechnet werden, die einen Einblick in die glykolytische Aktivität der Zellen gibt9. Bildgebende Ganzkörpermethoden, einschließlich 2-[Fluor-18] Fluor-D-Glucose-Positronen-Emissions-Tomographie (FDG PET) und Magnetresonanzspektroskopie (MRS), sind nichtinvasive Bildgebungsmodalitäten, die klinisch verwendet werden, um Tumorrezidive und Arzneimittelwirksamkeit durch metabolische Messungen zu identifizieren10,11,12,13,14.
FDG-PET bildet die Gewebeaufnahme von FDG, einem radioaktiv markierten Glukoseanalogon, ab. Die erhöhte Aufnahme von FDG-PET durch Tumore im Verhältnis zum umgebenden Gewebe ist auf den Warburg-Effekt zurückzuführen12,13. MRS bildet gemeinsame Kerne von Molekülen ab, die für den Stoffwechsel verwendet werden, wie 13C und 31P, und kann dynamische Informationen darüber erhalten, wie sich der Stoffwechsel als Reaktion auf Reize wie Bewegung oder Essen verändert14. Obwohl FDG-PET und MRS klinisch eingesetzt werden können, fehlt diesen Technologien die räumliche Auflösung, um die intratumorale Heterogenität aufzulösen. Ebenso werden Sauerstoffverbrauchsmessungen an einer Massenpopulation von Zellen durchgeführt. Die Autofluoreszenzbildgebung überwindet das räumliche Auflösungshindernis dieser Technologien und bietet eine nichtinvasive Methode zur Quantifizierung des Zellstoffwechsels.
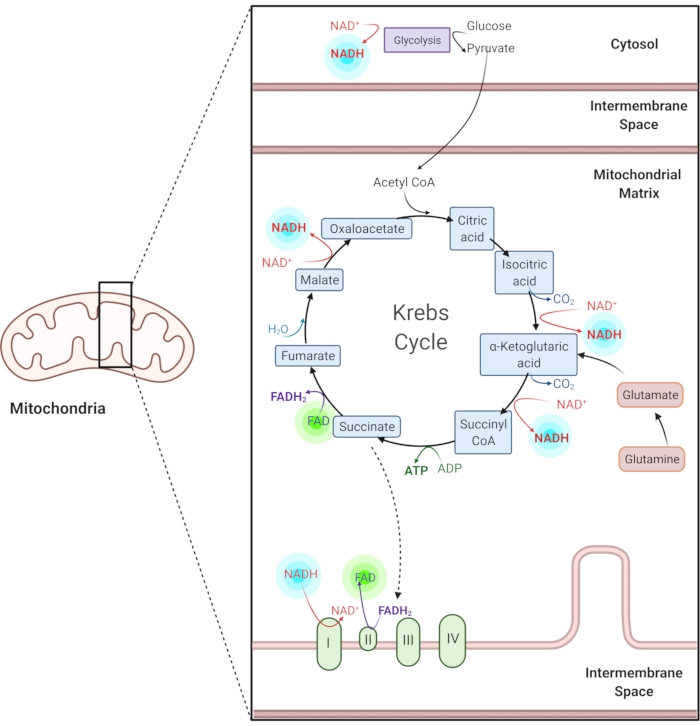
Abbildung 1: NADH und FAD in gemeinsamen Stoffwechselwegen. NADH und FAD sind Coenzyme, die in der Glykolyse, im Krebszyklus und in der Elektronentransportkette verwendet werden. Die Autofluoreszenzbildgebung dieser Moleküle liefert Informationen über den Zellstoffwechsel. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Reduziertes Nicotinamidadenin (Phosphat) Dinukleotid (NAD(P)H) und oxidiertes Flavinadenindinukleotid (FAD) sind Coenzyme von Stoffwechselreaktionen, einschließlich Glykolyse, oxidativer Phosphorylierung und Glutaminolyse (Abbildung 1). Sowohl NAD(P)H als auch FAD sind autofluoreszierend und bieten einen endogenen Kontrast für die Fluoreszenzbildgebung1,15. NADPH hat ähnliche fluoreszierende Eigenschaften wie NADH. Aus diesem Grund wird NAD(P)H häufig verwendet, um das kombinierte Signal von NADH und NADPH2,16 darzustellen.
Die Fluoreszenzlebensdauerbildgebung (FLIM) quantifiziert die Fluoreszenzlebensdauer oder die Zeit, für die sich ein Fluorophor im angeregten Zustand befindet. Fluoreszenzlebensdauern reagieren auf die Mikroumgebung der Fluorophore und liefern Informationen über den Zellstoffwechsel17. NAD(P)H und FAD können in Zellen entweder in proteingebundenen oder freien Konformationen existieren, von denen jede eine andere Lebensdauer hat. Freies NAD(P)H hat eine kürzere Lebensdauer als proteingebundenes NAD(P)H; Umgekehrt hat die freie FAD eine längere Lebensdauer als die gebundenen FAD18,19. Die Lebensdauern und Lebensdauerkomponentengewichte können aus Fluoreszenz-Lebensdauerzerfallsdaten durch Gl. (1)20 quantifiziert werden:
I(t) = α 1e-t/τ1 + α 2e-t/τ2 + C (1)
Eq (1) stellt die normierte Fluoreszenzintensität als Funktion der Zeit dar. Die α 1 und α 2 in dieser Gleichung stellen die proportionalen Komponenten der kurzen und langen Lebensdauer (α 1+ α 2=1) dar, τ1 und τ2 die kurze bzw. lange Lebensdauer und C das Hintergrundlicht7,20. Die amplitudengewichtete Lebensdauer, hier dargestellt als τm, wird mit Gleichung (2) berechnet.
τm= α 1τ1+ α 2τ2 (2)
Eine mittlere Lebensdauer kann durch Mittelung von “t” über den Intensitätszerfall des Fluorophors berechnet werden, was für einen zweiexponentiellen Zerfall durch Gl. (3)17,21 dargestellt wird.
τ*m= (α 1τ12+ α 2τ22)/ (α 1τ1+ α 2τ2) (3)
Ein Fluoreszenzintensitätsbild kann aus dem Lebensdauerbild berechnet werden, indem der Fluoreszenzlebensdauerzerfall integriert wird. Die Autofluoreszenzbildgebung ist eine zerstörungsfreie und markierungsfreie Methode, mit der der Stoffwechsel lebender Zellen mit subzellulärer Auflösung charakterisiert werden kann. Das optische Redoxverhältnis liefert eine optische Analogmetrik des chemischen Redoxzustands der Zelle und wird als Verhältnis von NAD(P)H- und FAD-Intensitäten berechnet. Obwohl die Formel zur Berechnung des optischen Redoxverhältnisses nicht standardisiert ist22,23,24,25, wird sie hier als die Intensität von FAD über die kombinierten Intensitäten von NAD(P)H und FAD definiert. Diese Definition wird verwendet, weil die summierte Intensität im Nenner die Metrik zwischen 0 und 1 normalisiert und das erwartete Ergebnis der Cyanidhemmung eine Abnahme des Redoxverhältnisses ist. Die Fluoreszenzlebensdauern von freiem NAD(P)H und FAD liefern Einblicke in Veränderungen in der metabolischen Lösungsmittelmikroumgebung, einschließlich pH-Wert, Temperatur, Nähe zu Sauerstoff und Osmolarität17.
Veränderungen in der Fluoreszenzlebensdauer der gebundenen Fraktionen von NAD(P)H und FAD können auf die Nutzung des Stoffwechselwegs und den substratspezifischen Stoffwechsel hinweisen26. Komponentengewichte können für Änderungen der freien zur gebundenen Fraktion der Coenzyme interpretiert werden18,19. Insgesamt ermöglichen diese quantitativen Autofluoreszenz-Lebenszeitmetriken die Analyse des Zellstoffwechsels, und die Autofluoreszenz-Bildgebung wurde zur Identifizierung von Neoplasmen aus normalem Gewebe verwendet27,28, zur Charakterisierung von Stammzellen29,30, zur Bewertung der Immunzellfunktion31,32,33,34,35, zur Messung der neurologischen Aktivität36, 37,38, und Verständnis der Arzneimittelwirksamkeit bei Krebsarten wie Brustkrebs und Kopf-Hals-Krebs21,39,40,41,42. Hochauflösende Autofluoreszenzbildgebung kann mit Bildsegmentierung für die Einzelzellanalyse und Quantifizierung der intrapopulationären Heterogenität kombiniert werden43,44,45,46,47.
NAD(P)H und FAD können auf Einzelphotonen- oder Multiphotonen-Fluoreszenzmikroskopen abgebildet werden, die für die Intensitäts- oder Lebensdauerbildgebung konfiguriert sind. Bei Einzelphotonenmikroskopen werden NAD(P)H und FAD aufgrund üblicher Laserquellen bei diesen Wellenlängen typischerweise bei Wellenlängen von 375-405 nm bzw. 488 nm angeregt48. Bei der Zwei-Photonen-Fluoreszenzanregung werden NAD(P)H und FAD bei Wellenlängen von etwa 700 bis 750 nm bzw. 700 bis 900 nm bzw. 15,49 anregen. Sobald die Fluorophore angeregt sind, emittieren NAD(P)H und FAD Photonen bei Wellenlängen zwischen ~410 nm bis ~490 nm bzw. ~510 nm bis ~640 nm15. Die maximalen Emissionswellenlängen NAD(P)H und FAD betragen etwa 450 nm bzw. 535 nm48.
Aufgrund ihrer unterschiedlichen Anregungs- und Emissionswellenlängen kann die Fluoreszenz der beiden metabolischen Coenzyme spektral isoliert werden. Ein Verständnis der spektralen Eigenschaften von NAD(P)H und FAD ist für das Design und die Optimierung von Autofluoreszenz-Bildgebungsprotokollen notwendig. Cyanid ist ein Elektrontransportkette (ETC) -Komplex-IV-Inhibitor. Die Auswirkungen von Cyanid auf den Zellstoffwechsel und die Autofluoreszenzintensitäten und Lebensdauern von NAD(P)H und FAD in Zellen sind gut charakterisiert27,40. Daher ist ein Cyanid-Störungsexperiment ein wirksames Mittel zur Validierung von NAD(P)H- und FAD-Bildgebungsprotokollen. Ein erfolgreiches Cyanidexperiment gibt die Gewissheit, dass das NAD(P)H- und FAD-Bildgebungsprotokoll verwendet werden kann, um den Stoffwechsel unbekannter Gruppen oder Störungen zu beurteilen.
Protocol
Representative Results
Discussion
Die Autofluoreszenzintensität und die lebenslange Bildgebung wurden häufig zur Beurteilung des Stoffwechsels in Zellen eingesetzt21,55. FLIM ist hochauflösend und löst daher einzelne Zellen auf, was für Krebsstudien wichtig ist, da die zelluläre Heterogenität zur Tumoraggression und Arzneimittelresistenz beiträgt7,39,41,44,45,46,58.</…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Zu den Finanzierungsquellen gehören das Cancer Prevention and Research Institute of Texas (CPRIT RP200668) und die Texas A & M University. Abbildung 1 wurde mit BioRender.com erstellt.
Materials
| 2-deoxy-d-glucose (2-DG) | Sigma | AC111980000; AC111980010; AC111980050; AC111980250 | |
| Antibiotic Antimicrobial (pen-strep) | Gibco | 15240096 | |
| Cell Samples | American Type Culture Collection | N/A | MCF-7 cancer line |
| CellProfiler | Broad Institute | N/A | Image analysis software |
| Conical Tube | VWR | 89039-664 | 15 mL conical tube |
| DMEM | ThermoFisher | 11965092 | Culture media |
| FAD dichroic mirror | Semrock | FF495-Di03-25×36 | 495 nm |
| FAD emission filter | Semrock | FF01-550/88-25 | 550/88 nm |
| FAD excitation filter | Semrock | FF01-458/64-25 | 458/64 nm |
| FBS | ThermoFisher | 16000036 | |
| Fluorescence Lifetime Microscope | 3i | N/A | |
| Glass bottom dish | MatTek Corp | P35G-1.0-14-C | |
| Multiphoton Laser | Coherent | N/A | 2P Coherent Laser, Tunable 680 nm-1080 nm |
| NAD(P)H dichroic mirror | Semrock | FF409-Di03-25×36 | 409 nm |
| NAD(P)H emission filter | Semrock | FF02-447/60-25 | 447/60 nm |
| NAD(P)H excitation filter | Semrock | FF01-357/44-25 | 357/44 nm |
| PBS | ThermoFisher | 70011044 | |
| Potassium Cyanide | Sigma-Aldrich | 380970 | |
| SlideBooks 6 | 3i | N/A | Image acquisition software |
| SPCImage | Becker & Hickl GmbH | N/A | Fluorescence lifetime analysis software |
| Stage Top Incubator | okoLab | N/A | |
| Trypsin | Biosciences | 786-262 | |
| Urea | Sigma-Aldrich | U5128 | |
| YG beads | Polysciences | 19096-2 | Yg microspheres (20.0 µm) |
Riferimenti
- Heikal, A. A. Intracellular coenzymes as natural biomarkers for metabolic activities and mitochondrial anomalies. Biomarkers in Medicine. 4 (2), 241-263 (2010).
- Georgakoudi, I., Quinn, K. P. Optical imaging using endogenous contrast to assess metabolic state. Annual Review of Biomedical Engineering. 14, 351-367 (2012).
- Zheng, J. Energy metabolism of cancer: Glycolysis versus oxidative phosphorylation (Review). Oncology Letters. 4 (6), 1151-1157 (2012).
- Potter, M., Newport, E., Morten, K. J. The Warburg effect: 80 years on. Biochemical Society Transactions. 44 (5), 1499-1505 (2016).
- Zhao, Y., Butler, E. B., Tan, M. Targeting cellular metabolism to improve cancer therapeutics. Cell Death and Disease. 4 (3), 532 (2013).
- Patel, S., Ahmed, S. Emerging field of metabolomics: Big promise for cancer biomarker identification and drug discovery. Journal of Pharmaceutical and Biomedical Analysis. 107, 63-74 (2015).
- Walsh, A. J., Cook, R. S., Skala, M. C. Functional optical imaging of primary human tumor organoids: Development of a personalized drug screen. Journal of Nuclear Medicine. 58 (9), 1367-1372 (2017).
- Zaal, E. A., Berkers, C. R. The influence of metabolism on drug response in cancer. Frontiers in Oncology. 8, 500 (2018).
- Little, A. C., et al. High-content fluorescence imaging with the metabolic flux assay reveals insights into mitochondrial properties and functions. Communications Biology. 3 (1), 271 (2020).
- Wang, X., et al. Comparison of magnetic resonance spectroscopy and positron emission tomography in detection of tumor recurrence in posttreatment of glioma: A diagnostic meta-analysis. Asia-Pacific Journal of Clinical Oncology. 11 (2), 97-105 (2015).
- Nabi, H. A., Zubeldia, J. M. Clinical applications of 18F-FDG in oncology. Journal of Nuclear Medicine Technology. 30 (1), 3-9 (2002).
- Kostakoglu, L., Agress, H., Goldsmith, S. J. Clinical role of FDG PET in evaluation of cancer patients. Radiographics. 23 (2), 315-340 (2003).
- Hoh, C. K. Clinical use of FDG PET. Nuclear Medicine and Biology. 34 (7), 737-742 (2007).
- van de Weijer, T., Schrauwen-Hinderling, V. B. Application of magnetic resonance spectroscopy in metabolic research. Biochimica et Biophysica Acta. Molecular Basis of Disease. 1865 (4), 741-748 (2019).
- Huang, S., Heikal, A. A., Webb, W. W. Two-photon fluorescence spectroscopy and microscopy of NAD(P)H and Flavoprotein. Biophysical Journal. 82, 2811-2825 (2002).
- Lagarto, J. L., et al. Characterization of NAD(P)H and FAD autofluorescence signatures in a Langendorff isolated-perfused rat heart model. Biomedical Optics Express. 9 (10), 4961-4978 (2018).
- Lakowicz, J. R. . Principles of fluorescence spectroscopy. , (2013).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proceedings of the National Academy of the Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Nakashima, N., Yoshihara, K., Tanaka, F., Yagi, K. Picosecond fluorescence lifetime of the coenzyme of D-amino acid oxidase. Journal of Biological Chemistry. 255 (11), 5261-5263 (1980).
- Hu, L., Wang, N., Cardona, E., Walsh, A. J. Fluorescence intensity and lifetime redox ratios detect metabolic perturbations in T cells. Biomedical Optics Express. 11 (10), 5674-5688 (2020).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (7), 1-43 (2020).
- Liu, Z., et al. Mapping metabolic changes by noninvasive, multiparametric, high-resolution imaging using endogenous contrast. Science Advances. 4 (3), (2018).
- Georgakoudi, I., Quinn, K. P. Optical imaging using endogenous contrast to assess metabolic state. Annual Review of Biomedical Engineering. 14, 351-367 (2012).
- Varone, A., et al. Endogenous two-photon fluorescence imaging elucidates metabolic changes related to enhanced glycolysis and glutamine consumption in precancerous epithelial tissues. Ricerca sul cancro. 74 (11), 3067-3075 (2014).
- Chance, B., Schoener, B., Oshino, R., Itshak, F., Nakase, Y. Oxidation-reduction ratio studies of mitochondria in freeze-trapped samples. NADH and flavoprotein fluorescence signals. Journal of Biological Chemistry. 254 (11), 4764-4771 (1979).
- Sharick, J. T., et al. Protein-bound NAD(P)H lifetime is sensitive to multiple fates of glucose carbon. Scientific Reports. 8 (1), 5456 (2018).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proceedings of the National Academy of Sciences of the United States of America. 104 (49), 19494-19499 (2007).
- Skala, M. C., et al. In vivo multiphoton fluorescence lifetime imaging of protein-bound and free nicotinamide adenine dinucleotide in normal and precancerous epithelia. Journal of Biomedical Optics. 12 (2), 024014 (2007).
- Uchugonova, A. A., König, K. Two-photon autofluorescence and second-harmonic imaging of adult stem cells. Journal of Biomedical Optics. 13 (5), 054068 (2008).
- Miranda-Lorenzo, I., et al. Intracellular autofluorescence: a biomarker for epithelial cancer stem cells. Nature Methods. 11 (11), 1161-1169 (2014).
- Walsh, A. J., et al. Classification of T-cell activation via autofluorescence lifetime imaging. Nature Biomedical Engineering. 5 (1), 77-88 (2021).
- Heaster, T. M., Humayun, M., Yu, J., Beebe, D. J., Skala, M. C. Autofluorescence imaging of 3D tumor-macrophage microscale cultures resolves spatial and temporal dynamics of macrophage metabolism. Ricerca sul cancro. 80 (23), 5408-5423 (2020).
- Pavillon, N., Hobro, A. J., Akira, S., Smith, N. I. Noninvasive detection of macrophage activation with single-cell resolution through machine learning. Proceedings of the National Academy of Sciences of the United States of America. 115 (12), 2676-2685 (2018).
- Chang, C. H., et al. Posttranscriptional control of T cell effector function by aerobic glycolysis. Cell. 153 (6), 1239-1251 (2013).
- Kaech, S. M., Cui, W. Transcriptional control of effector and memory CD8+ T cell differentiation. Nature Reviews. Immunology. 12 (11), 749-761 (2012).
- Gómez, C. A., Fu, B., Sakadžić, S., Yaseena, M. A. Cerebral metabolism in a mouse model of Alzheimer’s disease characterized by two-photon fluorescence lifetime microscopy of intrinsic NADH. Neurophotonics. 5 (4), 045008 (2018).
- Yaseen, M. A., et al. In vivo imaging of cerebral energy metabolism with two-photon fluorescence lifetime microscopy of NADH. Biomedical Optics Express. 4 (2), 307-321 (2013).
- Bower, A. J., et al. High-speed label-free two-photon fluorescence microscopy of metabolic transients during neuronal activity. Applied Physics Letters. 118 (8), 081104 (2021).
- Walsh, A. J., et al. Quantitative optical imaging of primary tumor organoid metabolism predicts drug response in breast cancer. Ricerca sul cancro. 74 (18), 5184-5194 (2014).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, subtypes, and early-treatment response in breast cancer. Ricerca sul cancro. 73 (20), 6164-6174 (2013).
- Chowdary, M. V. P., et al. Autofluorescence of breast tissues: Evaluation of discriminating algorithms for diagnosis of normal, benign, and malignant conditions. Photomedicine and Laser Surgery. 27 (2), 241-252 (2009).
- Demos, S. G., Bold, R., White, R. D., Ramsamooj, R. Investigation of near-infrared autofluorescence imaging for the detection of breast cancer. IEEE Journal of Selected Topics in Quantum Electronics. 11 (4), 791-798 (2005).
- Heaster, T. M., Humayun, M., Yu, J., Beebe, D. J., Skala, M. C. Autofluorescence imaging of 3D tumor-macrophage microscale cultures resolves spatial and temporal dynamics of macrophage metabolism. Ricerca sul cancro. 80 (23), 5408-5423 (2020).
- Sharick, J. T., et al. Cellular metabolic heterogeneity in vivo is recapitulated in tumor organoids. Neoplasia. 21 (6), 615-626 (2019).
- Shah, A. T., Diggins, K. E., Walsh, A. J., Irish, J. M., Skala, M. C. In vivo autofluorescence imaging of tumor heterogeneity in response to treatment. Neoplasia. 17 (12), 862-870 (2015).
- Walsh, A. J., Skala, M. C. Optical metabolic imaging quantifies heterogeneous cell populations. Biomedical Optics Express. 6 (2), 559-573 (2015).
- Walsh, A. J., Skala, M. C. An automated image processing routine for segmentation of cell cytoplasms in high-resolution autofluorescence images. Multiphoton Microscopy in the Biomedical Sciences XIV. , (2014).
- Skala, M., Ramanujam, N. . Methods in Molecular Biology. 594, 155-162 (2010).
- Stringari, C., et al. Multicolor two-photon imaging of endogenous fluorophores in living tissues by wavelength mixing. Scientific Reports. 7, 3792 (2017).
- SPCImage 2.9: Data analysis software for fluorescence lifetime imaging microscopy. SPCImage Available from: https://biology.uiowa.edu/sites/biology.uiowa.edu/files/SPCIMAGE29.pdf (2007)
- . CellProfiler Available from: https://cellprofiler.org/releases (2007)
- Autofluorescence Imaging. GitHub Available from: https://github.com/walshlab/Autofluorescence-Imaging (2021)
- Ramey, N. A., Park, C. Y., Gehlbach, P. L., Chuck, R. S. Imaging mitochondria in living corneal endothelial cells using autofluorescence microscopy. Photochemistry and Photobiology. 83 (6), 1325-1329 (2007).
- Walsh, A., Cook, R. S., Rexer, B., Arteaga, C. L., Skala, M. C. Optical imaging of metabolism in HER2 overexpressing breast cancer cells. Biomedical Optics Express. 3 (1), 75-85 (2012).
- Kolenc, O. I., Quinn, K. P. Evaluating cell metabolism through autofluorescence imaging of NAD(P)H and FAD. Antioxidants & Redox Signaling. 30, 875-889 (2019).
- Bird, D. K., et al. Metabolic mapping of MCF10A human breast cells via multiphoton fluorescence lifetime imaging of the coenzyme NADH. Ricerca sul cancro. 65, 8766-8773 (2005).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, subtypes, and early-treatment response in breast cancer. Ricerca sul cancro. 73 (20), 6164-6174 (2013).
- Walsh, A. J., Castellanos, J. A., Nagathihalli, N. S., Merchant, N. B., Skala, M. C. Optical imaging of drug-induced metabolism changes in murine and human pancreatic cancer organoids reveals heterogeneous drug response. Pancreas. 45 (6), 863-869 (2016).
- Gubser, P. M., et al. Rapid effector function of memory CD8+ T cells requires an immediate-early glycolytic switch. Nature Immunology. 14 (10), 1064-1072 (2013).
- Papalexi, E., Satija, R. Single-cell RNA sequencing to explore immune cell heterogeneity. Nature Review. Immunology. 18 (1), 35-45 (2018).
- Horan, M. P., Pichaud, N., Ballard, J. W. O. Review: Quantifying mitochondrial dysfunction in complex diseases of aging. The Journals of Gerontology: Series A. 67 (10), 1022-1035 (2012).
- Plitzko, B., Loesgen, S. Measurement of oxygen consumption rate (OCR) and extracellular acidification rate (ECAR) in culture cells for assessment of the energy metabolism. Bio-protocol. 8 (10), 2850 (2018).
- . The bh TCSPC Handbook Available from: https://www.becker-hickl.com/wp-content/uploads/2021/10/SPC-handbook-9ed-05a.pdf (2021)
- Gadella, T. W. J., Mason, W. T. . Fluorescent and Luminescent Probes for Biological Activity. 34, 467-479 (1999).
- Miller, D. R., Jarrett, J. W., Hassan, A. M., Dunna, A. K. Deep tissue iImaging with multiphoton fluorescence microscopy. Current Opinion in Biomedical Engineering. 4, 32-39 (2017).
- Berezin, M. Y., Achilefu, S. Fluorescence lifetime measurements and biological imaging. Chemical Reviews. 110 (5), 2641-2684 (2010).

