Hücresel Metabolizmayı Değerlendirmek için Otofloresan Görüntüleme
Summary
Bu protokol, endojen metabolik koenzimlerin, indirgenmiş nikotinamid adenin (fosfat) dinükleotid (NAD (P) H) ve oksitlenmiş flavin adenin dinükleotidin (FAD) floresan görüntülemesini ve analizini tanımlar. NAD(P)H ve FAD’ın otofloresan görüntülemesi, hücresel metabolizmayı değerlendirmek için etiketsiz, tahribatsız bir yöntem sağlar.
Abstract
Hücresel metabolizma, hücrelerin enerji ürettiği süreçtir ve kanser de dahil olmak üzere birçok hastalık anormal metabolizma ile karakterizedir. İndirgenmiş nikotinamid adenin (fosfat) dinükleotid (NAD(P)H) ve oksitlenmiş flavin adenin dinükleotid (FAD) metabolik reaksiyonların koenzimleridir. NAD (P) H ve FAD otofloresan sergiler ve uyarma ve emisyon dalga boyları ile spektral olarak izole edilebilir. Her iki koenzim, NAD (P) H ve FAD, her biri ayrı bir floresan ömrüne sahip olan serbest veya proteine bağlı bir konfigürasyonda bulunabilir – floroforun uyarılmış durumda kaldığı süre. Floresan ömür boyu görüntüleme (FLIM), hücresel metabolizmanın etiketsiz analizi için NAD (P) H ve FAD’nin floresan yoğunluğunun ve ömürlerinin ölçülmesini sağlar. Floresan yoğunluğu ve ömür boyu mikroskoplar, uygun uyarma ve emisyon dalga boyları seçilerek NAD(P)H ve FAD’yi görüntülemek için optimize edilebilir. Siyanür ile metabolik pertürbasyonlar, hücrelerdeki metabolik değişiklikleri tespit etmek için otofloresan görüntüleme protokollerini doğrular. Bu makalede, hücresel metabolizmayı ölçmek için NAD (P) H ve FAD’nin otofloresan görüntüleme tekniği gösterilecektir.
Introduction
Metabolizma, enerji üretmenin hücresel sürecidir. Hücresel metabolizma, glikoliz, oksidatif fosforilasyon ve glutaminoliz dahil olmak üzere birçok yolu kapsar. Sağlıklı hücreler, bağışıklık hücreleri tarafından sitokinlerin üretimi gibi çoğalma ve işlev için enerji üretmek için bu metabolik yolları kullanır. Metabolik bozukluklar, kanser ve nörodejenerasyon dahil olmak üzere birçok hastalık, değişmiş hücresel metabolizma ile karakterizedir1. Örneğin, bazı kanser hücresi tipleri, nükleik asitlerin, proteinlerin ve lipitlerin sentezi için moleküller üretmek üzere oksijen varlığında bile glikoliz oranlarını yükseltmiştir2,3. Warburg etkisi olarak bilinen bu fenomen, meme kanseri, akciğer kanseri ve glioblastomlar dahil olmak üzere birçok kanser türünün ayırt edici özelliğidir4. Kanser progresyonu ile ilişkili hücresel metabolizmadaki değişiklikler nedeniyle, hücresel metabolizma ilaç yanıtı için vekil bir biyobelirteç olabilir5,6. Ayrıca, hücre heterojenliği bireylerde farklı ilaç yanıtlarına yol açabileceğinden, ilaç etkinliğini hücresel düzeyde anlamak çok önemlidir7,8.
Hücresel metabolizmadaki değişiklikleri tanımlayan ve ölçen teknolojiler, kanser ve ilaç yanıtı çalışmaları için gereklidir. Kimyasal ve protein analizleri, hücrelerin veya dokuların metabolizmasını değerlendirmek için kullanılır, ancak tek hücreli çözünürlük ve mekansal bilgiden yoksundur. Metabolik plaka okuyucu tabanlı testler, numunedeki pH ve oksijen tüketimini zaman içinde ve ardından kimyasallar tarafından metabolik bozulmayı ölçebilir. PH, hücrelerin glikolitik aktivitesi hakkında bir fikir veren hücre dışı asitleşme oranını (ECAR) hesaplamak için kullanılabilir9. 2-[flor-18] floro-D-glukoz pozitron emisyon tomografisi (FDG PET) ve manyetik rezonans spektroskopisi (MRS) dahil olmak üzere tüm vücut görüntüleme yöntemleri, metabolik ölçümler yoluyla tümör nüksünü ve ilaç etkinliğini tanımlamak için klinik olarak kullanılan noninvaziv görüntüleme yöntemleridir10,11,12,13,14.
FDG-PET, RADYO-etiketli bir glikoz analoğu olan FDG’nin doku alımını görüntüler. Tümörler tarafından çevre dokuya göre FDG-PET alımının artması, Warburg etkisinden kaynaklanmaktadır12,13. MRS, 13C ve 31P gibi metabolizma için kullanılan moleküllerin ortak çekirdeklerini görüntüler ve metabolizmanın egzersiz veya yemek yeme gibi uyaranlara yanıt olarak nasıl değiştiği hakkında dinamik bilgiler elde edebilir14. FDG-PET ve MRS klinik olarak kullanılabilse de, bu teknolojiler intratümöral heterojenliği çözmek için uzamsal çözünürlükten yoksundur. Aynı şekilde, oksijen tüketimi ölçümleri büyük miktarda hücre popülasyonu üzerinde yapılır. Otofloresan görüntüleme, bu teknolojilerin mekansal çözünürlük engelinin üstesinden gelir ve hücresel metabolizmayı ölçmek için invaziv olmayan bir yöntem sağlar.
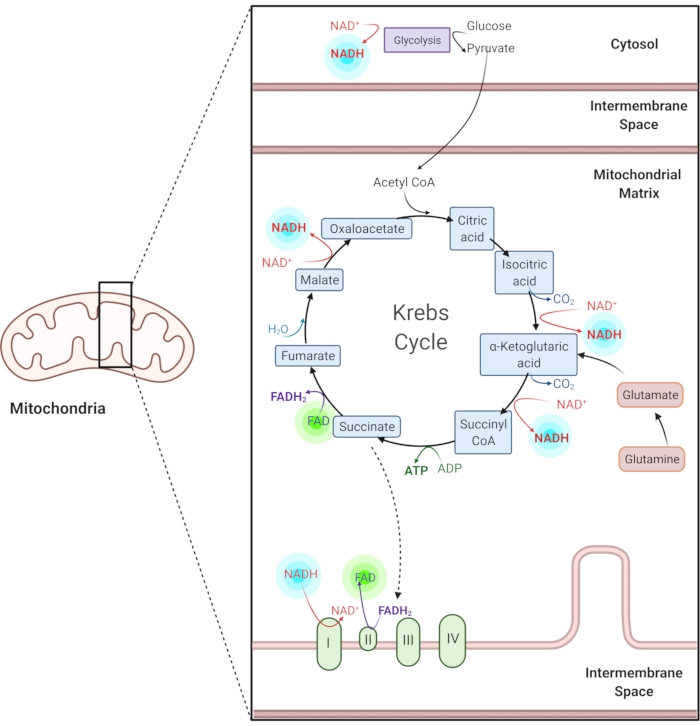
Şekil 1: Ortak metabolik yollarda NADH ve FAD. NADH ve FAD, glikoliz, Krebs döngüsü ve elektron taşıma zincirinde kullanılan koenzimlerdir. Bu moleküllerin otofloresan görüntülemesi hücresel metabolizma hakkında bilgi sağlar. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
İndirgenmiş nikotinamid adenin (fosfat) dinükleotid (NAD (P) H) ve oksitlenmiş flavin adenin dinükleotid (FAD), glikoliz, oksidatif fosforilasyon ve glutaminoliz dahil olmak üzere metabolik reaksiyonların koenzimleridir (Şekil 1). Hem NAD(P)H hem de FAD otofloresandır ve floresan görüntüleme için endojen kontrast sağlar1,15. NADPH, NADH’ye benzer floresan özelliklere sahiptir. Bu nedenle, NAD (P) H genellikle NADH ve NADPH2,16’nın birleşik sinyalini temsil etmek için kullanılır.
Floresan ömür boyu görüntüleme (FLIM), floresan ömrünü veya bir floroforun uyarılmış durumda olduğu süreyi ölçer. Floresan ömürleri, floroforların mikro ortamına duyarlıdır ve hücresel metabolizma hakkında bilgi sağlar17. NAD (P) H ve FAD, her biri farklı bir ömre sahip olan proteine bağlı veya serbest konformasyonlardaki hücrelerde bulunabilir. Serbest NAD (P) H, proteine bağlı NAD (P) H’den daha kısa bir ömre sahiptir; tersine, serbest FAD, bağlı FAD18,19’dan daha uzun bir ömre sahiptir. Yaşam süreleri ve ömür boyu bileşen ağırlıkları, floresan ömür boyu bozunma verilerinden Ek. (1)20 aracılığıyla ölçülebilir:
I(t) = α 1e-t/τ1 + α 2e-t/τ2 + C (1)
Eq (1), zamanın bir fonksiyonu olarak normalleştirilmiş floresan yoğunluğunu temsil eder. Bu denklemdeki α 1 ve α 2, sırasıyla kısa ve uzun ömürlerin orantılı bileşenlerini temsil eder (α 1+ α 2=1), τ1 ve τ2 sırasıyla kısa ve uzun ömürleri temsil eder ve C, arka plan ışığını7,20 oluşturur. Burada τm olarak temsil edilen genlik ağırlıklı yaşam süresi, Ek. (2) kullanılarak hesaplanır.
τm= α 1τ1+ α 2τ2 (2)
Ortalama bir ömür, floroforun yoğunluk bozunumu üzerinde ortalama “t” ile hesaplanabilir, bu da iki üstel bir bozunma için Eq. (3) 17,21 ile gösterilir.
τ*m= (α 1τ12+ α 2τ22)/ (α 1τ1+ α 2τ2) (3)
Bir floresan yoğunluğu görüntüsü, floresan ömür boyu bozunumu entegre edilerek ömür boyu görüntüden hesaplanabilir. Otofloresan görüntüleme, canlı hücrelerin metabolizmasını hücre altı çözünürlükte karakterize etmek için kullanılabilecek tahribatsız ve etiketsiz bir yöntemdir. Optik redoks oranı, hücrenin kimyasal redoks durumunun optik analog metriğini sağlar ve NAD (P) H ve FAD yoğunluklarının oranı olarak hesaplanır. Optik redoks oranının hesaplanması için formül standartlaştırılmamış olsa da22,23,24,25, burada NAD (P) H ve FAD’nin birleşik yoğunlukları üzerindeki FAD yoğunluğu olarak tanımlanmaktadır. Bu tanım kullanılır, çünkü paydadaki toplam yoğunluk metriği 0 ile 1 arasında normalleştirir ve siyanür inhibisyonunun beklenen sonucu redoks oranında bir azalmadır. Serbest NAD(P)H ve FAD’ın floresan ömürleri, pH, sıcaklık, oksijene yakınlık ve ozmolarite dahil olmak üzere metabolik çözücü mikro ortamındaki değişiklikler hakkında fikir verir17.
NAD(P)H ve FAD’nin bağlı fraksiyonlarının floresan ömründeki değişiklikler, metabolik yol kullanımını ve substrata özgü metabolizmayı gösterebilir26. Komponent ağırlıkları, koenzimlerin serbest ila bağlı fraksiyonundaki değişiklikler için yorumlanabilir18,19. Toplamda, bu kantitatif otofloresan yaşam boyu metrikleri hücresel metabolizmanın analizine izin verir ve otofloresan görüntüleme normal dokulardan neoplazmları tanımlamak27,28, kök hücreleri karakterize etmek29,30, bağışıklık hücresi fonksiyonunu değerlendirmek31,32,33,34,35, nörolojik aktiviteyi ölçmek için kullanılmıştır36, 37,38 ve meme kanseri ve baş boyun kanseri gibi kanser türlerinde ilaç etkinliğini anlamak21,39,40,41,42. Yüksek çözünürlüklü otofloresan görüntüleme, tek hücreli analiz ve popülasyon içi heterojenitenin nicelleştirilmesi için görüntü segmentasyonu ile birleştirilebilir43,44,45,46,47.
NAD (P) H ve FAD, yoğunluk veya ömür boyu görüntüleme için yapılandırılmış tek fotonlu veya çok fotonlu floresan mikroskoplarda görüntülenebilir. Tek fotonlu mikroskoplar için, NAD (P) H ve FAD, bu dalga boylarındaki ortak lazer kaynakları nedeniyle, sırasıyla 375-405 nm ve 488 nm dalga boylarında tipik olarak uyarılır48. İki fotonlu floresan uyarımında, NAD (P) H ve FAD sırasıyla yaklaşık 700 ila 750 nm ve 700 ila 900 nm dalga boylarında uyaracaktır, 15,49. Floroforlar uyarıldığında, NAD (P) H ve FAD sırasıyla ~ 410 nm ila ~ 490 nm ve ~ 510 nm ila ~ 640 nm arasındaki dalga boylarında fotonlar yayar15. NAD (P) H ve FAD maksimuma emisyon dalga boyları sırasıyla yaklaşık 450 nm ve 535 nm’dir48.
Farklı uyarma ve emisyon dalga boyları nedeniyle, iki metabolik koenzimin floresansı spektral olarak izole edilebilir. NAD(P)H ve FAD’ın spektral özelliklerinin anlaşılması, otofloresan görüntüleme protokollerinin tasarımı ve optimizasyonu için gereklidir. Siyanür bir elektron taşıma zinciri (ETC) kompleks IV inhibitörüdür. Siyanürün hücresel metabolizma üzerindeki etkileri ve hücrelerdeki NAD(P)H ve FAD’ın otofloresan yoğunlukları ve ömürleri iyi karakterize edilmiştir27,40. Bu nedenle, bir siyanür pertürbasyon deneyi, NAD (P) H ve FAD görüntüleme protokollerini doğrulamak için etkili bir araçtır. Başarılı bir siyanür deneyi, NAD (P) H ve FAD görüntüleme protokolünün bilinmeyen grupların veya bozulmaların metabolizmasını değerlendirmek için kullanılabileceğine dair güven sağlar.
Protocol
Representative Results
Discussion
Otofloresan yoğunluğu ve ömür boyu görüntüleme, hücrelerdeki metabolizmayı değerlendirmek için yaygın olarak kullanılmaktadır21,55. FLIM yüksek çözünürlüktedir ve bu nedenle kanser çalışmaları için önemli olan tek hücreleri çözer, çünkü hücresel heterojenlik tümör saldırganlığına ve ilaç direncine katkıda bulunur7,39,41,44,45,46,58.</su…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Finansman kaynakları arasında Teksas Kanser Önleme ve Araştırma Enstitüsü (CPRIT RP200668) ve Texas A & M Üniversitesi bulunmaktadır. Şekil 1, BioRender.com ile oluşturulmuştur.
Materials
| 2-deoxy-d-glucose (2-DG) | Sigma | AC111980000; AC111980010; AC111980050; AC111980250 | |
| Antibiotic Antimicrobial (pen-strep) | Gibco | 15240096 | |
| Cell Samples | American Type Culture Collection | N/A | MCF-7 cancer line |
| CellProfiler | Broad Institute | N/A | Image analysis software |
| Conical Tube | VWR | 89039-664 | 15 mL conical tube |
| DMEM | ThermoFisher | 11965092 | Culture media |
| FAD dichroic mirror | Semrock | FF495-Di03-25×36 | 495 nm |
| FAD emission filter | Semrock | FF01-550/88-25 | 550/88 nm |
| FAD excitation filter | Semrock | FF01-458/64-25 | 458/64 nm |
| FBS | ThermoFisher | 16000036 | |
| Fluorescence Lifetime Microscope | 3i | N/A | |
| Glass bottom dish | MatTek Corp | P35G-1.0-14-C | |
| Multiphoton Laser | Coherent | N/A | 2P Coherent Laser, Tunable 680 nm-1080 nm |
| NAD(P)H dichroic mirror | Semrock | FF409-Di03-25×36 | 409 nm |
| NAD(P)H emission filter | Semrock | FF02-447/60-25 | 447/60 nm |
| NAD(P)H excitation filter | Semrock | FF01-357/44-25 | 357/44 nm |
| PBS | ThermoFisher | 70011044 | |
| Potassium Cyanide | Sigma-Aldrich | 380970 | |
| SlideBooks 6 | 3i | N/A | Image acquisition software |
| SPCImage | Becker & Hickl GmbH | N/A | Fluorescence lifetime analysis software |
| Stage Top Incubator | okoLab | N/A | |
| Trypsin | Biosciences | 786-262 | |
| Urea | Sigma-Aldrich | U5128 | |
| YG beads | Polysciences | 19096-2 | Yg microspheres (20.0 µm) |
Riferimenti
- Heikal, A. A. Intracellular coenzymes as natural biomarkers for metabolic activities and mitochondrial anomalies. Biomarkers in Medicine. 4 (2), 241-263 (2010).
- Georgakoudi, I., Quinn, K. P. Optical imaging using endogenous contrast to assess metabolic state. Annual Review of Biomedical Engineering. 14, 351-367 (2012).
- Zheng, J. Energy metabolism of cancer: Glycolysis versus oxidative phosphorylation (Review). Oncology Letters. 4 (6), 1151-1157 (2012).
- Potter, M., Newport, E., Morten, K. J. The Warburg effect: 80 years on. Biochemical Society Transactions. 44 (5), 1499-1505 (2016).
- Zhao, Y., Butler, E. B., Tan, M. Targeting cellular metabolism to improve cancer therapeutics. Cell Death and Disease. 4 (3), 532 (2013).
- Patel, S., Ahmed, S. Emerging field of metabolomics: Big promise for cancer biomarker identification and drug discovery. Journal of Pharmaceutical and Biomedical Analysis. 107, 63-74 (2015).
- Walsh, A. J., Cook, R. S., Skala, M. C. Functional optical imaging of primary human tumor organoids: Development of a personalized drug screen. Journal of Nuclear Medicine. 58 (9), 1367-1372 (2017).
- Zaal, E. A., Berkers, C. R. The influence of metabolism on drug response in cancer. Frontiers in Oncology. 8, 500 (2018).
- Little, A. C., et al. High-content fluorescence imaging with the metabolic flux assay reveals insights into mitochondrial properties and functions. Communications Biology. 3 (1), 271 (2020).
- Wang, X., et al. Comparison of magnetic resonance spectroscopy and positron emission tomography in detection of tumor recurrence in posttreatment of glioma: A diagnostic meta-analysis. Asia-Pacific Journal of Clinical Oncology. 11 (2), 97-105 (2015).
- Nabi, H. A., Zubeldia, J. M. Clinical applications of 18F-FDG in oncology. Journal of Nuclear Medicine Technology. 30 (1), 3-9 (2002).
- Kostakoglu, L., Agress, H., Goldsmith, S. J. Clinical role of FDG PET in evaluation of cancer patients. Radiographics. 23 (2), 315-340 (2003).
- Hoh, C. K. Clinical use of FDG PET. Nuclear Medicine and Biology. 34 (7), 737-742 (2007).
- van de Weijer, T., Schrauwen-Hinderling, V. B. Application of magnetic resonance spectroscopy in metabolic research. Biochimica et Biophysica Acta. Molecular Basis of Disease. 1865 (4), 741-748 (2019).
- Huang, S., Heikal, A. A., Webb, W. W. Two-photon fluorescence spectroscopy and microscopy of NAD(P)H and Flavoprotein. Biophysical Journal. 82, 2811-2825 (2002).
- Lagarto, J. L., et al. Characterization of NAD(P)H and FAD autofluorescence signatures in a Langendorff isolated-perfused rat heart model. Biomedical Optics Express. 9 (10), 4961-4978 (2018).
- Lakowicz, J. R. . Principles of fluorescence spectroscopy. , (2013).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proceedings of the National Academy of the Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Nakashima, N., Yoshihara, K., Tanaka, F., Yagi, K. Picosecond fluorescence lifetime of the coenzyme of D-amino acid oxidase. Journal of Biological Chemistry. 255 (11), 5261-5263 (1980).
- Hu, L., Wang, N., Cardona, E., Walsh, A. J. Fluorescence intensity and lifetime redox ratios detect metabolic perturbations in T cells. Biomedical Optics Express. 11 (10), 5674-5688 (2020).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (7), 1-43 (2020).
- Liu, Z., et al. Mapping metabolic changes by noninvasive, multiparametric, high-resolution imaging using endogenous contrast. Science Advances. 4 (3), (2018).
- Georgakoudi, I., Quinn, K. P. Optical imaging using endogenous contrast to assess metabolic state. Annual Review of Biomedical Engineering. 14, 351-367 (2012).
- Varone, A., et al. Endogenous two-photon fluorescence imaging elucidates metabolic changes related to enhanced glycolysis and glutamine consumption in precancerous epithelial tissues. Ricerca sul cancro. 74 (11), 3067-3075 (2014).
- Chance, B., Schoener, B., Oshino, R., Itshak, F., Nakase, Y. Oxidation-reduction ratio studies of mitochondria in freeze-trapped samples. NADH and flavoprotein fluorescence signals. Journal of Biological Chemistry. 254 (11), 4764-4771 (1979).
- Sharick, J. T., et al. Protein-bound NAD(P)H lifetime is sensitive to multiple fates of glucose carbon. Scientific Reports. 8 (1), 5456 (2018).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proceedings of the National Academy of Sciences of the United States of America. 104 (49), 19494-19499 (2007).
- Skala, M. C., et al. In vivo multiphoton fluorescence lifetime imaging of protein-bound and free nicotinamide adenine dinucleotide in normal and precancerous epithelia. Journal of Biomedical Optics. 12 (2), 024014 (2007).
- Uchugonova, A. A., König, K. Two-photon autofluorescence and second-harmonic imaging of adult stem cells. Journal of Biomedical Optics. 13 (5), 054068 (2008).
- Miranda-Lorenzo, I., et al. Intracellular autofluorescence: a biomarker for epithelial cancer stem cells. Nature Methods. 11 (11), 1161-1169 (2014).
- Walsh, A. J., et al. Classification of T-cell activation via autofluorescence lifetime imaging. Nature Biomedical Engineering. 5 (1), 77-88 (2021).
- Heaster, T. M., Humayun, M., Yu, J., Beebe, D. J., Skala, M. C. Autofluorescence imaging of 3D tumor-macrophage microscale cultures resolves spatial and temporal dynamics of macrophage metabolism. Ricerca sul cancro. 80 (23), 5408-5423 (2020).
- Pavillon, N., Hobro, A. J., Akira, S., Smith, N. I. Noninvasive detection of macrophage activation with single-cell resolution through machine learning. Proceedings of the National Academy of Sciences of the United States of America. 115 (12), 2676-2685 (2018).
- Chang, C. H., et al. Posttranscriptional control of T cell effector function by aerobic glycolysis. Cell. 153 (6), 1239-1251 (2013).
- Kaech, S. M., Cui, W. Transcriptional control of effector and memory CD8+ T cell differentiation. Nature Reviews. Immunology. 12 (11), 749-761 (2012).
- Gómez, C. A., Fu, B., Sakadžić, S., Yaseena, M. A. Cerebral metabolism in a mouse model of Alzheimer’s disease characterized by two-photon fluorescence lifetime microscopy of intrinsic NADH. Neurophotonics. 5 (4), 045008 (2018).
- Yaseen, M. A., et al. In vivo imaging of cerebral energy metabolism with two-photon fluorescence lifetime microscopy of NADH. Biomedical Optics Express. 4 (2), 307-321 (2013).
- Bower, A. J., et al. High-speed label-free two-photon fluorescence microscopy of metabolic transients during neuronal activity. Applied Physics Letters. 118 (8), 081104 (2021).
- Walsh, A. J., et al. Quantitative optical imaging of primary tumor organoid metabolism predicts drug response in breast cancer. Ricerca sul cancro. 74 (18), 5184-5194 (2014).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, subtypes, and early-treatment response in breast cancer. Ricerca sul cancro. 73 (20), 6164-6174 (2013).
- Chowdary, M. V. P., et al. Autofluorescence of breast tissues: Evaluation of discriminating algorithms for diagnosis of normal, benign, and malignant conditions. Photomedicine and Laser Surgery. 27 (2), 241-252 (2009).
- Demos, S. G., Bold, R., White, R. D., Ramsamooj, R. Investigation of near-infrared autofluorescence imaging for the detection of breast cancer. IEEE Journal of Selected Topics in Quantum Electronics. 11 (4), 791-798 (2005).
- Heaster, T. M., Humayun, M., Yu, J., Beebe, D. J., Skala, M. C. Autofluorescence imaging of 3D tumor-macrophage microscale cultures resolves spatial and temporal dynamics of macrophage metabolism. Ricerca sul cancro. 80 (23), 5408-5423 (2020).
- Sharick, J. T., et al. Cellular metabolic heterogeneity in vivo is recapitulated in tumor organoids. Neoplasia. 21 (6), 615-626 (2019).
- Shah, A. T., Diggins, K. E., Walsh, A. J., Irish, J. M., Skala, M. C. In vivo autofluorescence imaging of tumor heterogeneity in response to treatment. Neoplasia. 17 (12), 862-870 (2015).
- Walsh, A. J., Skala, M. C. Optical metabolic imaging quantifies heterogeneous cell populations. Biomedical Optics Express. 6 (2), 559-573 (2015).
- Walsh, A. J., Skala, M. C. An automated image processing routine for segmentation of cell cytoplasms in high-resolution autofluorescence images. Multiphoton Microscopy in the Biomedical Sciences XIV. , (2014).
- Skala, M., Ramanujam, N. . Methods in Molecular Biology. 594, 155-162 (2010).
- Stringari, C., et al. Multicolor two-photon imaging of endogenous fluorophores in living tissues by wavelength mixing. Scientific Reports. 7, 3792 (2017).
- SPCImage 2.9: Data analysis software for fluorescence lifetime imaging microscopy. SPCImage Available from: https://biology.uiowa.edu/sites/biology.uiowa.edu/files/SPCIMAGE29.pdf (2007)
- . CellProfiler Available from: https://cellprofiler.org/releases (2007)
- Autofluorescence Imaging. GitHub Available from: https://github.com/walshlab/Autofluorescence-Imaging (2021)
- Ramey, N. A., Park, C. Y., Gehlbach, P. L., Chuck, R. S. Imaging mitochondria in living corneal endothelial cells using autofluorescence microscopy. Photochemistry and Photobiology. 83 (6), 1325-1329 (2007).
- Walsh, A., Cook, R. S., Rexer, B., Arteaga, C. L., Skala, M. C. Optical imaging of metabolism in HER2 overexpressing breast cancer cells. Biomedical Optics Express. 3 (1), 75-85 (2012).
- Kolenc, O. I., Quinn, K. P. Evaluating cell metabolism through autofluorescence imaging of NAD(P)H and FAD. Antioxidants & Redox Signaling. 30, 875-889 (2019).
- Bird, D. K., et al. Metabolic mapping of MCF10A human breast cells via multiphoton fluorescence lifetime imaging of the coenzyme NADH. Ricerca sul cancro. 65, 8766-8773 (2005).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, subtypes, and early-treatment response in breast cancer. Ricerca sul cancro. 73 (20), 6164-6174 (2013).
- Walsh, A. J., Castellanos, J. A., Nagathihalli, N. S., Merchant, N. B., Skala, M. C. Optical imaging of drug-induced metabolism changes in murine and human pancreatic cancer organoids reveals heterogeneous drug response. Pancreas. 45 (6), 863-869 (2016).
- Gubser, P. M., et al. Rapid effector function of memory CD8+ T cells requires an immediate-early glycolytic switch. Nature Immunology. 14 (10), 1064-1072 (2013).
- Papalexi, E., Satija, R. Single-cell RNA sequencing to explore immune cell heterogeneity. Nature Review. Immunology. 18 (1), 35-45 (2018).
- Horan, M. P., Pichaud, N., Ballard, J. W. O. Review: Quantifying mitochondrial dysfunction in complex diseases of aging. The Journals of Gerontology: Series A. 67 (10), 1022-1035 (2012).
- Plitzko, B., Loesgen, S. Measurement of oxygen consumption rate (OCR) and extracellular acidification rate (ECAR) in culture cells for assessment of the energy metabolism. Bio-protocol. 8 (10), 2850 (2018).
- . The bh TCSPC Handbook Available from: https://www.becker-hickl.com/wp-content/uploads/2021/10/SPC-handbook-9ed-05a.pdf (2021)
- Gadella, T. W. J., Mason, W. T. . Fluorescent and Luminescent Probes for Biological Activity. 34, 467-479 (1999).
- Miller, D. R., Jarrett, J. W., Hassan, A. M., Dunna, A. K. Deep tissue iImaging with multiphoton fluorescence microscopy. Current Opinion in Biomedical Engineering. 4, 32-39 (2017).
- Berezin, M. Y., Achilefu, S. Fluorescence lifetime measurements and biological imaging. Chemical Reviews. 110 (5), 2641-2684 (2010).

