인간 췌장 조직에서 N-글리코펩티드와 포스포펩티드의 동시 분석을 위한 스핀-팁 농축 전략
Summary
번역 후 변형(PTM)은 단백질 구조와 기능을 변화시킵니다. 여러 PTM 유형을 동시에 농축하는 방법은 분석에서 적용 범위를 최대화할 수 있습니다. 우리는 이중 작용성 Ti (IV) 고정화 금속 친화성 크로마토그래피를 사용한 프로토콜을 제시하고 췌장 조직에서 단백질 N- 글리코실화 및 인산화의 동시 농축 및 분석을 위해 질량 분광법을 제시합니다.
Abstract
질량 분광법은 번역 후 변형 (PTM)에 대한 깊은 범위를 제공 할 수 있지만, 복잡한 생물학적 매트릭스로부터의 이러한 변형의 농축은 종종 비 변형 된 분석물에 비해 낮은 화학량론으로 인해 필요합니다. 상향식 프로테오믹스 워크플로우에서 펩티드에 대한 PTM의 대부분의 농축 워크플로우는 생성된 펩티드가 분석되기 전에 단백질이 효소적으로 소화되고, 오직 한 가지 유형의 변형만을 풍부하게 한다. 그러나 PTM의 전체 보완은 생물학적 기능을 유도하고 단일 유형의 PTM의 농축은 PTM의 이러한 누화를 놓칠 수 있습니다. PTM 누화는 단백질 글리코실화와 인산화 사이에서 관찰되었으며, 인간 단백질에서 가장 일반적인 두 가지 PTM과 질량 분광법 워크 플로우를 사용하여 가장 많이 연구 된 두 가지 PTM입니다. 본원에 기재된 동시 농축 전략을 사용하여, 두 PTM 모두 사후 인간 췌장 조직, 복잡한 생물학적 매트릭스로부터 농축된다. 이중 작용성 Ti(IV) 고정화 금속 친화성 크로마토그래피는 편리한 스핀 팁 기반 방법으로 여러 분획에서 다양한 형태의 글리코실화 및 인산화를 동시에 분리하는 데 사용되므로 잠재적인 PTM 누화 상호작용의 다운스트림 분석이 가능합니다. 글리코 및 포스포펩타이드에 대한 이러한 농축 워크플로우는 다양한 샘플 유형에 적용되어 여러 PTM의 심층 프로파일링을 달성하고 향후 연구를 위한 잠재적 표적 분자를 식별할 수 있습니다.
Introduction
단백질 번역 후 변형 (PTM)은 단백질 구조와 결과적으로 기능 및 하류 생물학적 과정을 조절하는 데 중요한 역할을합니다. 인간 프로테옴의 다양성은 다양한 PTM에 의해 제공되는 조합 변동성으로 인해 기하 급수적으로 증가합니다. 게놈에 의해 예측 된 바와 같이 정준 서열로부터의 단백질의 다른 변이체는 프로테오폼으로 알려져 있으며, 많은 프로테오형은 PTMs1에서 발생합니다. 건강과 질병의 프로테오폼 다양성을 연구하는 것은 최근 몇 년 동안 큰 관심의 연구 분야가되었습니다 2,3.
더 깊은 깊이를 가진 프로테오폼 및 보다 구체적으로 PTM에 대한 연구는 질량 분광분석법(MS) 기반 프로테오믹스 방법의 개발을 통해 더욱 용이해졌다. MS를 사용하여, 분석물은 이온화되고, 단편화되고, 단편화되고, 단편의 m/z 에 기초하여 식별된다. 농축 방법은 종종 단백질의 비 변형 형태에 비해 PTM의 상대적 풍부도가 낮기 때문에 필요합니다. 하향식 분석이라고 불리는 손상되지 않은 단백질과 PTM의 분석이 더욱 일상화되었지만, 단백질의 효소 소화와 상향식 분석에서 구성 요소 펩티드의 분석은 여전히 PTM 분석에 가장 널리 사용되는 경로입니다. 가장 널리 연구된 두 개의 PTM과 생체내에서 가장 흔한 두 개의 PTM은 글리코실화와 인산화4이다. 이 두 PTM은 세포 신호 전달 및 인식에 중요한 역할을하므로 질병 연구에서 특성화하는 중요한 변형입니다.
다양한 PTM의 화학적 특성은 종종 분석 전에 단백질 및 펩티드 수준에서 이러한 PTM을 농축하는 경로를 제공합니다. 글리코실화는 각 단당류에 히드록실기가 풍부하기 때문에 친수성 PTM이다. 이러한 특성은 친수성 상호작용 크로마토그래피(HILIC)에서 글리코펩티드를 풍부하게 하는데 사용될 수 있으며, 이는 소수성 비변형된 펩티드5로부터 더 친수성 글리코펩티드를 분리할 수 있다. 인산화는 산성 pH를 제외하고 음전하를 띤 포스페이트 모이어티를 추가한다. 이러한 전하로 인해 티타늄을 포함한 다양한 금속 양이온을 사용하여 포스포 펩타이드를 유인하고 결합 할 수 있으며 비 인산화 된 종은 씻겨 나갈 수 있습니다. 이것은 고정화 된 금속 친화성 크로마토그래피 (IMAC)의 원리입니다. 글리코실화 및 인산화를 위한 이들 및 다른 농축 전략에 대한 추가 논의는 최근 리뷰 6,7에서 찾을 수 있다.
비교적 많은 양의 출발 펩티드 물질 (0.5 mg 이상)은 펩티드 상의 PTMs의 낮은 화학량론으로 인해 농축 프로토콜에 종종 필요하다. 종양 코어 생검 또는 뇌척수액 분석과 같이 이러한 양의 샘플을 쉽게 얻을 수 없는 시나리오에서는 최대 생체 분자 정보를 생성하는 facile 워크플로우를 사용하는 것이 좋습니다. 우리 연구실과 다른 사람들이 개발 한 최근의 전략은 동일한 PTM 농축 워크 플로 8,9,10,11,12를 사용하여 글리코실화 및 인산화의 동시 및 병렬 분석을 강조했습니다. 이 두 PTM의 화학적 특성은 다를 수 있지만 이러한 PTM은 혁신적인 분리 기술 및 사용 된 재료로 인해 여러 단계로 분석 될 수 있습니다. 예를 들어, 정전기 반발-친수성 상호작용 크로마토그래피(ERLIC)는 분석물과 이동상 사이의 친수성 상호작용에 기초한 분리를 분석물과 고정상 물질 사이의 전하-전하 상호작용(13,14,15,16)에 기초하여 분리를 오버레이한다. 산성 pH에서, 고정상으로의 인산화된 펩티드의 인력은 그들의 보유 및 비변형된 펩티드로부터의 분리를 향상시킬 수 있다. 친수성 미소 구체 상에 고정화된 Ti(IV)로 구성된 물질은 포스포펩티드와 중성, 산성 및 만노스-6-인산화된 글리코펩티드17,18을 분리하기 위해 HILIC 및 IMAC 기반 용출에 사용될 수 있다. 이 전략은 이중 기능 Ti(IV)-IMAC로 알려져 있습니다. 단일 워크플로우에서 여러 PTM을 보강하기 위해 이러한 전략을 사용하면 잠재적인 PTM 누화 상호 작용에 대한 분석에 더 쉽게 액세스할 수 있습니다. 추가적으로, 총 샘플 양 및 시간 요건은 병렬로 수행될 때 종래의 농축 방법(즉, 개별 샘플 분취량 상의 HILIC 및 IMAC)보다 작다.
단백질 글리코실화 및 인산화의 동시 분석을 위한 이중 기능적 Ti(IV)-IMAC 전략을 입증하기 위해, 우리는 사후 인간 췌장 조직을 분석하기 위해 이를 적용했습니다. 췌장은 인슐린과 글루카곤을 포함한 소화 효소와 조절 호르몬을 모두 생산합니다. 췌장 기능은 췌장 질환에서 손상됩니다. 당뇨병에서는 혈당 조절이 영향을 받아 혈액 내 포도당 수치가 높아집니다. 췌장염에서 염증은 장기의 자동 소화로 인해 발생합니다3. 글리코실화 및 인산화를 포함한 PTM 프로파일의 변화는 종종 그렇듯이 다른 질병에서 발생할 수 있습니다.
여기에서는 췌장 조직에서 추출한 단백질로부터 유래된 N-글리코펩티드 및 포스포펩티드에 대한 이중 기능성 Ti(IV)-IMAC 전략에 기초한 스핀팁 기반 동시 농축 방법에 대한 프로토콜을 설명한다. 이 프로토콜은 도 1에서 볼 수 있는 바와 같이 단백질 추출 및 소화, 농축, MS 데이터 수집 및 데이터 처리를 포함한다. 이 연구의 대표적인 데이터는 식별자 PXD033065를 가진 ProteomeXchange 컨소시엄을 통해 구할 수 있습니다.
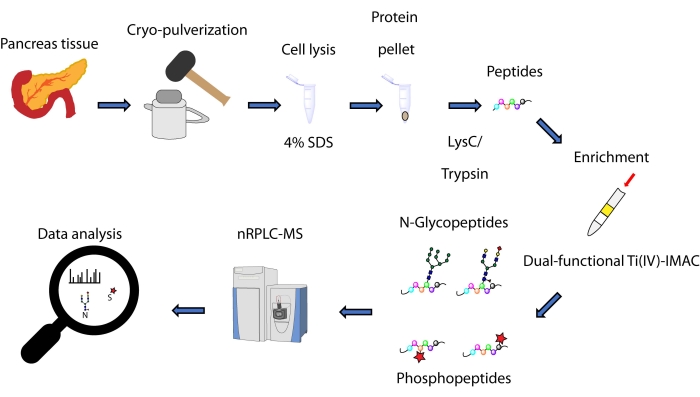
그림 1: 인간 췌장 조직에서 N-글리코펩티드와 포스포펩티드를 동시에 분석하기 위한 워크플로우. 조직은 먼저 세제 소듐 도데실 설페이트(SDS)를 사용하여 단백질 추출 전에 미세한 분말로 분쇄된다. 그런 다음 단백질은 효소 소화를 받는다. 생성된 펩티드는 이중 작용성 Ti(IV)-IMAC를 사용하여 농축하기 전에 분취된다. 원시 데이터는 나노 스케일 역상 액체 크로마토그래피 – 질량 분광법 (nRPLC-MS)을 사용하여 수집되며 데이터베이스 검색 소프트웨어를 사용하여 분석됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
이 프로토콜은 PTM 분석의 접근성을 높이고 동일한 워크플로우에서 여러 PTM을 보다 광범위하게 분석할 수 있도록 하기 위한 것입니다. 이 프로토콜은 세포 및 생체유체를 포함한 다른 복잡한 생물학적 매트릭스에 적용될 수 있다.
Protocol
Representative Results
Discussion
이중 기능적 Ti(IV)-IMAC 전략은 단일 샘플 준비 워크플로우에서 동일한 샘플의 N-글리코펩티드와 포스포펩티드를 동시에 분석하는 데 유용합니다. ERLIC 기반 방법은 또한 PTM의 동시 농축을 수행하는 것으로 나타났습니다. 두 전략 모두 이전에 PTM 분석14,18에서 심층적 인 커버리지를 위해 사용되었습니다. 스핀 팁을 사용하여 샘플 인큐베이션 시간을 줄이?…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
이 연구는 NIH (R01DK071801, RF1AG052324, P01CA250972 및 R21AG065728) 및 청소년 당뇨병 연구 재단 (1-PNF-2016-250-S-B 및 SRA-2016-168-S-B)의 보조금으로 부분적으로 지원되었습니다. 여기에 제시된 데이터는 위스콘신 대학교 임상 및 번역 연구 연구소를 통해 NIH / NCATS UL1TR002373 어워드의 지원을 통해 부분적으로 획득되었습니다. Orbitrap 장비는 NIH 공유 기기 보조금 (NIH-NCRR S10RR029531)과 위스콘신-매디슨 대학의 연구 및 대학원 교육 부총장 사무실의 지원을 통해 구입되었습니다. 우리는 또한 연구를 위해 인간의 췌장을 제공 한 위스콘신 대학 장기 및 조직 기증기구의 관대 한 지원과 Dan Tremmel, Sara D. Sackett 박사 및 Jon Odorico 교수의 도움을 받아 실험실에 샘플을 제공 한 것을 인정하고 싶습니다. 우리 연구팀은이 연구를 위해 조직을 기증 한 가족에게 특별한 감사를하고 싶습니다. L.L.은 위스콘신 대학 카르본 암 센터 (233-AAI9632)의 췌장암 파일럿 보조금 인 NIH 보조금 S10OD025084와 Vilas Distinguished Achievement Professorship 및 Charles Melbourne Johnson Distinguished Chair Professorship을 인정합니다.
Materials
| Acetic Acid, Glacial (Certified ACS) | Fisher Scientific | A38S-500 | |
| Acetone (Certified ACS) | Fisher Scientific | A18-1 | |
| Acetonitrile, Optima LC/MS Grade | Fisher Scientific | A955-4 | |
| Ammonium Acetate (Crystalline/Certified ACS) | Fisher Scientific | A637-500 | |
| Ammonium Hydroxide (Certified ACS Plus) | Fisher Scientific | A669-212 | |
| Byonic software | Protein Metrics | n/a | Commercial software used for glycoproteomic analysis (https://proteinmetrics.com/byos/) |
| C18 BEH material | Waters | 186002353 | Material removed from column and used to pack nano capillaries (pulledto integrate tip used directly in line with instrument inlet) |
| CAE-Ti-IMAC, 100% | J&K Scientific | 2749380-1G | Material used for dual-functional Ti(IV)-IMAC; can also be used for conventional IMAC/conventional phosphopeptide enrichment |
| Cellcrusher kit | Cellcrusher | n/a | Used for grinding tissue samples into powder before extraction |
| Eppendorf 5424R Microcentrifuge | Fisher Scientific | 05-401-205 | For temperature-controlled centrifugation |
| cOmplete protease inhibitor cocktail tablets | Sigma | 11697498001 | |
| DTT, Molecular Grade (DL-Dithiothreitol) | Promega | V3151 | Protein reducing agent |
| Ethanol, 200 proof (100%), USP | Fisher | 22-032-601 | |
| Fisherbrand Analog Vortex Mixer | Fisher Scientific | 02-215-414 | |
| Fisherbrand Low-Retention Microcentrifuge Tubes (1.5 mL) | Fisher Scientific | 02-681-320 | |
| Fisherbrand Low-Retention Microcentrifuge Tubes (2 mL) | Fisher Scientific | 02-681-321 | |
| Fisherbrand Model 120 Sonic Dismembrator | Fisher Scientific | FB120110 | For sample lysis using ultrasonication |
| Formic Acid, 99.0+%, Optima LC/MS Grade | Fisher Scientific | A117-50 | |
| Fused silica capillary (75 μm inner diameter, 360 μm outer diameter) | Polymicro Technologies LLC | 100 m TSP075375 | For in-house pulled and packed columns with integrated emitter |
| Hydrofluoric acid (48 wt. % in H2O) | Sigma-Aldrich | 339261-100ML | Used for opening emitter of pulled capillary column |
| Iodoacetamide, BioUltra | Sigma | I1149-5G | Protein reducing reagent |
| MaxQuant software | n/a | n/a | Free software used for phosphoproteomic analysis (https://www.maxquant.org/) |
| Multi-therm Shaker with heating and cooling | Benchmark Scientific | H5000-HC | Heating block |
| Oasis HLB 1 cc Vac Cartridge, 10 mg Sorbent per Cartridge, 30 µm, 100/pk | Waters | 186000383 | Larger-scale cartridge desalting for tryptic digests (loading capacity approximately up to 1 mg each) |
| OMIX C18 pipette tips, 100 µL tip, 10 – 100 μL elution volume, 1 x 96 tips | Agilent | A57003100 | Smaller-scale packed pipette tip for desalting for enrichment elutions |
| P-2000 Micropipette Puller | Sutter Instrument Co. | P-2000/F | For pulling nano-capillary columns for LC-MS |
| PhosSTOP phosphatase inhibitor tablets | Sigma | 4906845001 | |
| Pierce BCA Protein Assay Kit | Thermo Fisher Scientific | 23225 | |
| Pierce Quantitative Colorimetric Peptide Assay | Thermo Fisher Scientific | 23275 | |
| PolySAX LP (12 μm, pore size 300 Å) | PolyLC | BMSX1203 | Material for strong anion-exchange chromatography used for ERLIC/conventional glycopeptide enrichment |
| Potassium Phosphate Monobasic (Crystalline/Certified ACS) | Fisher Scientific | P285-500 | |
| Pressure injection cell with integrated magnetic stirplate | Next Advance | PC77-MAG | For packing nano-capillary columns with stationary phase up to 2500 psi limit |
| Proteome Discoverer software | Thermo Fisher Scientific | n/a | Commercial software for proteomics anaysis (with integrated database searching software nodes) and data visualization (https://www.thermofisher.com/us/en/home/industrial/mass-spectrometry/liquid-chromatography-mass-spectrometry-lc-ms/lc-ms-software/multi-omics-data-analysis/proteome-discoverer-software.html) |
| SpeedVac SC110 Vacuum Concentrator Model SC110-120 | Savant | n/a | Centrifugal vacuum concentrator for drying samples (under heat) |
| SDS Solution, 10% Sodium Dodecyl Sulfate Solution, Molecular Biology/Electrophoresis | Fisher Scientific | BP2436200 | |
| Sequencing Grade Modified Trypsin | Promega | V5111 | |
| Sodium Chloride (Crystalline/Certified ACS) | Fisher Scientific | S271-500 | |
| TopTip, Empty, 10-200 µL, Pack of 96 | Glygen Corporation | TT2EMT.96 | Empty pipette tip with micron-sized hole used that can be used to pack chromatographic materials for enrichments, bundled with tube adapters |
| Triethylammonium bicarbonate buffer (TEAB, 1 M, pH 8.5 (volatile)) | Sigma | 90360-100ML | |
| Trifluoroacetic acid, Reagent Grade, 99% | Fisher Scientific | 60-017-61 | |
| Tris Base (White Crystals or Crystalline Powder/Molecular Biology) | Fisher Scientific | BP152-500 | |
| Trypsin/Lys-C Mix, Mass Spec Grade | Promega | V5071 | |
| Urea (Certified ACS) | Fisher Scientific | U15-500 | |
| Water, Optima LC/MS Grade | Fisher Scientific | W64 |
Riferimenti
- Smith, L. M., Kelleher, N. L. Proteoform: a single term describing protein complexity. Nature Methods. 10 (3), 186-187 (2013).
- Pan, S., Brentnall, T. A., Chen, R. Glycoproteins and glycoproteomics in pancreatic cancer. World Journal of Gastroenterology. 22 (42), 9288-9299 (2016).
- Tabang, D. N., Ford, M., Li, L. Recent advances in mass spectrometry-based glycomic and glycoproteomic studies of pancreatic diseases. Frontiers in Chemistry. 9, 707387 (2021).
- Khoury, G. A., Baliban, R. C., Floudas, C. A. Proteome-wide post-translational modification statistics: frequency analysis and curation of the swiss-prot database. Scientific Reports. 1, 90 (2011).
- Alpert, A. J. Hydrophilic-interaction chromatography for the separation of peptides, nucleic acids and other polar compounds. Journal of Chromatography A. 499, 177-196 (1990).
- Riley, N. M., Bertozzi, C. R., Pitteri, S. J. A pragmatic guide to enrichment strategies for mass spectrometry-based glycoproteomics. Molecular & Cellular Proteomics. 20, 100029 (2020).
- Low, T. Y., et al. Widening the bottleneck of phosphoproteomics: Evolving strategies for phosphopeptide enrichment. Mass Spectrometry Reviews. 40 (4), 309-333 (2021).
- Cho, K. C., Chen, L., Hu, Y., Schnaubelt, M., Zhang, H. Developing workflow for simultaneous analyses of phosphopeptides and glycopeptides. ACS Chemical Biology. 14 (1), 58-66 (2019).
- Zhou, Y., et al. An integrated workflow for global, glyco-, and phospho-proteomic analysis of tumor tissues. Analytical Chemistry. 92 (2), 1842-1849 (2020).
- Tang, R., et al. Facile preparation of bifunctional adsorbents for efficiently enriching N-glycopeptides and phosphopeptides. Analytica Chimica Acta. 1144, 111-120 (2021).
- Wang, Z., Wang, J., Sun, N., Deng, C. A promising nanoprobe based on hydrophilic interaction liquid chromatography and immobilized metal affinity chromatography for capture of glycopeptides and phosphopeptides. Analytica Chimica Acta. 1067, 1-10 (2019).
- Glover, M. S., et al. Characterization of intact sialylated glycopeptides and phosphorylated glycopeptides from IMAC enriched samples by EThcD fragmentation: Toward combining phosphoproteomics and glycoproteomics. International Journal of Mass Spectrometry. 427, 35-42 (2018).
- Alpert, A. J. Electrostatic repulsion hydrophilic interaction chromatography for isocratic separation of charged solutes and selective isolation of phosphopeptides. Analytical Chemistry. 80 (1), 62-76 (2008).
- Cui, Y., et al. Counterion optimization dramatically improves selectivity for phosphopeptides and glycopeptides in electrostatic repulsion-hydrophilic interaction chromatography. Analytical Chemistry. 93 (22), 7908-7916 (2021).
- Cui, Y., et al. Finding the sweet spot in ERLIC mobile phase for simultaneous enrichment of N-Glyco and phosphopeptides. Journal of the American Society for Mass Spectrometry. 30 (12), 2491-2501 (2019).
- Tabang, D. N., et al. Analysis of pancreatic extracellular matrix protein post-translational modifications via electrostatic repulsion-hydrophilic interaction chromatography coupled with mass spectrometry. Molecular Omics. 17 (5), 652-664 (2021).
- Huang, J., et al. Dual-functional Titanium(IV) immobilized metal affinity chromatography approach for enabling large-scale profiling of protein Mannose-6-Phosphate glycosylation and revealing its predominant substrates. Analytical Chemistry. 91 (18), 11589-11597 (2019).
- Huang, J., et al. Dual-functional Ti(IV)-IMAC material enables simultaneous enrichment and separation of diverse glycopeptides and phosphopeptides. Analytical Chemistry. 93 (24), 8568-8576 (2021).
- Jami-Alahmadi, Y., Pandey, V., Mayank, A. K., Wohlschlegel, J. A. A robust method for packing high resolution C18 RP-nano-HPLC columns. Journal of Visualized Experiments: JoVE. (171), e62380 (2021).
- Bern, M., Kil, Y. J., Becker, C. Byonic: advanced peptide and protein identification software. Current Protocols in Bioinformatics. , (2012).
- Tyanova, S., Temu, T., Cox, J. The MaxQuant computational platform for mass spectrometry-based shotgun proteomics. Nature Protocols. 11 (12), 2301-2319 (2016).
- Perez-Riverol, Y., et al. The PRIDE database and related tools and resources in 2019: improving support for quantification data. Nucleic Acids Research. 47, 442-450 (2019).
- Zhou, Y., et al. Metascape provides a biologist-oriented resource for the analysis of systems-level datasets. Nature Communications. 10 (1), 1523 (2019).
- Zacharias, L. G., et al. HILIC and ERLIC enrichment of glycopeptides derived from breast and brain cancer cells. Journal of Proteome Research. 15 (10), 3624-3634 (2016).
- Yang, W., et al. Comparison of enrichment methods for intact N- and O-linked glycopeptides using strong anion exchange and hydrophilic interaction liquid chromatography. Analytical Chemistry. 89 (21), 11193-11197 (2017).
- Toghi Eshghi, S., Shah, P., Yang, W., Li, X., Zhang, H. GPQuest: A spectral library matching algorithm for site-specific assignment of tandem mass spectra to intact N-glycopeptides. Analytical Chemistry. 87 (10), 5181-5188 (2015).
- Liu, M. -. Q., et al. pGlyco 2.0 enables precision N-glycoproteomics with comprehensive quality control and one-step mass spectrometry for intact glycopeptide identification. Nature Communications. 8 (1), 438 (2017).
- Lu, L., Riley, N. M., Shortreed, M. R., Bertozzi, C. R., Smith, L. M. O-Pair Search with MetaMorpheus for O-glycopeptide characterization. Nature Methods. 17 (11), 1133-1138 (2020).
- Caval, T., Heck, A. J. R., Reiding, K. R. Meta-heterogeneity: Evaluating and describing the diversity in glycosylation between sites on the same glycoprotein. Molecular & Cellular Proteomics. 20, 100010 (2021).
- Lee, J. S., Smith, E., Shilatifard, A. The language of histone crosstalk. Cell. 142 (5), 682-685 (2010).
- Leutert, M., Entwisle, S. W., Villén, J. Decoding post-translational modification crosstalk with proteomics. Molecular & Cellular Proteomics. 20, 100129 (2021).
- Hart, G. W., Slawson, C., Ramirez-Correa, G., Lagerlof, O. Cross talk between O-GlcNAcylation and phosphorylation: Roles in signaling, transcription, and chronic disease. Annual Review of Biochemistry. 80 (1), 825-858 (2011).

