Preparazione e analisi morfologica di colture cellulari di cresta neurale cranica di pulcino
Summary
Questo protocollo versatile descrive l’isolamento delle cellule della cresta neurale premigratoria (NCC) attraverso l’escissione di pieghe neurali craniche da embrioni di pulcino. Dopo la placcatura e l’incubazione, gli NCC migratori emergono dagli espianti di piega neurale, consentendo la valutazione della morfologia cellulare e della migrazione in un ambiente 2D semplificato.
Abstract
Durante lo sviluppo dei vertebrati, le cellule della cresta neurale (NCC) migrano ampiamente e si differenziano in vari tipi di cellule che contribuiscono a strutture come lo scheletro craniofacciale e il sistema nervoso periferico. Mentre è fondamentale comprendere la migrazione NCC nel contesto di un embrione 3D, l’isolamento delle cellule migratorie in coltura 2D facilita la visualizzazione e la caratterizzazione funzionale, integrando gli studi embrionali. Il presente protocollo dimostra un metodo per isolare le pieghe neurali craniche dei pulcini per generare colture NCC primarie. Gli NCC migratori emergono da espianti di piega neurale placcati su un substrato rivestito di fibronectina. Ciò si traduce in popolazioni NCC disperse e aderenti che possono essere valutate mediante colorazione e analisi morfologiche quantitative. Questo approccio culturale semplificato è altamente adattabile e può essere combinato con altre tecniche. Ad esempio, l’emigrazione NCC e i comportamenti migratori possono essere valutati mediante imaging time-lapse o interrogati funzionalmente includendo inibitori o manipolazioni sperimentali dell’espressione genica (ad esempio, DNA, morfolino o elettroporazione CRISPR). Grazie alla sua versatilità, questo metodo fornisce un potente sistema per studiare lo sviluppo di NCC cranici.
Introduction
Le cellule della cresta neurale (NCC) sono una popolazione di cellule transitorie negli embrioni vertebrati. Le NCC sono specificate ai bordi della piastra neurale e subiscono una transizione epiteliale-mesenchimale (EMT) per migrare dal tubo neurale dorsale1. Dopo l’EMT, le NCC si disperdono ampiamente in tutto l’embrione, differenziandosi e contribuendo a varie strutture, tra cui lo scheletro craniofacciale, il tratto di deflusso del cuore e la maggior parte del sistema nervoso periferico2. I cambiamenti nella polarità cellulare, nel citoscheletro e nelle proprietà di adesione sono alla base di questo passaggio da una popolazione cellulare preliminaria a una popolazione di cellule migratorie3. Lo studio dell’EMT e della migrazione NCC fornisce approfondimenti sui meccanismi fondamentali della motilità cellulare e informa gli sforzi per prevenire e curare i difetti alla nascita e le metastasi del cancro.
Mentre l’analisi in vivo è vitale per comprendere i processi di sviluppo NCC in un contesto embrionale, i metodi in vitro offrono accessibilità visiva e fisica che facilitano ulteriori vie sperimentali. In un ambiente 2D semplificato, è possibile valutare la morfologia NCC, le strutture citoscheletriche e la distanza migrata. Inoltre, gli effetti della perturbazione genetica o solubile dei fattori sui comportamenti migratori delle NCC mobili possono essere analizzati 4,5,6,7,8,9,10. Inoltre, NCC premigratory o migratorie isolate possono essere raccolte, raggruppate e utilizzate per metodologie ad alto rendimento per studiare la regolazione dello sviluppo delle NCC attraverso profili proteomici, trascrittomici ed epigenomici 7,11. Mentre sono disponibili metodi per la preparazione di NCC cranici da vari organismi modello di sviluppo12,13,14, questo articolo dimostra la meccanica dell’approccio per coloro che imparano per la prima volta a coltivare NCC cranici da embrioni di pulcino.
L’attuale protocollo descrive una tecnica versatile per la preparazione di colture di NCC craniche di pulcino (Figura 1). Poiché gli NCC migrano facilmente dalle pieghe neurali espiantate su un substrato di coltura, gli NCC di pulcino si separano naturalmente dal tessuto embrionale e le colture primarie sono facilmente generate. Poiché le NCC del mesencefalo migrano in massa dalle pieghe neurali craniche (di contrasto con la delaminazione prolungata, cellula per cellula nel tronco15), queste colture consistono principalmente di cellule migratorie della cresta neurale cranica, con l’escissione iniziale della piega neurale che fornisce un metodo di raccolta per le NCC premigatorie. Viene dettagliato un metodo di base per sezionare e coltivare le pieghe neurali craniche dei pulcini e vengono offerti suggerimenti per diverse applicazioni e variazioni di questo metodo.
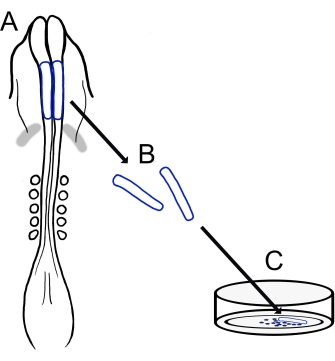
Figura 1: Panoramica schematica del protocollo di coltura neurale cranica del pulcino. (A,B) Le pieghe neurali craniche (delineate in blu) sono asportate da un embrione di pulcino con cinque somiti (mostrati in vista dorsale in A). Bande grigie, mezzaluna cardiaca. (C) Quando placcate sulla fibronectina, le cellule della cresta neurale migratoria emergono dalle pieghe neurali e si disperdono sul substrato. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Protocol
Representative Results
Discussion
La tecnica qui descritta fornisce un metodo adattabile per isolare le pieghe neurali dei pulcini e placcarle per creare colture di NCC cranici migratori. Queste colture forniscono condizioni 2D semplificate per una facile analisi della migrazione e della morfologia NCC del pulcino che possono integrare metodi di imaging più tecnicamente impegnativi in ovo 24,25,26. Mentre questo metodo in vitro è relativament…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Ringraziamo Corinne A. Fairchild e Katie L. Vermillion, che hanno partecipato allo sviluppo della nostra versione del protocollo di coltura della piega neurale cranica del pulcino.
Materials
| AxioObserver equipped with an LSM710 confocal scan head controlled by ZEN 3.0 SR software | Zeiss | Used alpha Plan-Apochromat 100x/1.46 Oil DIC M27 objective | |
| CaCl2 | Sigma-Aldrich | C3306 | |
| Chamber dishes (glass bottom, single or divided) | MatTek; Cell Vis | P35G-1.5-14-C (MatTek) X000NOJQGX (Cellvis) X000NOK1OJ (Cellvis) |
Single chamber 35 mm or 4 chamber 35 mm |
| Cover glass | Carolina Biological Supply Company | 633029, 633031, 633033, 633035, 633037 | circles, 0.13–0.17 mm thickness, available in 12-25 mm diameter |
| DMEM/F12 | ThermoFisher Scientific | 11320033 | Alternative for L15 media |
| Egg incubator | Sportsman | 1502 | |
| FBS | Life Technologies | 10437-028 | |
| Fibronectin | Fisher Scientific | CB-40008A | |
| Filter paper | Whatman | grade 3MM chromatography | |
| Forceps (blunt) | Fisher Scientific; Thomas Scientific | 08-890 (Fisher);1141W97 (Thomas) | |
| Forceps (fine) | Fine Science Tools | 11252-20 | Dumont #5 |
| Image J | https://fiji.sc/ | Free image analysis software | |
| KCl | Sigma-Aldrich | P3911 | |
| KH2PO4 | Sigma-Aldrich | P0662 | |
| L15 media | Invitrogen | 11415064 | |
| L-glutamine | Invitrogen | 25030 | |
| Mounting Media (Vectashield or ProLong Gold) | Vector Laboratories; Thermofisher Scientific | H-1700 (Vectashield); P36930 (ProLong Gold) | |
| Na2HPO4 | Sigma-Aldrich | S9638 | |
| NaCl | Sigma-Aldrich | S9888 | |
| Paraformaldehyde | Sigma-Aldrich | P6148 | |
| Penicillin/streptomycin | Life Technologies | 15140-148 | 10,000 Units/mL Penicillin; 10,000 mg/mL Streptomycin |
| Petri Dishes | VWR (or similar) | 60 mm, 100 mm | |
| Phalloidin | Sigma-Aldrich | P1951 | multiple flurophores available |
| Pin holder | Fine Science Tools | 26016-12 | For tungsten needle (alternative for spring scissors) |
| Scissors (dissection) | Fine Science Tools | 14061-10 | |
| Spring Scissors | Fine Science Tools | 15000-08 | 2.5 mm cutting edge (alternative for tungsten needle) |
| Sylgard | Krayden | Sylgard 184 | |
| Syringe Filters | Sigma-Aldrich | SLGVM33RS | Millex-GV Syringe Filter Unit, 0.22 µm, PVDF, 33 mm, gamma sterilized |
| Tissue culture dishes | Sarstedt | 83-3900 | 35 mm culture dishes for bulk neural fold cultures |
| Triton X-100 | Sigma-Aldrich | X100 | |
| Tungsten wire | Variety of sources | 0.01" diameter for tungsten needle (alternative for spring scissors) |
Riferimenti
- Pla, P., Monsoro-Burq, A. H. The neural border: Induction, specification and maturation of the territory that generates neural crest cells. Biologia dello sviluppo. 444, 36-46 (2018).
- Tang, W., Bronner, M. E. Neural crest lineage analysis: From past to future trajectory. Development. 147 (20), (2021).
- Piacentino, M. L., Li, Y., Bronner, M. E. Epithelial-to-mesenchymal transition and different migration strategies as viewed from the neural crest. Current Opinion in Cell Biology. 66, 43-50 (2020).
- McLennan, R., et al. Neural crest cells bulldoze through the microenvironment using Aquaporin 1 to stabilize filopodia. Development. 147 (1), 185231 (2020).
- Carmona-Fontaine, C., et al. Complement fragment C3a controls mutual cell attraction during collective cell migration. Developmental Cell. 21 (6), 1026-1037 (2011).
- Giovannone, D., et al. Slits affect the timely migration of neural crest cells via robo receptor. Developmental Dynamics. 241 (8), 1274-1288 (2012).
- Vermillion, K. L., Lidberg, K. A., Gammill, L. S. Cytoplasmic protein methylation is essential for neural crest migration. Journal of Cell Biology. 204 (1), 95-109 (2014).
- Yang, X., Li, J., Zeng, W., Li, C., Mao, B. Elongator Protein 3 (Elp3) stabilizes Snail1 and regulates neural crest migration in Xenopus. Scientific Reports. 6 (1), 1-9 (2016).
- Gonzalez Malagon, S. G., et al. Glycogen synthase kinase 3 controls migration of the neural crest lineage in mouse and Xenopus. Nature Communications. 9 (1), 1-15 (2018).
- Bhattacharya, D., Azambuja, A. P., Simoes-Costa, M. Metabolic reprogramming promotes neural crest migration via yap/tead signaling. Developmental Cell. 53 (2), 199-211 (2020).
- Jacques-Fricke, B. T., et al. Profiling NSD3-dependent neural crest gene expression reveals known and novel candidate regulatory factors. Biologia dello sviluppo. 475, 118-130 (2021).
- Bronner-Fraser, M., García-Castro, M. Chapter 4 manipulations of neural crest cells or their migratory pathways. Methods in Cell Biology. 87, 75-96 (2008).
- Milet, C., Monsoro-Burq, A. H. Dissection of xenopus laevis neural crest for in vitro explant culture or in vivo transplantation. Journal of Visualized Experiments. (85), e51118 (2014).
- Malagon, S. G. G., et al. Dissection, culture and analysis of primary cranial neural crest cells from mouse for the study of neural crest cell delamination and migration. Journal of Visualized Experiments. (152), e60051 (2019).
- Theveneau, E., Mayor, R. Neural crest delamination and migration: From epithelium-to-mesenchyme transition to collective cell migration. Biologia dello sviluppo. 366 (1), 34-54 (2012).
- Conrad, G. W., Bee, J. A., Roche, S. M., Teillet, M. A. Fabrication of microscalpels by electrolysis of tungsten wire in a meniscus. Journal of Neuroscience Methods. 50 (1), 123-127 (1993).
- Hamburger, V., Hamilton, H. L. A series of normal stages in the development of the chick embryo. Journal of Morphology. 88 (1), 49-92 (1951).
- Gammill, L. S., Jacques-Fricke, B., Roffers-Agarwal, J. Embryological and genetic manipulation of chick development. Methods in Molecular Biology. 1920, 75-97 (2019).
- Vandekerckhove, J., Deboben, A., Nassal, M., Wieland, T. The phalloidin binding site of F-actin. The EMBO Journal. 4 (11), 2815-2818 (1985).
- Bronner-Fraser, M. Analysis of the early stages of trunk neural crest migration in avian embryos using monoclonal antibody HNK-1. Biologia dello sviluppo. 115 (1), 44-55 (1986).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Soille, P., Vincent, L. Determining watersheds in digital pictures via flooding simulations. Visual Communications and Image Processing ’90: Fifth in a Series. (1360), 240-250 (1990).
- Haupt, A., Minc, N. How cells sense their own shape – mechanisms to probe cell geometry and their implications in cellular organization and function. Journal of Cell Science. 131 (6), (2018).
- Ezin, M., Fraser, S. Chapter 11 time-lapse imaging of the early avian embryo. Methods in Cell Biology. 87, 211-236 (2008).
- Kulesa, P. M., Bailey, C. M., Cooper, C., Fraser, S. E. In ovo live imaging of avian embryos. Cold Spring Harbor Protocols. 5 (6), (2010).
- McKinney, M. C., Kulesa, P. M. Live imaging of the neural crest cell epithelial-to-mesenchymal transition in the chick embryo. Methods in Molecular Biology. 2179, 107-114 (2021).
- Gustafson, C. M., Roffers-Agarwal, J., Gammill, L. S. Chick cranial neural crest cells release extracellular vesicles that are critical for their migration. Journal of Cell Science. , (2022).
- Williams, R., Sauka-Spengler, T. Ex ovo electroporation of early chicken embryos. STAR Protocols. 2 (2), 100424 (2021).
- Moulton, J. D. Using morpholinos to control gene expression. Current Protocols in Nucleic Acid Chemistry. 68 (1), 4-30 (2017).
- Gandhi, S., et al. A single-plasmid approach for genome editing coupled with long-term lineage analysis in chick embryos. Development. 148 (7), (2021).
- Williams, R. M., et al. Reconstruction of the Global Neural Crest Gene Regulatory Network In Vivo. Developmental Cell. 51 (2), 255-267 (2019).

