نهج قائم على قياس الطيف الكتلي لتحديد فوسفاتيز فوسفوبروتين وتفاعلاتها
Summary
هنا ، نقدم بروتوكولا لإثراء فوسفاتيز البروتين الفوسفوبروتين الداخلي المنشأ وبروتيناتها المتفاعلة من الخلايا والأنسجة وتحديدها وتحديدها كميا بواسطة البروتيوميات القائمة على قياس الطيف الكتلي.
Abstract
يتم تنظيم معظم العمليات الخلوية عن طريق فسفرة البروتين الديناميكية. أكثر من ثلاثة أرباع البروتينات مفسفرة ، وفوسفاتيز البروتين الفوسفوبروتيني (PPPs) تنسق أكثر من 90٪ من جميع عمليات إزالة الفوسفور الخلوية من السيرين / الثريونين. وقد تورط تحرير فسفرة البروتين في الفيزيولوجيا المرضية لمختلف الأمراض، بما في ذلك السرطان والتنكس العصبي. وعلى الرغم من نشاطها الواسع النطاق، فإن الآليات الجزيئية التي تتحكم في الشراكات بين القطاعين العام والخاص وتلك التي تسيطر عليها الشراكات بين القطاعين العام والخاص لا تتسم بخصائص كافية. هنا ، يتم وصف نهج بروتيني يسمى حبات مثبطات الفوسفاتيز وقياس الطيف الكتلي (PIB-MS) لتحديد وقياس PPPs ، وتعديلاتها بعد الترجمة ، ومفاعلاتها المتفاعلة في أقل من 12 ساعة باستخدام أي خط خلية أو أنسجة. يستخدم PIB-MS مثبطا غير انتقائي لتعادل القوة الشرائية ، microcystin-LR (MCLR) ، مثبتا على حبات السيفاروز لالتقاط وإثراء PPPpps الذاتية المنشأ والبروتينات المرتبطة بها (تسمى PPPome). لا تتطلب هذه الطريقة التعبير الخارجي للإصدارات الموسومة من PPPs أو استخدام أجسام مضادة محددة. يقدم PIB-MS طريقة مبتكرة لدراسة الشراكات بين القطاعين العام والخاص المحفوظة تطوريا وتوسيع فهمنا الحالي لإشارات إزالة الفسفرة.
Introduction
تتحكم فسفرة البروتين في معظم العمليات الخلوية ، بما في ذلك على سبيل المثال لا الحصر الاستجابة لتلف الحمض النووي ، وإشارات عامل النمو ، والمرور عبر الانقسام1،2،3. في خلايا الثدييات ، يتم فسفوريلات غالبية البروتينات في واحد أو أكثر من بقايا سيرين أو ثريونين أو التيروزين في وقت ما ، مع فوسفوسرين وفوسفوثريونين يشكلون حوالي 98 ٪ من جميع مواقع الفسفرة 2,3. في حين تمت دراسة الكينازات على نطاق واسع في الإشارات الخلوية ، فإن دور الشراكات بين القطاعين العام والخاص في تنظيم العمليات الخلوية الديناميكية لا يزال ناشئا.
يتم التحكم في ديناميكيات الفسفرة من خلال التفاعل الديناميكي بين الكينازات والفوسفاتيز. في خلايا الثدييات ، هناك أكثر من 400 كيناز بروتين يحفز فسفرة سيرين / ثريونين. أكثر من 90٪ من هذه المواقع يتم نزع الفوسفوريلات بواسطة فوسفاتيز البروتين الفوسفوبروتيني (PPPs) ، وهي عائلة صغيرة من الإنزيمات التي تتكون من PP1 و PP2A و PP2B و PP4-7 و PPT و PPZ 2,3. PP1 و PP2A مسؤولان عن غالبية إزالة الفوسفوسرين والفوسفوثريونين داخل الخلية2،3،4. أدى الاختلاف الملحوظ في العدد بين الكينازات والفوسفاتيز وعدم خصوصية الوحدات الفرعية الحفازة PPP في المختبر إلى الاعتقاد بأن الكينازات هي المحدد الرئيسي للفسفرة 2,3. ومع ذلك ، فقد أظهرت دراسات متعددة أن الفوسفاتيز يؤسس خصوصية الركيزة من خلال تكوين إنزيمات هولو متعددة الميريك5،6،7،8،9. على سبيل المثال ، PP1 هو غير متجانس يتكون من وحدة فرعية حفازة ، وفي وقت معين ، واحد من أكثر من 150 وحدة فرعية تنظيمية 6,7,8. على العكس من ذلك ، PP2A هو متغاير يتكون من سقالات (A) ، ووحدة تنظيمية (B) ، ووحدة فرعية حفازة (C) 2,3,9. هناك أربع عائلات متميزة من الوحدات الفرعية التنظيمية PP2A (B55 و B56 و PR72 و striatin) ، لكل منها جينات متعددة ، ومتغيرات الربط ، وأنماط التوطين2،3،9. وتسد الطبيعة المتعددة للشراكات بين القطاعين العام والخاص الفجوة في عدد الكينازات والوحدات الفرعية الحفازة لتعادل القوة الشرائية. ومع ذلك ، فإنه يخلق تحديات تحليلية لدراسة إشارات الشراكة بين القطاعين العام والخاص. لتحليل إشارات PPP بشكل شامل ، من الأهمية بمكان التحقيق في مختلف الإنزيمات الهولونية داخل الخلية أو الأنسجة. تم إحراز تقدم كبير في دراسة الكينومي البشري من خلال استخدام حبات مثبطات الكيناز ، التي يطلق عليها اسم حبات مثبطات الإرسال المتعددة أو الخرز ، وهي استراتيجية بروتينية كيميائية حيث يتم تجميد مثبطات الكيناز على الخرز ويستخدم قياس الطيف الكتلي لتحديد الكينازات المخصبة وتفاعلها10،11،12،13.
لقد أنشأنا نهجا مشابها لدراسة بيولوجيا الشراكة بين القطاعين العام والخاص. تتضمن هذه التقنية التقاط التقارب للوحدات الفرعية الحفازة PPP باستخدام الخرز مع مثبط PPP غير انتقائي غير متحرك يسمى microcystin-LR (MCLR) يسمى حبات مثبطات الفوسفاتيز (PIBs) 14,15. على عكس الطرق الأخرى التي تتطلب وضع علامات داخلية أو التعبير عن الوحدات الفرعية لتعادل القوة الشرائية الخارجية التي يمكن أن تغير نشاط البروتين أو توطينه ، يسمح PIB-MS بإثراء الوحدات الفرعية المحفزة لتعادل القوة الشرائية الذاتية المنشأ ، والوحدات الفرعية التنظيمية والسقالات المرتبطة بها ، والبروتينات المتفاعلة (تسمى PPPome) من الخلايا والأنسجة في نقطة زمنية معينة أو في ظل ظروف علاج محددة. يمنع MCLR PP1 و PP2A و PP4-6 و PPT و PPZ بتركيزات نانومولية ، مما يجعل PIBs فعالة للغاية في إثراء PPPome16. يمكن تحجيم هذه الطريقة لاستخدامها على أي مادة أولية من الخلايا إلى العينات السريرية. هنا ، نصف بالتفصيل استخدام PIBs وقياس الطيف الكتلي (PIB-MS) لالتقاط وتحديد وقياس PPPome الداخلي وحالات تعديله بكفاءة.
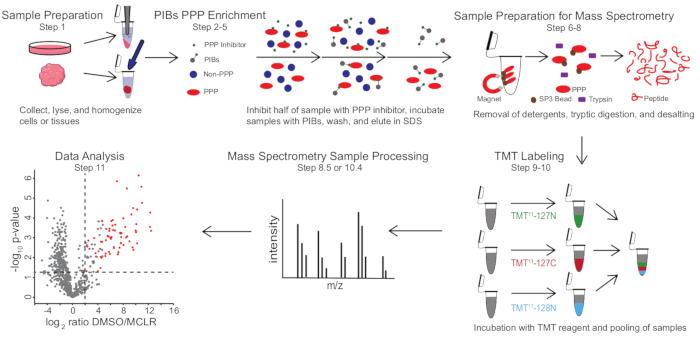
الشكل 1: ملخص مرئي لبروتوكول PIB-MS. في تجربة PIB-MS ، يمكن الحصول على عينات بأشكال مختلفة ، من الخلايا إلى الأورام. يتم جمع العينة وتحليلها وتجانسها قبل إثراء تعادل القوة الشرائية. للإثراء من أجل الشراكات بين القطاعين العام والخاص، يتم احتضان الليزات مع PIBs مع أو بدون مثبط PPP، مثل MCLR. ثم يتم غسل PIBs ، ويتم التخلص من PPPs في ظروف تمسخ. يتم إعداد العينات لتحليل الطيف الكتلي عن طريق إزالة المنظفات من خلال إثراء البروتين SP3 ، والهضم التربتيكي ، وإزالة الملح. يمكن بعد ذلك وضع علامة TMT اختياريا على العينات قبل تحليل مطياف الكتلة. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
يتضمن PIB-MS تحلل وتوضيح الخلايا أو الأنسجة ، وحضانة الليزات باستخدام PIBs ، والاستخلاص ، وتحليل اللوات عبر النشاف الغربي أو النهج القائمة على قياس الطيف الكتلي (الشكل 1). يمكن استخدام إضافة MCLR المجاني كعنصر تحكم لتمييز روابط PIB المحددة عن الجهات الفاعلة غير المحددة. بالنسبة لمعظم التطبيقات ، يمكن استخدام نهج خال من الملصقات لتحديد البروتينات مباشرة في المراوغة. وفي الحالات التي تكون فيها هناك حاجة إلى مزيد من الدقة في التحديد الكمي أو تحديد الأنواع منخفضة الوفرة، يمكن استخدام مزيد من المعالجة باستخدام وسم العلامات الجماعية الترادفية (TMT) لزيادة التغطية وتقليل المدخلات.
Protocol
Representative Results
Discussion
PIB-MS هو نهج بروتينات كيميائية يستخدم لتحديد ملامح PPPome كميا من مصادر عينة مختلفة في تحليل واحد. تم القيام بالكثير من العمل باستخدام حبات مثبطات كيناز لدراسة الكينوم وكيف يتغير في السرطان والحالات المرضية الأخرى10،11،12،13. …
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
تعترف A.N.K. بالدعم المقدم من NIH R33 CA225458 و R35 GM119455. نشكر مختبري Kettenbach و Gerber على مناقشتهما المفيدة.
Materials
| Acetonitrile (ACN) | Honeywell | AH015-4 | CAUTION: ACN is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Anhydrous Acetonitrile | Sigma-Aldrich | 271004-100ML | CAUTION: ACN is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Benchtop centrifuge | Eppendorf | model no. 5424 | |
| Beta-glycerophosphoric acid, disodium salt pentahydrate | Acros Organics | 410991000 | |
| Centrifuge | Eppendorf | model no. 5810 R 15 amp version | |
| Distilled water | |||
| DMSO | Fisher Scientific | BP231-100 | |
| Dounce tissue grinder | Fisherbrand Pellet Pestles | 12-141-363 | |
| Empore solid phase extraction disk, C18 | CDS Analytical | 76333-132 | |
| Eppendorf tubes, 1.5 mL | Eppendorf | 22363204 | CRITICAL: Other tubes may leach polymer into sample, contaminating the analysis. |
| Eppendorf tubes, 2 mL | Eppendorf | 22363352 | CRITICAL: Other tubes may leach polymer into sample, contaminating the analysis. |
| Extraction plate manifold | Waters | WAT097944 | |
| Falcon tubes, 50 mL | VWR | 21008 | |
| Generic blunt end needle and plunger | |||
| Generic magnetic separation rack | |||
| HEPES | Sigma-Aldrich | H3375 | |
| Hydrogen chloride (HCl) | VWR Chemicals BDH | BDH3028 | CAUTION: HCl is corrosive; wear gloves and work in a chemical fume hood. |
| Hydroxylamine solution 50% (wt/vol) | Sigma-Aldrich | 467804 | |
| Incubator, 65 °C | VWR | model no. 1380FM | |
| Koptec Pure Ethanol, 200 Proof | Decon Labs | V1001 | |
| Methanol for HPLC (MeOH) | Sigma-Aldrich | 34860-4L-R | CAUTION: MeOH is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Microcystin LR (MCLR) | Cayman Chemical | 10007188 | CAUTION: MCLR is toxic; wear gloves when handling and avoid skin contact. |
| PBS, 1× without calcium and magnesium, pH 7.4 ± 0.1 | Corning | 21-040-CV | |
| pH test strips, such as MilliporeSigma MColorpHast pH test strips and indicator papers | Fisher Scientific | M1095310001 | |
| PIBs | For protocol for the generation of PIBs, see Moorhead et al., 2007. | ||
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23225 | |
| Pipette tips, 10 μL | Eppendorf | 22491504 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| Pipette tips, 1000 μL | Eppendorf | 22491555 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| Pipette tips, 200 μL | Eppendorf | 22491539 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| plastic syringe, 10 mL | BD | 309604 | |
| Protease inhibitor cocktail III | Research Products International | P50700-1 | |
| Q Exactive Plus Hybrid Quadrupole-Orbitrap Mass Spectrometer, Oribtrap Fusion, Orbitrap Fusion Lumos, or Orbitrap Eclipse Tribrid Mass Spectrometer | Thermo Scientific | ||
| Refrigerated benchtop centrifuge | Eppendorf | model no. 5424 R | |
| Rotator (Labquake Shaker Rotisserie) | Thermo Scientific | 13-687-12Q | 8 rpm rotation |
| Sample collection plate, 96- well, 1 mL | Waters | WAT058957 | |
| SDS | Fisher Scientific | BP1311-1 | |
| Sequencing grade modified trypsin | Promega | V511C | |
| Sodium azide | EMD Chemicals | SX0299-1 | CAUTION: Sodium azide is explosive and toxic; wear gloves, work in a chemical fume hood and avoid contact with metals. |
| Sodium chloride (NaCl) | Fisher Chemical | S27110 | |
| Sonicator (Branson digital sonifier) | model no. SFX 250 | ||
| SPE C18 desalting plate | Waters | 186001828BA | |
| SpeedBeads magnetic carboxylate modified particles (SP3 beads) | Cytiva | 6.51521E+13 | |
| Thermomixer | Eppendorf | model no. 5350 | |
| TMT10plex Isobaric Label Reagent Set plus TMT11-131C Label Reagent, 3 × 0.8 mg per tag | ThermoFisher | A37725 | |
| Trifluoroacetic acid (TFA) | Honeywell | T6508-25ML | CAUTION: TFA is corrosive and will irritate skin on contact. Wear gloves and eye protection, and work in a chemical fume hood. |
| Tris Base | Research Products International | T60040 | |
| Triton X-100 | Sigma-Aldrich | T9284 | |
| Vacuum centrifuge and vapor trap | Thermo Scientific | model nos. SpeedVac SPD120 and RVT5105 | |
| Vortexer (Vortex-Genie 2) | Scientific Industries | ||
| Water LC-MS | Honeywell | LC365-4 |
Riferimenti
- Nilsson, J. Protein phosphatases in the regulation of mitosis. Journal of Cell Biology. 218 (2), 395-409 (2019).
- Brautigan, D. L. Protein Ser/Thr phosphatases–the ugly ducklings of cell signalling. The FEBS Journal. 280 (2), 324-345 (2013).
- Brautigan, D. L., Shenolikar, S. Protein serine/threonine phosphatases: keys to unlocking regulators and substrates. Annual Review of Biochemistry. 87, 921-964 (2018).
- Janssens, V., Goris, J. Protein phosphatase 2A: A highly regulated family of serine/threonine phosphatases implicated in cell growth and signalling. Biochemical Journal. 353, 417-439 (2001).
- Virshup, D. M., Shenolikar, S. From promiscuity to precision: protein phosphatases get a makeover. Molecular Cell. 33 (5), 537-545 (2009).
- Bollen, M., Peti, W., Ragusa, M. J., Beullens, M. The extended PP1 toolkit: designed to create specificity. Trends in Biochemical Sciences. 35 (8), 450-458 (2010).
- Qian, J., Winkler, C., Bollen, M. 4D-networking by mitotic phosphatases. Current Opinion in Cell Biology. 25 (6), 697-703 (2013).
- Heroes, E., et al. The PP1 binding code: a molecular-lego strategy that governs specificity. The FEBS Journal. 280 (2), 584-595 (2013).
- Eichhorn, P. J., Creyghton, M. P., Bernards, R. Protein phosphatase 2A regulatory subunits and cancer. Biochimica et Biophysica Acta. 1795 (1), 1-15 (2009).
- Bantscheff, M., et al. Quantitative chemical proteomics reveals mechanisms of action of clinical ABL kinase inhibitors. Nature Biotechnology. 25 (9), 1035-1044 (2007).
- Klaeger, S., et al. Chemical proteomics reveals ferrochelatase as a common off-target of kinase inhibitors. ACS Chemical Biology. 11 (5), 1245-1254 (2016).
- Duncan, J. S., et al. Dynamic reprogramming of the kinome in response to targeted MEK inhibition in triple-negative breast cancer. Cell. 149 (2), 307-321 (2012).
- Cooper, M. J., et al. Application of multiplexed kinase inhibitor beads to study kinome adaptations in drug-resistant leukemia. PLoS ONE. 8 (6), 66755 (2013).
- Lyons, S. P., et al. A quantitative chemical proteomic strategy for profiling phosphoprotein phosphatases from yeast to humans. Molecular and Cellular Proteomics. 17 (12), 2448-2461 (2018).
- Nasa, I., et al. Quantitative kinase and phosphatase profiling reveal that CDK1 phosphorylates PP2Ac to promote mitotic entry. Science Signaling. 13 (648), (2020).
- Swingle, M., Ni, L., Honkanen, R. E. Small-molecule inhibitors of ser/thr protein phosphatases: specificity, use and common forms of abuse. Methods in Molecular Biology. 365, 23-38 (2007).
- Moorhead, G. B. G., Haystead, T. A. J., MacKintosh, C. Synthesis and use of the protein phosphatase affinity matrices microcystin-sepharose and microcystin-biotin-sepharose. Methods in Molecular Biology. 365, 39-45 (2007).
- Brauer, B. L., Wiredu, K., Mitchell, S., Moorhead, G. B., Gerber, S. A., Kettenbach, A. N. Affinity-based profiling of endogenous phosphoprotein phosphatases by mass spectrometry. Nature Protocols. 16 (10), 4919-4943 (2021).
- Hughes, C. S., Moggridge, S., Müller, T., Sorensen, P. H., Morin, G. B., Krijgsveld, J. Single-pot, solid-phase-enhanced sample preparation for proteomics experiments. Nature Protocols. 14 (1), 68-85 (2019).
- Rappsilber, J., Mann, M., Ishihama, Y. Protocol for micro-purification, enrichment, pre-fractionation and storage of peptides for proteomics using StageTips. Nature Protocols. 2 (8), 1896-1906 (2007).
- Zecha, J., et al. TMT labeling for the masses: A robust and cost-efficient, in-solution labeling approach. Molecular and Cellular Proteomics. 18 (7), 1468-1478 (2019).
- Elias, J. E., Gygi, S. P. Target-decoy search strategy for increased confidence in large-scale protein identifications by mass spectrometry. Nature Methods. 4 (3), 207-214 (2007).
- Eng, J. K., Jahan, T. A., Hoopmann, M. R. Comet: an open-source MS/MS sequence database search tool. Proteomics. 13 (1), 22-24 (2013).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nature Methods. 13 (9), 731-740 (2016).
- Yu, S. H., Ferretti, D., Schessner, J. P., Rudolph, J. D., Borner, G. H. H., Cox, J. Expanding the Perseus software for omics data analysis With custom plugins. Current Protocols in Bioinformatics. 71 (1), 1-29 (2020).

