Ein massenspektrometriebasierter Ansatz zur Identifizierung von Phosphoproteinphosphatasen und ihren Interaktoren
Summary
Hier stellen wir ein Protokoll zur Anreicherung von endogenen Phosphoproteinphosphatasen und ihren interagierenden Proteinen aus Zellen und Geweben und deren Identifizierung und Quantifizierung durch massenspektrometriebasierte Proteomik vor.
Abstract
Die meisten zellulären Prozesse werden durch dynamische Proteinphosphorylierung reguliert. Mehr als drei Viertel der Proteine sind phosphoryliert, und Phosphoproteinphosphatasen (PPPs) koordinieren über 90% der gesamten zellulären Serin/Threonin-Dephosphorylierung. Die Deregulierung der Proteinphosphorylierung wurde mit der Pathophysiologie verschiedener Krankheiten, einschließlich Krebs und Neurodegeneration, in Verbindung gebracht. Trotz ihrer weit verbreiteten Aktivität sind die molekularen Mechanismen, die PPPs kontrollieren, und diejenigen, die von PPPs kontrolliert werden, schlecht charakterisiert. Hier wird ein proteomischer Ansatz beschrieben, der als Phosphatase-Inhibitor-Beads und Massenspektrometrie (PIB-MS) bezeichnet wird, um PPPs, ihre posttranslationalen Modifikationen und ihre Interaktoren in nur 12 h unter Verwendung einer Zelllinie oder eines Gewebes zu identifizieren und zu quantifizieren. PIB-MS verwendet einen nicht-selektiven PPP-Inhibitor, Microcystin-LR (MCLR), der auf Sepharosekügelchen immobilisiert wird, um endogene PPPs und ihre assoziierten Proteine (PPPom genannt) einzufangen und anzureichern. Diese Methode erfordert weder die exogene Expression markierter Versionen von PSM noch die Verwendung spezifischer Antikörper. PIB-MS bietet eine innovative Möglichkeit, die evolutionär konservierten PPPs zu untersuchen und unser derzeitiges Verständnis der Dephosphorylierungssignalisierung zu erweitern.
Introduction
Die Proteinphosphorylierung steuert die meisten zellulären Prozesse, einschließlich, aber nicht beschränkt auf die Reaktion auf DNA-Schäden, die Signalisierung des Wachstumsfaktors und die Passage durch Mitose 1,2,3. In Säugetierzellen wird die Mehrheit der Proteine zu einem bestimmten Zeitpunkt an einem oder mehreren Serin-, Threonin- oder Tyrosinresten phosphoryliert, wobei Phosphosrine und Phosphothreonine etwa 98% aller Phosphorylierungsstellen ausmachen 2,3. Während Kinasen in der zellulären Signalgebung ausführlich untersucht wurden, zeichnet sich die Rolle von PPPs bei der Regulation dynamischer zellulärer Prozesse noch ab.
Die Phosphorylierungsdynamik wird durch das dynamische Zusammenspiel von Kinasen und Phosphatasen gesteuert. In Säugetierzellen gibt es mehr als 400 Proteinkinasen, die die Serin/Threonin-Phosphorylierung katalysieren. Über 90% dieser Stellen werden durch Phosphoproteinphosphatasen (PPPs) dephosphoryliert, eine kleine Familie von Enzymen, die aus PP1, PP2A, PP2B, PP4-7, PPT und PPZ 2,3 besteht. PP1 und PP2A sind für den Großteil der Phosphoserin- und Phosphothreonin-Dephosphorylierung innerhalb einer Zelle verantwortlich 2,3,4. Der bemerkenswerte Unterschied in der Anzahl zwischen Kinasen und Phosphatasen und die mangelnde Spezifität der PPP-katalytischen Untereinheiten in vitro führten zu der Annahme, dass Kinasen die Hauptdeterminante der Phosphorylierungsind 2,3. Mehrere Studien haben jedoch gezeigt, dass Phosphatasen die Substratspezifität durch die Bildung multimerer Holoenzyme 5,6,7,8,9 etablieren. Zum Beispiel ist PP1 ein Heterodimer, das aus einer katalytischen Untereinheit und zu einem bestimmten Zeitpunkt aus einer der mehr als 150 regulatorischen Untereinheiten 6,7,8 besteht. Umgekehrt ist PP2A ein Heterotrimer, der aus einem Gerüst (A), einer regulatorischen (B) und einer katalytischen (C)Untereinheit 2,3,9 besteht. Es gibt vier verschiedene Familien von regulatorischen PP2A-Untereinheiten (B55, B56, PR72 und Striatin), jede mit mehreren Genen, Spleißvarianten und Lokalisationsmustern 2,3,9. Die multimere Natur von PPPs füllt die Lücke in der Anzahl der Kinasen und PPP-katalytischen Untereinheiten. Es schafft jedoch analytische Herausforderungen für die Untersuchung der PPP-Signalgebung. Um die PPP-Signalgebung umfassend analysieren zu können, ist es wichtig, die verschiedenen Holoenzyme in einer Zelle oder einem Gewebe zu untersuchen. Große Fortschritte wurden bei der Untersuchung des menschlichen Kinoms durch die Verwendung von Kinase-Inhibitor-Perlen erzielt, die als Multiplex-Inhibitor-Perlen oder Kinobeads bezeichnet werden, eine chemische Proteomstrategie, bei der Kinase-Inhibitoren auf Perlen immobilisiert werden und Massenspektrometrie verwendet wird, um angereicherte Kinasen und ihre Interaktorenzu identifizieren 10,11,12,13.
Wir haben einen ähnlichen Ansatz für das Studium der PPP-Biologie etabliert. Diese Technik beinhaltet die Affinitätserfassung von PPP-katalytischen Untereinheiten unter Verwendung von Perlen mit einem immobilisierten, nicht-selektiven PPP-Inhibitor namens Microcystin-LR (MCLR), der als Phosphatase-Inhibitor-Perlen (PIBs) bezeichnet wird14,15. Im Gegensatz zu anderen Methoden, die die endogene Markierung oder Expression exogener PPP-Untereinheiten erfordern, die die Proteinaktivität oder -lokalisation verändern könnten, ermöglicht PIB-MS die Anreicherung von endogenen PPP-katalytischen Untereinheiten, ihren assoziierten regulatorischen und gerüstbildenden Untereinheiten und interagierenden Proteinen (als PPPom bezeichnet) aus Zellen und Geweben zu einem bestimmten Zeitpunkt oder unter bestimmten Behandlungsbedingungen. MCLR hemmt PP1, PP2A, PP4-6, PPT und PPZ bei nanomolaren Konzentrationen, wodurch PIBs bei der Anreicherung für dasPPPome 16 hochwirksam sind. Diese Methode kann für den Einsatz auf jedem Ausgangsmaterial von Zellen bis hin zu klinischen Proben skaliert werden. Hier beschreiben wir detailliert den Einsatz von PIBs und Massenspektrometrie (PIB-MS), um das endogene PPPom und seine Modifikationszustände effizient zu erfassen, zu identifizieren und zu quantifizieren.
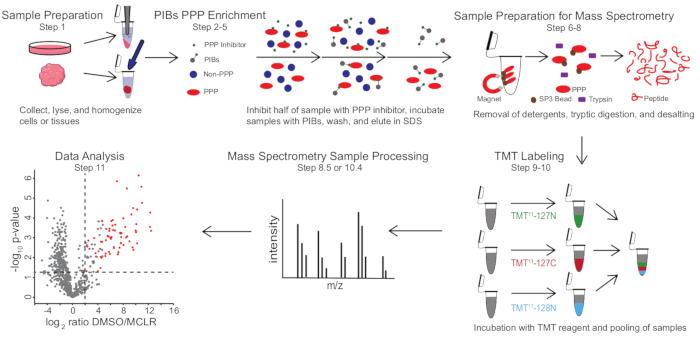
Abbildung 1: Visuelle Zusammenfassung des PIB-MS-Protokolls. In einem PIB-MS-Experiment können Proben in verschiedenen Formen gewonnen werden, von Zellen bis hin zu Tumoren. Die Probe wird vor der PPP-Anreicherung gesammelt, lysiert und homogenisiert. Zur Anreicherung für PPPs wird das Lysat mit PIBs mit oder ohne PPP-Inhibitor, wie MCLR, inkubiert. Die PIBs werden dann gewaschen und PPPs werden unter Denaturierungsbedingungen eluiert. Die Proben werden für die massenspektrometrische Analyse durch Entfernen von Reinigungsmitteln durch SP3-Proteinanreicherung, tryptische Verdauung und Entsalzung vorbereitet. Proben können dann vor der massenspektrometrischen Analyse optional TMT-markiert werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
PIB-MS umfasst die Lyse und Klärung von Zellen oder Geweben, die Inkubation des Lysats mit PIBs, die Elution und die Analyse des Eluats über westliche Blotting- oder Massenspektrometrie-basierte Ansätze (Abbildung 1). Die Zugabe von freiem MCLR kann als Steuerung verwendet werden, um spezifische PIB-Bindemittel von unspezifischen Interaktoren zu unterscheiden. Für die meisten Anwendungen kann ein markierungsfreier Ansatz verwendet werden, um Proteine in Eluaten direkt zu identifizieren. In Fällen, in denen eine genauere Quantifizierung oder die Identifizierung von Arten mit geringer Häufigkeit erforderlich ist, kann die Weiterverarbeitung mit Tandem-Massen-Tag-Kennzeichnung (TMT) verwendet werden, um die Abdeckung zu erhöhen und den Input zu verringern.
Protocol
Representative Results
Discussion
PIB-MS ist ein chemischer Proteomik-Ansatz, der verwendet wird, um das PPPom aus verschiedenen Probenquellen in einer einzigen Analyse quantitativ zu profilieren. Es wurde viel Arbeit mit Kinase-Inhibitor-Perlen geleistet, um das Kinom zu untersuchen und wie es sich bei Krebs und anderen Krankheitszuständenverändert 10,11,12,13. Die Untersuchung des PPPoms hinkt jedoch hinterher. Wir gehen da…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
A.N.K. erkennt die Unterstützung von NIH R33 CA225458 und R35 GM119455 an. Wir danken den Laboren Kettenbach und Gerber für das hilfreiche Gespräch.
Materials
| Acetonitrile (ACN) | Honeywell | AH015-4 | CAUTION: ACN is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Anhydrous Acetonitrile | Sigma-Aldrich | 271004-100ML | CAUTION: ACN is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Benchtop centrifuge | Eppendorf | model no. 5424 | |
| Beta-glycerophosphoric acid, disodium salt pentahydrate | Acros Organics | 410991000 | |
| Centrifuge | Eppendorf | model no. 5810 R 15 amp version | |
| Distilled water | |||
| DMSO | Fisher Scientific | BP231-100 | |
| Dounce tissue grinder | Fisherbrand Pellet Pestles | 12-141-363 | |
| Empore solid phase extraction disk, C18 | CDS Analytical | 76333-132 | |
| Eppendorf tubes, 1.5 mL | Eppendorf | 22363204 | CRITICAL: Other tubes may leach polymer into sample, contaminating the analysis. |
| Eppendorf tubes, 2 mL | Eppendorf | 22363352 | CRITICAL: Other tubes may leach polymer into sample, contaminating the analysis. |
| Extraction plate manifold | Waters | WAT097944 | |
| Falcon tubes, 50 mL | VWR | 21008 | |
| Generic blunt end needle and plunger | |||
| Generic magnetic separation rack | |||
| HEPES | Sigma-Aldrich | H3375 | |
| Hydrogen chloride (HCl) | VWR Chemicals BDH | BDH3028 | CAUTION: HCl is corrosive; wear gloves and work in a chemical fume hood. |
| Hydroxylamine solution 50% (wt/vol) | Sigma-Aldrich | 467804 | |
| Incubator, 65 °C | VWR | model no. 1380FM | |
| Koptec Pure Ethanol, 200 Proof | Decon Labs | V1001 | |
| Methanol for HPLC (MeOH) | Sigma-Aldrich | 34860-4L-R | CAUTION: MeOH is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Microcystin LR (MCLR) | Cayman Chemical | 10007188 | CAUTION: MCLR is toxic; wear gloves when handling and avoid skin contact. |
| PBS, 1× without calcium and magnesium, pH 7.4 ± 0.1 | Corning | 21-040-CV | |
| pH test strips, such as MilliporeSigma MColorpHast pH test strips and indicator papers | Fisher Scientific | M1095310001 | |
| PIBs | For protocol for the generation of PIBs, see Moorhead et al., 2007. | ||
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23225 | |
| Pipette tips, 10 μL | Eppendorf | 22491504 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| Pipette tips, 1000 μL | Eppendorf | 22491555 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| Pipette tips, 200 μL | Eppendorf | 22491539 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| plastic syringe, 10 mL | BD | 309604 | |
| Protease inhibitor cocktail III | Research Products International | P50700-1 | |
| Q Exactive Plus Hybrid Quadrupole-Orbitrap Mass Spectrometer, Oribtrap Fusion, Orbitrap Fusion Lumos, or Orbitrap Eclipse Tribrid Mass Spectrometer | Thermo Scientific | ||
| Refrigerated benchtop centrifuge | Eppendorf | model no. 5424 R | |
| Rotator (Labquake Shaker Rotisserie) | Thermo Scientific | 13-687-12Q | 8 rpm rotation |
| Sample collection plate, 96- well, 1 mL | Waters | WAT058957 | |
| SDS | Fisher Scientific | BP1311-1 | |
| Sequencing grade modified trypsin | Promega | V511C | |
| Sodium azide | EMD Chemicals | SX0299-1 | CAUTION: Sodium azide is explosive and toxic; wear gloves, work in a chemical fume hood and avoid contact with metals. |
| Sodium chloride (NaCl) | Fisher Chemical | S27110 | |
| Sonicator (Branson digital sonifier) | model no. SFX 250 | ||
| SPE C18 desalting plate | Waters | 186001828BA | |
| SpeedBeads magnetic carboxylate modified particles (SP3 beads) | Cytiva | 6.51521E+13 | |
| Thermomixer | Eppendorf | model no. 5350 | |
| TMT10plex Isobaric Label Reagent Set plus TMT11-131C Label Reagent, 3 × 0.8 mg per tag | ThermoFisher | A37725 | |
| Trifluoroacetic acid (TFA) | Honeywell | T6508-25ML | CAUTION: TFA is corrosive and will irritate skin on contact. Wear gloves and eye protection, and work in a chemical fume hood. |
| Tris Base | Research Products International | T60040 | |
| Triton X-100 | Sigma-Aldrich | T9284 | |
| Vacuum centrifuge and vapor trap | Thermo Scientific | model nos. SpeedVac SPD120 and RVT5105 | |
| Vortexer (Vortex-Genie 2) | Scientific Industries | ||
| Water LC-MS | Honeywell | LC365-4 |
Riferimenti
- Nilsson, J. Protein phosphatases in the regulation of mitosis. Journal of Cell Biology. 218 (2), 395-409 (2019).
- Brautigan, D. L. Protein Ser/Thr phosphatases–the ugly ducklings of cell signalling. The FEBS Journal. 280 (2), 324-345 (2013).
- Brautigan, D. L., Shenolikar, S. Protein serine/threonine phosphatases: keys to unlocking regulators and substrates. Annual Review of Biochemistry. 87, 921-964 (2018).
- Janssens, V., Goris, J. Protein phosphatase 2A: A highly regulated family of serine/threonine phosphatases implicated in cell growth and signalling. Biochemical Journal. 353, 417-439 (2001).
- Virshup, D. M., Shenolikar, S. From promiscuity to precision: protein phosphatases get a makeover. Molecular Cell. 33 (5), 537-545 (2009).
- Bollen, M., Peti, W., Ragusa, M. J., Beullens, M. The extended PP1 toolkit: designed to create specificity. Trends in Biochemical Sciences. 35 (8), 450-458 (2010).
- Qian, J., Winkler, C., Bollen, M. 4D-networking by mitotic phosphatases. Current Opinion in Cell Biology. 25 (6), 697-703 (2013).
- Heroes, E., et al. The PP1 binding code: a molecular-lego strategy that governs specificity. The FEBS Journal. 280 (2), 584-595 (2013).
- Eichhorn, P. J., Creyghton, M. P., Bernards, R. Protein phosphatase 2A regulatory subunits and cancer. Biochimica et Biophysica Acta. 1795 (1), 1-15 (2009).
- Bantscheff, M., et al. Quantitative chemical proteomics reveals mechanisms of action of clinical ABL kinase inhibitors. Nature Biotechnology. 25 (9), 1035-1044 (2007).
- Klaeger, S., et al. Chemical proteomics reveals ferrochelatase as a common off-target of kinase inhibitors. ACS Chemical Biology. 11 (5), 1245-1254 (2016).
- Duncan, J. S., et al. Dynamic reprogramming of the kinome in response to targeted MEK inhibition in triple-negative breast cancer. Cell. 149 (2), 307-321 (2012).
- Cooper, M. J., et al. Application of multiplexed kinase inhibitor beads to study kinome adaptations in drug-resistant leukemia. PLoS ONE. 8 (6), 66755 (2013).
- Lyons, S. P., et al. A quantitative chemical proteomic strategy for profiling phosphoprotein phosphatases from yeast to humans. Molecular and Cellular Proteomics. 17 (12), 2448-2461 (2018).
- Nasa, I., et al. Quantitative kinase and phosphatase profiling reveal that CDK1 phosphorylates PP2Ac to promote mitotic entry. Science Signaling. 13 (648), (2020).
- Swingle, M., Ni, L., Honkanen, R. E. Small-molecule inhibitors of ser/thr protein phosphatases: specificity, use and common forms of abuse. Methods in Molecular Biology. 365, 23-38 (2007).
- Moorhead, G. B. G., Haystead, T. A. J., MacKintosh, C. Synthesis and use of the protein phosphatase affinity matrices microcystin-sepharose and microcystin-biotin-sepharose. Methods in Molecular Biology. 365, 39-45 (2007).
- Brauer, B. L., Wiredu, K., Mitchell, S., Moorhead, G. B., Gerber, S. A., Kettenbach, A. N. Affinity-based profiling of endogenous phosphoprotein phosphatases by mass spectrometry. Nature Protocols. 16 (10), 4919-4943 (2021).
- Hughes, C. S., Moggridge, S., Müller, T., Sorensen, P. H., Morin, G. B., Krijgsveld, J. Single-pot, solid-phase-enhanced sample preparation for proteomics experiments. Nature Protocols. 14 (1), 68-85 (2019).
- Rappsilber, J., Mann, M., Ishihama, Y. Protocol for micro-purification, enrichment, pre-fractionation and storage of peptides for proteomics using StageTips. Nature Protocols. 2 (8), 1896-1906 (2007).
- Zecha, J., et al. TMT labeling for the masses: A robust and cost-efficient, in-solution labeling approach. Molecular and Cellular Proteomics. 18 (7), 1468-1478 (2019).
- Elias, J. E., Gygi, S. P. Target-decoy search strategy for increased confidence in large-scale protein identifications by mass spectrometry. Nature Methods. 4 (3), 207-214 (2007).
- Eng, J. K., Jahan, T. A., Hoopmann, M. R. Comet: an open-source MS/MS sequence database search tool. Proteomics. 13 (1), 22-24 (2013).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nature Methods. 13 (9), 731-740 (2016).
- Yu, S. H., Ferretti, D., Schessner, J. P., Rudolph, J. D., Borner, G. H. H., Cox, J. Expanding the Perseus software for omics data analysis With custom plugins. Current Protocols in Bioinformatics. 71 (1), 1-29 (2020).

