גישה מבוססת ספקטרומטריית מסות לזיהוי פוספטזות פוספופרוטאין והאינטראקציות שלהם
Summary
כאן אנו מציגים פרוטוקול להעשרת פוספטזות פוספופרוטאין אנדוגניות והחלבונים המתקשרים ביניהן מתאים ורקמות וזיהוי וכימות שלהם על ידי פרוטאומיקה מבוססת ספקטרומטריית מסות.
Abstract
רוב התהליכים התאיים מווסתים על ידי זרחון חלבונים דינמי. יותר משלושה רבעים מהחלבונים עוברים זרחון, ופוספופרוטאין פוספטזות (PPPs) מתאמות יותר מ-90% מכלל הדפוספורילציה של סרין/תראונין תאי. דה-רגולציה של זרחון חלבונים הייתה מעורבת בפתופיזיולוגיה של מחלות שונות, כולל סרטן וניוון עצבי. למרות פעילותם הנרחבת, המנגנונים המולקולריים השולטים ב-PPPs וכאלה הנשלטים על ידי PPPs מאופיינים בצורה גרועה. כאן מתוארת גישה פרוטאומית המכונה חרוזי מעכבי פוספטאז וספקטרומטריית מסה (PIB-MS) כדי לזהות ולכמת PPPs, את השינויים הפוסט-טרנסלציוניים שלהם ואת האינטראקטיביסטים שלהם תוך 12 שעות בלבד באמצעות כל קו תאים או רקמה. PIB-MS משתמש במעכב PPP לא סלקטיבי, microcystin-LR (MCLR), המשותק על חרוזי ספארוז כדי ללכוד ולהעשיר PPPs אנדוגניים ואת החלבונים הקשורים אליהם (המכונים PPPome). שיטה זו אינה דורשת ביטוי אקסוגני של גרסאות מתויגות של PPPs או שימוש בנוגדנים ספציפיים. PIB-MS מציעה דרך חדשנית לחקור את ה-PPPs השמורים מבחינה אבולוציונית ולהרחיב את ההבנה הנוכחית שלנו של איתות דה-פוספורילציה.
Introduction
זרחון חלבונים שולט ברוב התהליכים התאיים, כולל אך לא מוגבל לתגובה לנזק לדנ”א, איתות גורמי גדילה ומעבר דרך מיטוזה 1,2,3. בתאי יונקים, רוב החלבונים עוברים זרחון בשאריות סרין, תראונין או טירוזין אחד או יותר בנקודת זמן כלשהי, כאשר פוספוסרינה ופוספוטריאונינים מהווים כ-98% מכלל אתרי הזרחון 2,3. בעוד קינאזות נחקרו בהרחבה באיתות תאי, תפקידם של PPPs בוויסות תהליכים תאיים דינמיים עדיין מתפתח.
דינמיקת הזרחון נשלטת על ידי יחסי הגומלין הדינמיים בין קינאזות לפוספטזות. בתאי יונקים ישנם יותר מ-400 קינאזות חלבוניות המזרזות פוספורילציה של סרין/תראונין. יותר מ-90% מהאתרים האלה עוברים דה-פוספורילציה על-ידי פוספטאזות של פוספופרוטאין (PPPs), משפחה קטנה של אנזימים המורכבת מ-PP1, PP2A, PP2B, PP4-7, PPT ו-PPZ 2,3. PP1 ו-PP2A אחראים לרוב הפוספוסרין והפוספוטראונין דה-פוספורילציה בתוך תא 2,3,4. ההבדל הבולט במספר בין קינאזות לפוספטזות וחוסר הספציפיות של תת-יחידות קטליטיות PPP במבחנה הובילו לאמונה כי קינאזות הן הגורם העיקרי לזרחון 2,3. עם זאת, מחקרים רבים הראו כי פוספטזות מבססות את הספציפיות של המצע באמצעות היווצרות של הולואנזים רב-שמריים 5,6,7,8,9. לדוגמה, PP1 הוא הטרודימר המורכב מיחידת משנה קטליטית, ובזמן נתון, אחד מתוך יותר מ -150 תת-יחידות רגולטוריות 6,7,8. לעומת זאת, PP2A הוא הטרוטרימר שנוצר מפיגומים (A), יחידת משנה רגולטורית (B) ותת-יחידהקטליטית (C) 2,3,9. ישנן ארבע משפחות נפרדות של תת-יחידות רגולטוריות של PP2A (B55, B56, PR72 וסטריאטין), שלכל אחת מהן גנים מרובים, גרסאות שחבור ודפוסי לוקליזציה 2,3,9. האופי הרב-מהותי של PPPs ממלא את הפער במספר קינאזות ותת-יחידות קטליטיות של PPP. עם זאת, היא יוצרת אתגרים אנליטיים לחקר איתות PPP. כדי לנתח באופן מקיף איתות PPP, חשוב לחקור את ההולו-אנזים השונים בתוך תא או רקמה. התקדמות גדולה נעשתה בחקר הקינום האנושי באמצעות שימוש בחרוזי מעכבי קינאז, המכונים חרוזי מעכבי מולטיפלקס או קינוביאדות, אסטרטגיה פרוטאומית כימית שבה מעכבי קינאז משותקים על חרוזים וספקטרומטריית מסה משמשת לזיהוי קינאזות מועשרות והאינטראקצים שלהם 10,11,12,13.
ביססנו גישה דומה לחקר הביולוגיה של PPP. טכניקה זו כוללת לכידת זיקה של תת-יחידות קטליטיות PPP באמצעות חרוזים עם מעכב PPP משותק ולא סלקטיבי הנקרא מיקרוציסטין-LR (MCLR) המכונה חרוזי מעכבי פוספטאז (PIBs)14,15. בניגוד לשיטות אחרות הדורשות תיוג אנדוגני או ביטוי של תת-יחידות PPP אקסוגניות שיכולות לשנות את פעילות החלבון או לוקליזציה שלו, PIB-MS מאפשר העשרה של תת-יחידות קטליטיות אנדוגניות של PPP, תת-יחידות רגולטוריות ופיגומים הקשורות אליהן, וחלבונים מתקשרים (המכונים PPPome) מתאים ורקמות בנקודת זמן נתונה או בתנאי טיפול ספציפיים. MCLR מעכב PP1, PP2A, PP4-6, PPT ו-PPZ בריכוזי ננו-מולארים, מה שהופך את ה-PIBs ליעילים ביותר בהעשרת PPPome16. ניתן לשנות את קנה המידה של שיטה זו לשימוש בכל חומר מוצא, מתאים ועד דגימות קליניות. כאן נתאר בפירוט את השימוש ב-PIBs ובספקטרומטריית מסות (PIB-MS) כדי ללכוד, לזהות ולכמת ביעילות את ה-PPPome האנדוגני ואת מצבי השינוי שלו.
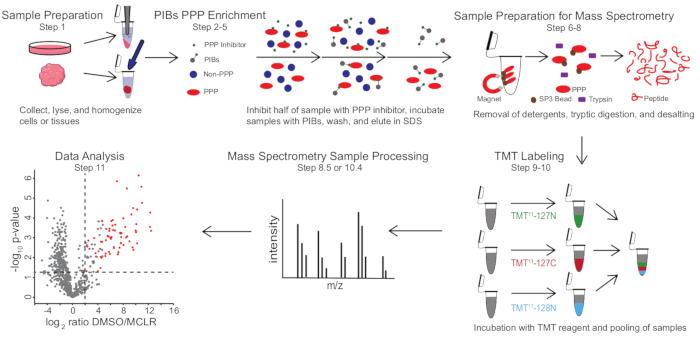
איור 1: סיכום חזותי של פרוטוקול PIB-MS. בניסוי PIB-MS ניתן לקבל דגימות בצורות שונות, מתאים ועד גידולים. הדגימה נאספת, משוקרת ומוגזמת לפני העשרת PPP. כדי להעשיר עבור PPPs, lysate הוא דגירה עם PIBs עם או בלי מעכב PPP, כגון MCLR. לאחר מכן נשטפים ה-PIBs, ו-PPPs נשטפים בתנאי דנטורציה. הדגימות מוכנות לניתוח ספקטרומטריית מסה על ידי הסרת חומרי ניקוי באמצעות העשרת חלבוני SP3, עיכול טריפטי והתפלה. לאחר מכן ניתן לתייג דגימות באופן אופציונלי TMT לפני ניתוח ספקטרומטריית מסות. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
PIB-MS כולל ליזה והבהרה של תאים או רקמות, דגירה של הליזאט עם PIBs, אלוטיון וניתוח של האלואט באמצעות כתמים מערביים או גישות מבוססות ספקטרומטריית מסה (איור 1). התוספת של MCLR חופשי יכולה לשמש כבקרה כדי להבחין בין קלסרים ספציפיים של PIB לבין אינטראקציות לא ספציפיות. עבור רוב היישומים, ניתן להשתמש בגישה נטולת תוויות כדי לזהות ישירות חלבונים ב- eluates. במקרים שבהם יש צורך בדיוק רב יותר בכימות או בזיהוי של מינים בעלי שפע נמוך, ניתן להשתמש בעיבוד נוסף עם תיוג תג מסה טנדם (TMT) כדי להגדיל את הכיסוי ולהפחית את הקלט.
Protocol
Representative Results
Discussion
PIB-MS היא גישה של פרוטאומיקה כימית המשמשת לפרופיל כמותי של ה-PPPome ממקורות דגימה שונים באנליזה אחת. עבודה רבה נעשתה באמצעות חרוזי מעכבי קינאז כדי לחקור את הקינום וכיצד הוא משתנה בסרטן ובמצבי מחלה אחרים 10,11,12,13. עם זאת, המחקר …
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
A.N.K. מכיר בתמיכה של NIH R33 CA225458 ו- R35 GM119455. אנו מודים למעבדות קטנבאך וגרבר על הדיון המועיל.
Materials
| Acetonitrile (ACN) | Honeywell | AH015-4 | CAUTION: ACN is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Anhydrous Acetonitrile | Sigma-Aldrich | 271004-100ML | CAUTION: ACN is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Benchtop centrifuge | Eppendorf | model no. 5424 | |
| Beta-glycerophosphoric acid, disodium salt pentahydrate | Acros Organics | 410991000 | |
| Centrifuge | Eppendorf | model no. 5810 R 15 amp version | |
| Distilled water | |||
| DMSO | Fisher Scientific | BP231-100 | |
| Dounce tissue grinder | Fisherbrand Pellet Pestles | 12-141-363 | |
| Empore solid phase extraction disk, C18 | CDS Analytical | 76333-132 | |
| Eppendorf tubes, 1.5 mL | Eppendorf | 22363204 | CRITICAL: Other tubes may leach polymer into sample, contaminating the analysis. |
| Eppendorf tubes, 2 mL | Eppendorf | 22363352 | CRITICAL: Other tubes may leach polymer into sample, contaminating the analysis. |
| Extraction plate manifold | Waters | WAT097944 | |
| Falcon tubes, 50 mL | VWR | 21008 | |
| Generic blunt end needle and plunger | |||
| Generic magnetic separation rack | |||
| HEPES | Sigma-Aldrich | H3375 | |
| Hydrogen chloride (HCl) | VWR Chemicals BDH | BDH3028 | CAUTION: HCl is corrosive; wear gloves and work in a chemical fume hood. |
| Hydroxylamine solution 50% (wt/vol) | Sigma-Aldrich | 467804 | |
| Incubator, 65 °C | VWR | model no. 1380FM | |
| Koptec Pure Ethanol, 200 Proof | Decon Labs | V1001 | |
| Methanol for HPLC (MeOH) | Sigma-Aldrich | 34860-4L-R | CAUTION: MeOH is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Microcystin LR (MCLR) | Cayman Chemical | 10007188 | CAUTION: MCLR is toxic; wear gloves when handling and avoid skin contact. |
| PBS, 1× without calcium and magnesium, pH 7.4 ± 0.1 | Corning | 21-040-CV | |
| pH test strips, such as MilliporeSigma MColorpHast pH test strips and indicator papers | Fisher Scientific | M1095310001 | |
| PIBs | For protocol for the generation of PIBs, see Moorhead et al., 2007. | ||
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23225 | |
| Pipette tips, 10 μL | Eppendorf | 22491504 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| Pipette tips, 1000 μL | Eppendorf | 22491555 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| Pipette tips, 200 μL | Eppendorf | 22491539 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| plastic syringe, 10 mL | BD | 309604 | |
| Protease inhibitor cocktail III | Research Products International | P50700-1 | |
| Q Exactive Plus Hybrid Quadrupole-Orbitrap Mass Spectrometer, Oribtrap Fusion, Orbitrap Fusion Lumos, or Orbitrap Eclipse Tribrid Mass Spectrometer | Thermo Scientific | ||
| Refrigerated benchtop centrifuge | Eppendorf | model no. 5424 R | |
| Rotator (Labquake Shaker Rotisserie) | Thermo Scientific | 13-687-12Q | 8 rpm rotation |
| Sample collection plate, 96- well, 1 mL | Waters | WAT058957 | |
| SDS | Fisher Scientific | BP1311-1 | |
| Sequencing grade modified trypsin | Promega | V511C | |
| Sodium azide | EMD Chemicals | SX0299-1 | CAUTION: Sodium azide is explosive and toxic; wear gloves, work in a chemical fume hood and avoid contact with metals. |
| Sodium chloride (NaCl) | Fisher Chemical | S27110 | |
| Sonicator (Branson digital sonifier) | model no. SFX 250 | ||
| SPE C18 desalting plate | Waters | 186001828BA | |
| SpeedBeads magnetic carboxylate modified particles (SP3 beads) | Cytiva | 6.51521E+13 | |
| Thermomixer | Eppendorf | model no. 5350 | |
| TMT10plex Isobaric Label Reagent Set plus TMT11-131C Label Reagent, 3 × 0.8 mg per tag | ThermoFisher | A37725 | |
| Trifluoroacetic acid (TFA) | Honeywell | T6508-25ML | CAUTION: TFA is corrosive and will irritate skin on contact. Wear gloves and eye protection, and work in a chemical fume hood. |
| Tris Base | Research Products International | T60040 | |
| Triton X-100 | Sigma-Aldrich | T9284 | |
| Vacuum centrifuge and vapor trap | Thermo Scientific | model nos. SpeedVac SPD120 and RVT5105 | |
| Vortexer (Vortex-Genie 2) | Scientific Industries | ||
| Water LC-MS | Honeywell | LC365-4 |
Riferimenti
- Nilsson, J. Protein phosphatases in the regulation of mitosis. Journal of Cell Biology. 218 (2), 395-409 (2019).
- Brautigan, D. L. Protein Ser/Thr phosphatases–the ugly ducklings of cell signalling. The FEBS Journal. 280 (2), 324-345 (2013).
- Brautigan, D. L., Shenolikar, S. Protein serine/threonine phosphatases: keys to unlocking regulators and substrates. Annual Review of Biochemistry. 87, 921-964 (2018).
- Janssens, V., Goris, J. Protein phosphatase 2A: A highly regulated family of serine/threonine phosphatases implicated in cell growth and signalling. Biochemical Journal. 353, 417-439 (2001).
- Virshup, D. M., Shenolikar, S. From promiscuity to precision: protein phosphatases get a makeover. Molecular Cell. 33 (5), 537-545 (2009).
- Bollen, M., Peti, W., Ragusa, M. J., Beullens, M. The extended PP1 toolkit: designed to create specificity. Trends in Biochemical Sciences. 35 (8), 450-458 (2010).
- Qian, J., Winkler, C., Bollen, M. 4D-networking by mitotic phosphatases. Current Opinion in Cell Biology. 25 (6), 697-703 (2013).
- Heroes, E., et al. The PP1 binding code: a molecular-lego strategy that governs specificity. The FEBS Journal. 280 (2), 584-595 (2013).
- Eichhorn, P. J., Creyghton, M. P., Bernards, R. Protein phosphatase 2A regulatory subunits and cancer. Biochimica et Biophysica Acta. 1795 (1), 1-15 (2009).
- Bantscheff, M., et al. Quantitative chemical proteomics reveals mechanisms of action of clinical ABL kinase inhibitors. Nature Biotechnology. 25 (9), 1035-1044 (2007).
- Klaeger, S., et al. Chemical proteomics reveals ferrochelatase as a common off-target of kinase inhibitors. ACS Chemical Biology. 11 (5), 1245-1254 (2016).
- Duncan, J. S., et al. Dynamic reprogramming of the kinome in response to targeted MEK inhibition in triple-negative breast cancer. Cell. 149 (2), 307-321 (2012).
- Cooper, M. J., et al. Application of multiplexed kinase inhibitor beads to study kinome adaptations in drug-resistant leukemia. PLoS ONE. 8 (6), 66755 (2013).
- Lyons, S. P., et al. A quantitative chemical proteomic strategy for profiling phosphoprotein phosphatases from yeast to humans. Molecular and Cellular Proteomics. 17 (12), 2448-2461 (2018).
- Nasa, I., et al. Quantitative kinase and phosphatase profiling reveal that CDK1 phosphorylates PP2Ac to promote mitotic entry. Science Signaling. 13 (648), (2020).
- Swingle, M., Ni, L., Honkanen, R. E. Small-molecule inhibitors of ser/thr protein phosphatases: specificity, use and common forms of abuse. Methods in Molecular Biology. 365, 23-38 (2007).
- Moorhead, G. B. G., Haystead, T. A. J., MacKintosh, C. Synthesis and use of the protein phosphatase affinity matrices microcystin-sepharose and microcystin-biotin-sepharose. Methods in Molecular Biology. 365, 39-45 (2007).
- Brauer, B. L., Wiredu, K., Mitchell, S., Moorhead, G. B., Gerber, S. A., Kettenbach, A. N. Affinity-based profiling of endogenous phosphoprotein phosphatases by mass spectrometry. Nature Protocols. 16 (10), 4919-4943 (2021).
- Hughes, C. S., Moggridge, S., Müller, T., Sorensen, P. H., Morin, G. B., Krijgsveld, J. Single-pot, solid-phase-enhanced sample preparation for proteomics experiments. Nature Protocols. 14 (1), 68-85 (2019).
- Rappsilber, J., Mann, M., Ishihama, Y. Protocol for micro-purification, enrichment, pre-fractionation and storage of peptides for proteomics using StageTips. Nature Protocols. 2 (8), 1896-1906 (2007).
- Zecha, J., et al. TMT labeling for the masses: A robust and cost-efficient, in-solution labeling approach. Molecular and Cellular Proteomics. 18 (7), 1468-1478 (2019).
- Elias, J. E., Gygi, S. P. Target-decoy search strategy for increased confidence in large-scale protein identifications by mass spectrometry. Nature Methods. 4 (3), 207-214 (2007).
- Eng, J. K., Jahan, T. A., Hoopmann, M. R. Comet: an open-source MS/MS sequence database search tool. Proteomics. 13 (1), 22-24 (2013).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nature Methods. 13 (9), 731-740 (2016).
- Yu, S. H., Ferretti, D., Schessner, J. P., Rudolph, J. D., Borner, G. H. H., Cox, J. Expanding the Perseus software for omics data analysis With custom plugins. Current Protocols in Bioinformatics. 71 (1), 1-29 (2020).

