फॉस्फोप्रोटीन फॉस्फेटेस और उनके इंटरैक्टर्स की पहचान करने के लिए एक मास स्पेक्ट्रोमेट्री-आधारित दृष्टिकोण
Summary
यहां, हम अंतर्जात फॉस्फोप्रोटीन फॉस्फेटेस के संवर्धन और कोशिकाओं और ऊतकों से उनके इंटरैक्टिंग प्रोटीन और मास स्पेक्ट्रोमेट्री-आधारित प्रोटिओमिक्स द्वारा उनकी पहचान और मात्रा का ठहराव के लिए एक प्रोटोकॉल प्रस्तुत करते हैं।
Abstract
अधिकांश सेलुलर प्रक्रियाओं को गतिशील प्रोटीन फॉस्फोराइलेशन द्वारा विनियमित किया जाता है। तीन-चौथाई से अधिक प्रोटीन फॉस्फोराइलेटेड होते हैं, और फॉस्फोप्रोटीन फॉस्फेट (पीपीपी) सभी सेलुलर सेरीन / थ्रेओनीन डिफॉस्फोराइलेशन के 90% से अधिक समन्वय करते हैं। प्रोटीन फॉस्फोराइलेशन के विनियमन को कैंसर और न्यूरोडीजेनेरेशन सहित विभिन्न बीमारियों के पैथोफिजियोलॉजी में फंसाया गया है। उनकी व्यापक गतिविधि के बावजूद, पीपीपी को नियंत्रित करने वाले आणविक तंत्र और पीपीपी द्वारा नियंत्रित लोगों की खराब विशेषता है। यहां, फॉस्फेट इनहिबिटर मोती और मास स्पेक्ट्रोमेट्री (पीआईबी-एमएस) नामक एक प्रोटिओमिक दृष्टिकोण को पीपीपी, उनके पोस्टट्रांसलेशनल संशोधनों और उनके इंटरैक्टर्स को किसी भी सेल लाइन या ऊतक का उपयोग करके 12 घंटे तक कम से कम पहचानने और मापने के लिए वर्णित किया गया है। पीआईबी-एमएस एक गैर-चयनात्मक पीपीपी अवरोधक, माइक्रोसिस्टिन-एलआर (एमसीएलआर) का उपयोग करता है, जो अंतर्जात पीपीपी और उनके संबंधित प्रोटीन (जिसे पीपीपीओम कहा जाता है) को पकड़ने और समृद्ध करने के लिए सेफरोज मोतियों पर स्थिर किया जाता है। इस विधि को पीपीपी के टैग किए गए संस्करणों की बहिर्जात अभिव्यक्ति या विशिष्ट एंटीबॉडी के उपयोग की आवश्यकता नहीं है। पीआईबी-एमएस विकासवादी रूप से संरक्षित पीपीपी का अध्ययन करने और डिफॉस्फोराइलेशन सिग्नलिंग की हमारी वर्तमान समझ का विस्तार करने का एक अभिनव तरीका प्रदान करता है।
Introduction
प्रोटीन फॉस्फोराइलेशन अधिकांश सेलुलर प्रक्रियाओं को नियंत्रित करता है, जिसमें डीएनए क्षति, विकास कारक सिग्नलिंग और माइटोसिस 1,2,3 के माध्यम से पारित होने की प्रतिक्रिया तक सीमित नहीं है। स्तनधारी कोशिकाओं में, अधिकांश प्रोटीन किसी समय एक या एक से अधिक सेरीन, थ्रेओनिन या टायरोसिन अवशेषों पर फॉस्फोराइलेटेड होते हैं, फॉस्फोसेरिन और फॉस्फोथ्रोनिन में सभी फॉस्फोराइलेशन साइटों का लगभग 98%शामिल होता है 2,3। जबकि सेलुलर सिग्नलिंग में किनेसेस का बड़े पैमाने पर अध्ययन किया गया है, गतिशील सेलुलर प्रक्रियाओं के विनियमन में पीपीपी की भूमिका अभी भी उभर रही है।
फॉस्फोराइलेशन गतिशीलता को किनेसेस और फॉस्फेटेस के बीच गतिशील परस्पर क्रिया द्वारा नियंत्रित किया जाता है। स्तनधारी कोशिकाओं में, 400 से अधिक प्रोटीन किनेसेस होते हैं जो सेरीन / थ्रेओनिन फॉस्फोराइलेशन को उत्प्रेरित करते हैं। इनमें से 90% से अधिक साइटें फॉस्फोप्रोटीन फॉस्फेटेस (पीपीपी) द्वारा डिफॉस्फोराइलेटेड हैं, एंजाइमों का एक छोटा सा परिवार जिसमें पीपी 1, पीपी 2 ए, पीपी 2 बी, पीपी 4-7, पीपीटी और पीपीजेड 2,3 शामिल हैं। पीपी 1 और पीपी 2 ए सेल 2,3,4 के भीतर फॉस्फोसेरिन और फॉस्फोथ्रोनिन डिफॉस्फोराइलेशन के बहुमत के लिए जिम्मेदार हैं। किनेसेस और फॉस्फेटेस के बीच संख्या में उल्लेखनीय अंतर और इन विट्रो में पीपीपी उत्प्रेरक सबयूनिट्स की विशिष्टता की कमी ने इस विश्वास को जन्म दिया कि किनेसेस फॉस्फोराइलेशन 2,3 के मुख्य निर्धारक हैं। हालांकि, कई अध्ययनों ने मल्टीमेरिक होलोनजाइम्स 5,6,7,8,9 के गठन के माध्यम से सब्सट्रेट विशिष्टता स्थापित करने के लिए फॉस्फेटेस दिखाए हैं। उदाहरण के लिए, पीपी 1 एक हेटेरोडिमर है जिसमें एक उत्प्रेरक सबयूनिट होता है और, एक निश्चित समय पर, 150 से अधिक नियामक सबयूनिट्स में से एक 6,7,8 होता है। इसके विपरीत, पीपी 2 ए एक हेटरोट्रिमर है जो एक मचान (ए), एक नियामक (बी), और एक उत्प्रेरक (सी) सबयूनिट 2,3,9 से बना है। पीपी 2 ए नियामक सबयूनिट्स (बी 55, बी 56, पीआर 72, और स्ट्रेटिन) के चार अलग-अलग परिवार हैं, जिनमें से प्रत्येक में कई जीन, स्प्लिस वेरिएंट और स्थानीयकरण पैटर्न 2,3,9 हैं। पीपीपी की बहुआयामी प्रकृति किनेसेस और पीपीपी उत्प्रेरक सबयूनिट्स की संख्या में अंतर को भरती है। हालांकि, यह पीपीपी सिग्नलिंग का अध्ययन करने के लिए विश्लेषणात्मक चुनौतियां पैदा करता है। पीपीपी सिग्नलिंग का व्यापक रूप से विश्लेषण करने के लिए, एक सेल या ऊतक के भीतर विभिन्न होलोएंजाइम की जांच करना महत्वपूर्ण है। किनेज अवरोधक मोतियों के उपयोग के माध्यम से मानव किनोम का अध्ययन करने में महान प्रगति हुई है, जिसे मल्टीप्लेक्स अवरोधक मोती या किनोबीड्स कहा जाता है, एक रासायनिक प्रोटिओमिक रणनीति जहां किनेज अवरोधकों को मोतियों पर स्थिर किया जाता है और मास स्पेक्ट्रोमेट्री का उपयोग समृद्ध किनेसेस और उनके इंटरैक्टर्स 10,11,12,13 की पहचान करने के लिए किया जाता है।
हमने पीपीपी जीव विज्ञान का अध्ययन करने के लिए एक समान दृष्टिकोण स्थापित किया है। इस तकनीक में माइक्रोसिस्टिन-एलआर (एमसीएलआर) नामक एक स्थिर, गैर-चयनात्मक पीपीपी अवरोधक के साथ मोतियों का उपयोग करके पीपीपी उत्प्रेरक सबयूनिट्स का आत्मीयता कैप्चर शामिल है जिसे फॉस्फेट इनहिबिटर मोती (पीआईबी) 14,15 कहा जाता है। अन्य तरीकों के विपरीत जिनके लिए अंतर्जात टैगिंग या बहिर्जात पीपीपी सबयूनिट्स की अभिव्यक्ति की आवश्यकता होती है जो प्रोटीन गतिविधि या स्थानीयकरण को बदल सकते हैं, पीआईबी-एमएस अंतर्जात पीपीपी उत्प्रेरक सबयूनिट्स, उनके संबंधित नियामक और मचान सबयूनिट्स, और किसी दिए गए समय बिंदु पर या विशिष्ट उपचार स्थितियों के तहत कोशिकाओं और ऊतकों से इंटरैक्टिंग प्रोटीन (जिसे पीपीपीओम कहा जाता है) के संवर्धन की अनुमति देता है। एमसीएलआर नैनोमोलर सांद्रता में पीपी 1, पीपी 2 ए, पीपी 4-6, पीपीटी और पीपीजेड को रोकता है, जिससे पीपीपीओएम16 के लिए समृद्ध करने में पीआईबी अत्यधिक प्रभावी हो जाता है। इस विधि को कोशिकाओं से नैदानिक नमूनों तक किसी भी प्रारंभिक सामग्री पर उपयोग के लिए स्केल किया जा सकता है। यहां, हम अंतर्जात पीपीपीओम और इसके संशोधन राज्यों को कुशलतापूर्वक पकड़ने, पहचानने और मात्रा निर्धारित करने के लिए पीआईबी और मास स्पेक्ट्रोमेट्री (पीआईबी-एमएस) के उपयोग का विस्तार से वर्णन करते हैं।
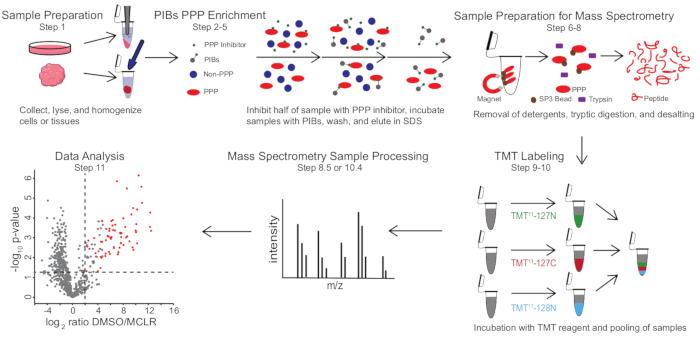
चित्रा 1: पीआईबी-एमएस प्रोटोकॉल का दृश्य सारांश। पीआईबी-एमएस प्रयोग में, नमूने कोशिकाओं से ट्यूमर तक विभिन्न रूपों में प्राप्त किए जा सकते हैं। नमूना पीपीपी संवर्धन से पहले एकत्र, लाइज्ड और होमोजेनाइज्ड है। पीपीपी के लिए समृद्ध करने के लिए, लाइसेट को पीपीपी-अवरोधक के साथ या बिना पीआईबी के साथ ऊष्मायन किया जाता है, जैसे कि एमसीएलआर। पीआईबी को तब धोया जाता है, और पीपीपी को विकृत करने की स्थिति में एल्यूट किया जाता है। नमूने एसपी 3 प्रोटीन संवर्धन, ट्रिप्टिक पाचन और डीसाल्टिंग के माध्यम से डिटर्जेंट को हटाकर मास स्पेक्ट्रोमेट्री विश्लेषण के लिए तैयार किए जाते हैं। नमूने तब मास स्पेक्ट्रोमेट्री विश्लेषण से पहले वैकल्पिक रूप से टीएमटी-लेबल किए जा सकते हैं। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें।
पीआईबी-एमएस में पश्चिमी सोख्ता या मास स्पेक्ट्रोमेट्री-आधारित दृष्टिकोण (चित्रा 1) के माध्यम से पीआईबी, क्षालन और एल्यूएट के विश्लेषण के साथ लाइसेट के इनक्यूबेशन कोशिकाओं या ऊतकों का लिसिस और स्पष्टीकरण शामिल है। मुफ्त एमसीएलआर के अलावा गैर-विशिष्ट इंटरएक्टर्स से विशिष्ट पीआईबी बाइंडर्स को अलग करने के लिए एक नियंत्रण के रूप में इस्तेमाल किया जा सकता है। अधिकांश अनुप्रयोगों के लिए, एक लेबल मुक्त दृष्टिकोण का उपयोग सीधे एलुएट्स में प्रोटीन की पहचान करने के लिए किया जा सकता है। ऐसे मामलों में जहां परिमाणीकरण में अधिक परिशुद्धता या कम बहुतायत वाली प्रजातियों की पहचान की आवश्यकता होती है, अग्रानुक्रम द्रव्यमान-टैग (टीएमटी) लेबलिंग के साथ आगे की प्रक्रिया का उपयोग कवरेज बढ़ाने और इनपुट को कम करने के लिए किया जा सकता है।
Protocol
Representative Results
Discussion
पीआईबी-एमएस एक रासायनिक प्रोटिओमिक्स दृष्टिकोण है जिसका उपयोग एक ही विश्लेषण में विभिन्न नमूना स्रोतों से पीपीपीओम को मात्रात्मक रूप से प्रोफाइल करने के लिए किया जाता है। किनोम का अध्ययन करने के लिए…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
एएनके एनआईएच आर 33 सीए 225458 और आर 35 जीएम 11 9 455 से समर्थन स्वीकार करता है। हम उनकी उपयोगी चर्चा के लिए केटेनबैक और गेरबर प्रयोगशालाओं को धन्यवाद देते हैं।
Materials
| Acetonitrile (ACN) | Honeywell | AH015-4 | CAUTION: ACN is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Anhydrous Acetonitrile | Sigma-Aldrich | 271004-100ML | CAUTION: ACN is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Benchtop centrifuge | Eppendorf | model no. 5424 | |
| Beta-glycerophosphoric acid, disodium salt pentahydrate | Acros Organics | 410991000 | |
| Centrifuge | Eppendorf | model no. 5810 R 15 amp version | |
| Distilled water | |||
| DMSO | Fisher Scientific | BP231-100 | |
| Dounce tissue grinder | Fisherbrand Pellet Pestles | 12-141-363 | |
| Empore solid phase extraction disk, C18 | CDS Analytical | 76333-132 | |
| Eppendorf tubes, 1.5 mL | Eppendorf | 22363204 | CRITICAL: Other tubes may leach polymer into sample, contaminating the analysis. |
| Eppendorf tubes, 2 mL | Eppendorf | 22363352 | CRITICAL: Other tubes may leach polymer into sample, contaminating the analysis. |
| Extraction plate manifold | Waters | WAT097944 | |
| Falcon tubes, 50 mL | VWR | 21008 | |
| Generic blunt end needle and plunger | |||
| Generic magnetic separation rack | |||
| HEPES | Sigma-Aldrich | H3375 | |
| Hydrogen chloride (HCl) | VWR Chemicals BDH | BDH3028 | CAUTION: HCl is corrosive; wear gloves and work in a chemical fume hood. |
| Hydroxylamine solution 50% (wt/vol) | Sigma-Aldrich | 467804 | |
| Incubator, 65 °C | VWR | model no. 1380FM | |
| Koptec Pure Ethanol, 200 Proof | Decon Labs | V1001 | |
| Methanol for HPLC (MeOH) | Sigma-Aldrich | 34860-4L-R | CAUTION: MeOH is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Microcystin LR (MCLR) | Cayman Chemical | 10007188 | CAUTION: MCLR is toxic; wear gloves when handling and avoid skin contact. |
| PBS, 1× without calcium and magnesium, pH 7.4 ± 0.1 | Corning | 21-040-CV | |
| pH test strips, such as MilliporeSigma MColorpHast pH test strips and indicator papers | Fisher Scientific | M1095310001 | |
| PIBs | For protocol for the generation of PIBs, see Moorhead et al., 2007. | ||
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23225 | |
| Pipette tips, 10 μL | Eppendorf | 22491504 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| Pipette tips, 1000 μL | Eppendorf | 22491555 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| Pipette tips, 200 μL | Eppendorf | 22491539 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| plastic syringe, 10 mL | BD | 309604 | |
| Protease inhibitor cocktail III | Research Products International | P50700-1 | |
| Q Exactive Plus Hybrid Quadrupole-Orbitrap Mass Spectrometer, Oribtrap Fusion, Orbitrap Fusion Lumos, or Orbitrap Eclipse Tribrid Mass Spectrometer | Thermo Scientific | ||
| Refrigerated benchtop centrifuge | Eppendorf | model no. 5424 R | |
| Rotator (Labquake Shaker Rotisserie) | Thermo Scientific | 13-687-12Q | 8 rpm rotation |
| Sample collection plate, 96- well, 1 mL | Waters | WAT058957 | |
| SDS | Fisher Scientific | BP1311-1 | |
| Sequencing grade modified trypsin | Promega | V511C | |
| Sodium azide | EMD Chemicals | SX0299-1 | CAUTION: Sodium azide is explosive and toxic; wear gloves, work in a chemical fume hood and avoid contact with metals. |
| Sodium chloride (NaCl) | Fisher Chemical | S27110 | |
| Sonicator (Branson digital sonifier) | model no. SFX 250 | ||
| SPE C18 desalting plate | Waters | 186001828BA | |
| SpeedBeads magnetic carboxylate modified particles (SP3 beads) | Cytiva | 6.51521E+13 | |
| Thermomixer | Eppendorf | model no. 5350 | |
| TMT10plex Isobaric Label Reagent Set plus TMT11-131C Label Reagent, 3 × 0.8 mg per tag | ThermoFisher | A37725 | |
| Trifluoroacetic acid (TFA) | Honeywell | T6508-25ML | CAUTION: TFA is corrosive and will irritate skin on contact. Wear gloves and eye protection, and work in a chemical fume hood. |
| Tris Base | Research Products International | T60040 | |
| Triton X-100 | Sigma-Aldrich | T9284 | |
| Vacuum centrifuge and vapor trap | Thermo Scientific | model nos. SpeedVac SPD120 and RVT5105 | |
| Vortexer (Vortex-Genie 2) | Scientific Industries | ||
| Water LC-MS | Honeywell | LC365-4 |
Riferimenti
- Nilsson, J. Protein phosphatases in the regulation of mitosis. Journal of Cell Biology. 218 (2), 395-409 (2019).
- Brautigan, D. L. Protein Ser/Thr phosphatases–the ugly ducklings of cell signalling. The FEBS Journal. 280 (2), 324-345 (2013).
- Brautigan, D. L., Shenolikar, S. Protein serine/threonine phosphatases: keys to unlocking regulators and substrates. Annual Review of Biochemistry. 87, 921-964 (2018).
- Janssens, V., Goris, J. Protein phosphatase 2A: A highly regulated family of serine/threonine phosphatases implicated in cell growth and signalling. Biochemical Journal. 353, 417-439 (2001).
- Virshup, D. M., Shenolikar, S. From promiscuity to precision: protein phosphatases get a makeover. Molecular Cell. 33 (5), 537-545 (2009).
- Bollen, M., Peti, W., Ragusa, M. J., Beullens, M. The extended PP1 toolkit: designed to create specificity. Trends in Biochemical Sciences. 35 (8), 450-458 (2010).
- Qian, J., Winkler, C., Bollen, M. 4D-networking by mitotic phosphatases. Current Opinion in Cell Biology. 25 (6), 697-703 (2013).
- Heroes, E., et al. The PP1 binding code: a molecular-lego strategy that governs specificity. The FEBS Journal. 280 (2), 584-595 (2013).
- Eichhorn, P. J., Creyghton, M. P., Bernards, R. Protein phosphatase 2A regulatory subunits and cancer. Biochimica et Biophysica Acta. 1795 (1), 1-15 (2009).
- Bantscheff, M., et al. Quantitative chemical proteomics reveals mechanisms of action of clinical ABL kinase inhibitors. Nature Biotechnology. 25 (9), 1035-1044 (2007).
- Klaeger, S., et al. Chemical proteomics reveals ferrochelatase as a common off-target of kinase inhibitors. ACS Chemical Biology. 11 (5), 1245-1254 (2016).
- Duncan, J. S., et al. Dynamic reprogramming of the kinome in response to targeted MEK inhibition in triple-negative breast cancer. Cell. 149 (2), 307-321 (2012).
- Cooper, M. J., et al. Application of multiplexed kinase inhibitor beads to study kinome adaptations in drug-resistant leukemia. PLoS ONE. 8 (6), 66755 (2013).
- Lyons, S. P., et al. A quantitative chemical proteomic strategy for profiling phosphoprotein phosphatases from yeast to humans. Molecular and Cellular Proteomics. 17 (12), 2448-2461 (2018).
- Nasa, I., et al. Quantitative kinase and phosphatase profiling reveal that CDK1 phosphorylates PP2Ac to promote mitotic entry. Science Signaling. 13 (648), (2020).
- Swingle, M., Ni, L., Honkanen, R. E. Small-molecule inhibitors of ser/thr protein phosphatases: specificity, use and common forms of abuse. Methods in Molecular Biology. 365, 23-38 (2007).
- Moorhead, G. B. G., Haystead, T. A. J., MacKintosh, C. Synthesis and use of the protein phosphatase affinity matrices microcystin-sepharose and microcystin-biotin-sepharose. Methods in Molecular Biology. 365, 39-45 (2007).
- Brauer, B. L., Wiredu, K., Mitchell, S., Moorhead, G. B., Gerber, S. A., Kettenbach, A. N. Affinity-based profiling of endogenous phosphoprotein phosphatases by mass spectrometry. Nature Protocols. 16 (10), 4919-4943 (2021).
- Hughes, C. S., Moggridge, S., Müller, T., Sorensen, P. H., Morin, G. B., Krijgsveld, J. Single-pot, solid-phase-enhanced sample preparation for proteomics experiments. Nature Protocols. 14 (1), 68-85 (2019).
- Rappsilber, J., Mann, M., Ishihama, Y. Protocol for micro-purification, enrichment, pre-fractionation and storage of peptides for proteomics using StageTips. Nature Protocols. 2 (8), 1896-1906 (2007).
- Zecha, J., et al. TMT labeling for the masses: A robust and cost-efficient, in-solution labeling approach. Molecular and Cellular Proteomics. 18 (7), 1468-1478 (2019).
- Elias, J. E., Gygi, S. P. Target-decoy search strategy for increased confidence in large-scale protein identifications by mass spectrometry. Nature Methods. 4 (3), 207-214 (2007).
- Eng, J. K., Jahan, T. A., Hoopmann, M. R. Comet: an open-source MS/MS sequence database search tool. Proteomics. 13 (1), 22-24 (2013).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nature Methods. 13 (9), 731-740 (2016).
- Yu, S. H., Ferretti, D., Schessner, J. P., Rudolph, J. D., Borner, G. H. H., Cox, J. Expanding the Perseus software for omics data analysis With custom plugins. Current Protocols in Bioinformatics. 71 (1), 1-29 (2020).

