En massespektrometribasert tilnærming for å identifisere fosfoproteinfosfater og deres interagere
Summary
Her presenterer vi en protokoll for berikelse av endogene fosfoproteinfosfater og deres interagerende proteiner fra celler og vev og deres identifisering og kvantifisering ved massespektrometribasert proteomikk.
Abstract
De fleste cellulære prosesser reguleres av dynamisk proteinfosforylering. Mer enn tre fjerdedeler av proteiner er fosforylert, og fosfoproteinfosfater (PPPer) koordinerer over 90% av all cellulær serin / treonin defosforylering. Deregulering av proteinfosforylering har vært involvert i patofysiologien til ulike sykdommer, inkludert kreft og nevrodegenerasjon. Til tross for deres utbredte aktivitet er de molekylære mekanismene som kontrollerer PPPer og de som kontrolleres av PPPer dårlig preget. Her beskrives en proteomisk tilnærming kalt fosfatasehemmerperler og massespektrometri (PIB-MS) for å identifisere og kvantifisere PPPer, deres posttranslasjonelle modifikasjoner og deres interaktivitet i så lite som 12 timer ved hjelp av en hvilken som helst cellelinje eller vev. PIB-MS bruker en ikke-selektiv PPP-hemmer, mikrocystin-LR (MCLR), immobilisert på sepharoseperler for å fange og berike endogene PPPer og deres tilknyttede proteiner (kalt PPPome). Denne metoden krever ikke det eksogene uttrykket av taggede versjoner av PPPer eller bruk av spesifikke antistoffer. PIB-MS tilbyr en innovativ måte å studere de evolusjonært bevarte PPP-ene og utvide vår nåværende forståelse av defosforyleringssignalering.
Introduction
Proteinfosforylering kontrollerer de fleste cellulære prosesser, inkludert, men ikke begrenset til, responsen på DNA-skade, vekstfaktorsignalering og passasjen gjennom mitose 1,2,3. I pattedyrceller er flertallet av proteiner fosforylert ved en eller flere serin-, treon- eller tyrosinrester på et tidspunkt, med fosforeriner og fosforetoniner som består av omtrent 98% av alle fosforyleringssteder 2,3. Mens kinases har blitt grundig studert i cellulær signalering, vises fortsatt PPPs rolle i reguleringen av dynamiske cellulære prosesser.
Fosforyleringsdynamikken styres av det dynamiske samspillet mellom kinaser og fosfater. I pattedyrceller er det mer enn 400 proteinkinaser som katalyserer serin / treoninfosforylering. Over 90% av disse nettstedene er defosforylatert av fosfoproteinfosfater (PPPer), en liten familie av enzymer som består av PP1, PP2A, PP2B, PP4-7, PPT og PPZ 2,3. PP1 og PP2A er ansvarlige for de fleste fosfor og fosforondefosforylering i en celle 2,3,4. Den bemerkelsesverdige forskjellen i antall mellom kinases og fosfater og mangelen på spesifisitet av PPP katalytiske subenheter in vitro førte til troen på at kinases er den viktigste determinanten for fosforylering 2,3. Imidlertid har flere studier vist fosfater for å etablere substratspesifikkitet gjennom dannelsen av multimeriske holoenzymer 5,6,7,8,9. For eksempel er PP1 en heterodimer som består av en katalytisk underenhet og på et gitt tidspunkt en av de mer enn 150 regulatoriske underenhetene 6,7,8. På den annen side er PP2A en heterotrimer som er dannet av et stillas (A), en regulatorisk (B) og en katalytisk (C) underenhet 2,3,9. Det er fire forskjellige familier av PP2A regulatoriske underenheter (B55, B56, PR72 og striatin), hver med flere gener, skjøtevarianter og lokaliseringsmønstre 2,3,9. Den multimeriske naturen til PPPer fyller gapet i antall kinases og PPP katalytiske underenheter. Det skaper imidlertid analytiske utfordringer for å studere PPP-signalering. For å analysere PPP-signalering grundig, er det viktig å undersøke de forskjellige holoenzymene i en celle eller et vev. Store fremskritt har blitt gjort i å studere den menneskelige kinome gjennom bruk av kinase inhibitor perler, betegnet multiplex inhibitor perler eller kinobeads, en kjemisk proteomisk strategi der kinase inhibitors er immobilisert på perler og masse spektrometri brukes til å identifisere beriket kinases og deres interagere 10,11,12,13.
Vi har etablert en lignende tilnærming for å studere PPP-biologi. Denne teknikken innebærer affinitetsfangst av PPP-katalytiske underenheter ved hjelp av perler med en immobilisert, ikke-selektiv PPP-hemmer kalt microcystin-LR (MCLR) kalt fosfatasehemmerperler (PIB)14,15. I motsetning til andre metoder som krever endogen merking eller uttrykk for eksogene PPP-underenheter som kan endre proteinaktivitet eller lokalisering, tillater PIB-MS berikelse av endogene PPP-katalytiske underenheter, deres tilhørende regulatoriske og stillasunderenheter, og interagerende proteiner (kalt PPPome) fra celler og vev på et gitt tidspunkt eller under spesifikke behandlingsforhold. MCLR hemmer PP1, PP2A, PP4-6, PPT og PPZ ved nanomolarkonsentrasjoner, noe som gjør PIBer svært effektive til å berike for PPPome16. Denne metoden kan skaleres for bruk på ethvert startmateriale fra celler til kliniske prøver. Her beskriver vi i detalj bruken av PIBer og massespektrometri (PIB-MS) for effektivt å fange, identifisere og kvantifisere den endogene PPPome og dens modifikasjonstilstander.
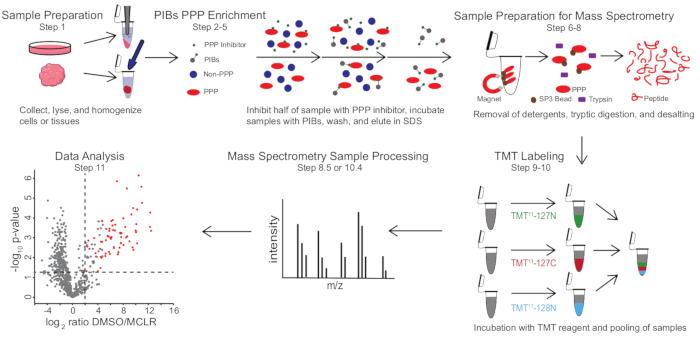
Figur 1: Visuelt sammendrag av PIB-MS-protokollen. I et PIB-MS-eksperiment kan prøver fås i forskjellige former, fra celler til svulster. Prøven samles inn, lyser og homogeniseres før PPP-berikelse. For å berike for PPPer inkuberes lysatet med PIBer med eller uten PPP-hemmer, for eksempel MCLR. PIB-ene vaskes deretter, og PPPer elutes under denatureringsforhold. Prøvene er utarbeidet for massespektrometrianalyse ved fjerning av vaskemidler gjennom SP3 proteinberikelse, tryptisk fordøyelse og avsalting. Prøver kan deretter eventuelt TMT-merket før massespektrometrianalyse. Klikk her for å se en større versjon av denne figuren.
PIB-MS innebærer lysis og avklaring av celler eller vev, inkubasjon av lysatet med PIBer, elution og analyse av eluate via vestlig blotting eller massespektrometribaserte tilnærminger (figur 1). Tillegg av gratis MCLR kan brukes som en kontroll for å skille spesifikke PIB-permer fra ikke-spesifikke interagere. For de fleste applikasjoner kan en etikettfri tilnærming brukes til å identifisere proteiner direkte i eluates. I tilfeller der større presisjon i kvantifisering eller identifisering av lav-overflod arter er nødvendig, kan videre behandling med tandem masse-tag (TMT) merking brukes til å øke dekningen og redusere inngang.
Protocol
Representative Results
Discussion
PIB-MS er en kjemisk proteomisk tilnærming som brukes til kvantitativ profilering av PPPome fra ulike prøvekilder i en enkelt analyse. Det er gjort mye arbeid med kinasehemmerperler for å studere kinome og hvordan det endrer seg i kreft og annen sykdom sier 10,11,12,13. Likevel henger studiet av PPPome etter. Vi forventer at denne tilnærmingen er i stand til å fylle dette gapet og belyse r…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
A.N.K. anerkjenner støtte fra NIH R33 CA225458 og R35 GM119455. Vi takker Kettenbach og Gerber labs for deres hjelpsomme diskusjon.
Materials
| Acetonitrile (ACN) | Honeywell | AH015-4 | CAUTION: ACN is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Anhydrous Acetonitrile | Sigma-Aldrich | 271004-100ML | CAUTION: ACN is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Benchtop centrifuge | Eppendorf | model no. 5424 | |
| Beta-glycerophosphoric acid, disodium salt pentahydrate | Acros Organics | 410991000 | |
| Centrifuge | Eppendorf | model no. 5810 R 15 amp version | |
| Distilled water | |||
| DMSO | Fisher Scientific | BP231-100 | |
| Dounce tissue grinder | Fisherbrand Pellet Pestles | 12-141-363 | |
| Empore solid phase extraction disk, C18 | CDS Analytical | 76333-132 | |
| Eppendorf tubes, 1.5 mL | Eppendorf | 22363204 | CRITICAL: Other tubes may leach polymer into sample, contaminating the analysis. |
| Eppendorf tubes, 2 mL | Eppendorf | 22363352 | CRITICAL: Other tubes may leach polymer into sample, contaminating the analysis. |
| Extraction plate manifold | Waters | WAT097944 | |
| Falcon tubes, 50 mL | VWR | 21008 | |
| Generic blunt end needle and plunger | |||
| Generic magnetic separation rack | |||
| HEPES | Sigma-Aldrich | H3375 | |
| Hydrogen chloride (HCl) | VWR Chemicals BDH | BDH3028 | CAUTION: HCl is corrosive; wear gloves and work in a chemical fume hood. |
| Hydroxylamine solution 50% (wt/vol) | Sigma-Aldrich | 467804 | |
| Incubator, 65 °C | VWR | model no. 1380FM | |
| Koptec Pure Ethanol, 200 Proof | Decon Labs | V1001 | |
| Methanol for HPLC (MeOH) | Sigma-Aldrich | 34860-4L-R | CAUTION: MeOH is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Microcystin LR (MCLR) | Cayman Chemical | 10007188 | CAUTION: MCLR is toxic; wear gloves when handling and avoid skin contact. |
| PBS, 1× without calcium and magnesium, pH 7.4 ± 0.1 | Corning | 21-040-CV | |
| pH test strips, such as MilliporeSigma MColorpHast pH test strips and indicator papers | Fisher Scientific | M1095310001 | |
| PIBs | For protocol for the generation of PIBs, see Moorhead et al., 2007. | ||
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23225 | |
| Pipette tips, 10 μL | Eppendorf | 22491504 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| Pipette tips, 1000 μL | Eppendorf | 22491555 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| Pipette tips, 200 μL | Eppendorf | 22491539 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| plastic syringe, 10 mL | BD | 309604 | |
| Protease inhibitor cocktail III | Research Products International | P50700-1 | |
| Q Exactive Plus Hybrid Quadrupole-Orbitrap Mass Spectrometer, Oribtrap Fusion, Orbitrap Fusion Lumos, or Orbitrap Eclipse Tribrid Mass Spectrometer | Thermo Scientific | ||
| Refrigerated benchtop centrifuge | Eppendorf | model no. 5424 R | |
| Rotator (Labquake Shaker Rotisserie) | Thermo Scientific | 13-687-12Q | 8 rpm rotation |
| Sample collection plate, 96- well, 1 mL | Waters | WAT058957 | |
| SDS | Fisher Scientific | BP1311-1 | |
| Sequencing grade modified trypsin | Promega | V511C | |
| Sodium azide | EMD Chemicals | SX0299-1 | CAUTION: Sodium azide is explosive and toxic; wear gloves, work in a chemical fume hood and avoid contact with metals. |
| Sodium chloride (NaCl) | Fisher Chemical | S27110 | |
| Sonicator (Branson digital sonifier) | model no. SFX 250 | ||
| SPE C18 desalting plate | Waters | 186001828BA | |
| SpeedBeads magnetic carboxylate modified particles (SP3 beads) | Cytiva | 6.51521E+13 | |
| Thermomixer | Eppendorf | model no. 5350 | |
| TMT10plex Isobaric Label Reagent Set plus TMT11-131C Label Reagent, 3 × 0.8 mg per tag | ThermoFisher | A37725 | |
| Trifluoroacetic acid (TFA) | Honeywell | T6508-25ML | CAUTION: TFA is corrosive and will irritate skin on contact. Wear gloves and eye protection, and work in a chemical fume hood. |
| Tris Base | Research Products International | T60040 | |
| Triton X-100 | Sigma-Aldrich | T9284 | |
| Vacuum centrifuge and vapor trap | Thermo Scientific | model nos. SpeedVac SPD120 and RVT5105 | |
| Vortexer (Vortex-Genie 2) | Scientific Industries | ||
| Water LC-MS | Honeywell | LC365-4 |
Riferimenti
- Nilsson, J. Protein phosphatases in the regulation of mitosis. Journal of Cell Biology. 218 (2), 395-409 (2019).
- Brautigan, D. L. Protein Ser/Thr phosphatases–the ugly ducklings of cell signalling. The FEBS Journal. 280 (2), 324-345 (2013).
- Brautigan, D. L., Shenolikar, S. Protein serine/threonine phosphatases: keys to unlocking regulators and substrates. Annual Review of Biochemistry. 87, 921-964 (2018).
- Janssens, V., Goris, J. Protein phosphatase 2A: A highly regulated family of serine/threonine phosphatases implicated in cell growth and signalling. Biochemical Journal. 353, 417-439 (2001).
- Virshup, D. M., Shenolikar, S. From promiscuity to precision: protein phosphatases get a makeover. Molecular Cell. 33 (5), 537-545 (2009).
- Bollen, M., Peti, W., Ragusa, M. J., Beullens, M. The extended PP1 toolkit: designed to create specificity. Trends in Biochemical Sciences. 35 (8), 450-458 (2010).
- Qian, J., Winkler, C., Bollen, M. 4D-networking by mitotic phosphatases. Current Opinion in Cell Biology. 25 (6), 697-703 (2013).
- Heroes, E., et al. The PP1 binding code: a molecular-lego strategy that governs specificity. The FEBS Journal. 280 (2), 584-595 (2013).
- Eichhorn, P. J., Creyghton, M. P., Bernards, R. Protein phosphatase 2A regulatory subunits and cancer. Biochimica et Biophysica Acta. 1795 (1), 1-15 (2009).
- Bantscheff, M., et al. Quantitative chemical proteomics reveals mechanisms of action of clinical ABL kinase inhibitors. Nature Biotechnology. 25 (9), 1035-1044 (2007).
- Klaeger, S., et al. Chemical proteomics reveals ferrochelatase as a common off-target of kinase inhibitors. ACS Chemical Biology. 11 (5), 1245-1254 (2016).
- Duncan, J. S., et al. Dynamic reprogramming of the kinome in response to targeted MEK inhibition in triple-negative breast cancer. Cell. 149 (2), 307-321 (2012).
- Cooper, M. J., et al. Application of multiplexed kinase inhibitor beads to study kinome adaptations in drug-resistant leukemia. PLoS ONE. 8 (6), 66755 (2013).
- Lyons, S. P., et al. A quantitative chemical proteomic strategy for profiling phosphoprotein phosphatases from yeast to humans. Molecular and Cellular Proteomics. 17 (12), 2448-2461 (2018).
- Nasa, I., et al. Quantitative kinase and phosphatase profiling reveal that CDK1 phosphorylates PP2Ac to promote mitotic entry. Science Signaling. 13 (648), (2020).
- Swingle, M., Ni, L., Honkanen, R. E. Small-molecule inhibitors of ser/thr protein phosphatases: specificity, use and common forms of abuse. Methods in Molecular Biology. 365, 23-38 (2007).
- Moorhead, G. B. G., Haystead, T. A. J., MacKintosh, C. Synthesis and use of the protein phosphatase affinity matrices microcystin-sepharose and microcystin-biotin-sepharose. Methods in Molecular Biology. 365, 39-45 (2007).
- Brauer, B. L., Wiredu, K., Mitchell, S., Moorhead, G. B., Gerber, S. A., Kettenbach, A. N. Affinity-based profiling of endogenous phosphoprotein phosphatases by mass spectrometry. Nature Protocols. 16 (10), 4919-4943 (2021).
- Hughes, C. S., Moggridge, S., Müller, T., Sorensen, P. H., Morin, G. B., Krijgsveld, J. Single-pot, solid-phase-enhanced sample preparation for proteomics experiments. Nature Protocols. 14 (1), 68-85 (2019).
- Rappsilber, J., Mann, M., Ishihama, Y. Protocol for micro-purification, enrichment, pre-fractionation and storage of peptides for proteomics using StageTips. Nature Protocols. 2 (8), 1896-1906 (2007).
- Zecha, J., et al. TMT labeling for the masses: A robust and cost-efficient, in-solution labeling approach. Molecular and Cellular Proteomics. 18 (7), 1468-1478 (2019).
- Elias, J. E., Gygi, S. P. Target-decoy search strategy for increased confidence in large-scale protein identifications by mass spectrometry. Nature Methods. 4 (3), 207-214 (2007).
- Eng, J. K., Jahan, T. A., Hoopmann, M. R. Comet: an open-source MS/MS sequence database search tool. Proteomics. 13 (1), 22-24 (2013).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nature Methods. 13 (9), 731-740 (2016).
- Yu, S. H., Ferretti, D., Schessner, J. P., Rudolph, J. D., Borner, G. H. H., Cox, J. Expanding the Perseus software for omics data analysis With custom plugins. Current Protocols in Bioinformatics. 71 (1), 1-29 (2020).

