Un enfoque basado en la espectrometría de masas para identificar fosfoproteínas fosfatasas y sus interactores
Summary
Aquí, presentamos un protocolo para el enriquecimiento de fosfoproteínas fosfatasas endógenas y sus proteínas que interactúan a partir de células y tejidos y su identificación y cuantificación mediante proteómica basada en espectrometría de masas.
Abstract
La mayoría de los procesos celulares están regulados por la fosforilación dinámica de proteínas. Más de tres cuartas partes de las proteínas están fosforiladas, y las fosfoproteínas fosfatasas (APP) coordinan más del 90% de toda la desfosforilación celular de serina/treonina. La desregulación de la fosforilación de proteínas se ha implicado en la fisiopatología de diversas enfermedades, incluyendo el cáncer y la neurodegeneración. A pesar de su actividad generalizada, los mecanismos moleculares que controlan las APP y los controlados por las APP están mal caracterizados. Aquí, se describe un enfoque proteómico denominado perlas inhibidoras de la fosfatasa y espectrometría de masas (PIB-MS) para identificar y cuantificar las APP, sus modificaciones posttraduccionales y sus interactores en tan solo 12 h utilizando cualquier línea celular o tejido. PIB-MS utiliza un inhibidor de PPP no selectivo, microcistina-LR (MCLR), inmovilizado en perlas de sefarosa para capturar y enriquecer ppp endógenos y sus proteínas asociadas (denominado PPPome). Este método no requiere la expresión exógena de versiones etiquetadas de PPP ni el uso de anticuerpos específicos. PIB-MS ofrece una forma innovadora de estudiar las APP conservadas evolutivamente y ampliar nuestra comprensión actual de la señalización de desfosforilación.
Introduction
La fosforilación de proteínas controla la mayoría de los procesos celulares, incluyendo pero no limitado a la respuesta al daño del ADN, la señalización del factor de crecimiento y el paso a través de la mitosis 1,2,3. En las células de mamíferos, la mayoría de las proteínas se fosforilan en uno o más residuos de serina, treonina o tirosina en algún momento, con fosfoserinas y fosfotreoninas que comprenden aproximadamente el 98% de todos los sitios de fosforilación 2,3. Si bien las quinasas se han estudiado ampliamente en la señalización celular, el papel de las APP en la regulación de los procesos celulares dinámicos aún está emergiendo.
La dinámica de fosforilación está controlada por la interacción dinámica entre las quinasas y las fosfatasas. En las células de mamíferos, hay más de 400 proteínas quinasas que catalizan la fosforilación de serina/treonina. Más del 90% de estos sitios están desfosforilados por fosfoproteínas fosfatasas (APP), una pequeña familia de enzimas que consiste en PP1, PP2A, PP2B, PP4-7, PPT y PPZ 2,3. PP1 y PP2A son responsables de la mayoría de la desfosforilación de fosfoserina y fosfotreonina dentro de una célula 2,3,4. La notable diferencia de número entre quinasas y fosfatasas y la falta de especificidad de las subunidades catalíticas de PPP in vitro llevaron a la creencia de que las quinasas son el principal determinante de la fosforilación 2,3. Sin embargo, múltiples estudios han demostrado que las fosfatasas establecen la especificidad del sustrato a través de la formación de holoenzimas multiméricas 5,6,7,8,9. Por ejemplo, PP1 es un heterodímero que consiste en una subunidad catalítica y, en un momento dado, una de las más de 150 subunidades reguladoras 6,7,8. Por el contrario, PP2A es un heterotrímero que está formado por un andamio (A), un regulador (B) y una subunidad catalítica (C) 2,3,9. Hay cuatro familias distintas de subunidades reguladoras de PP2A (B55, B56, PR72 y estriatina), cada una con múltiples genes, variantes de empalme y patrones de localización 2,3,9. La naturaleza multimérica de las APP llena el vacío en el número de quinasas y subunidades catalíticas de PPP. Sin embargo, crea desafíos analíticos para estudiar la señalización de PPP. Para analizar exhaustivamente la señalización de PPP, es fundamental investigar las diversas holoenzimas dentro de una célula o tejido. Se han logrado grandes avances en el estudio del quinasmo humano mediante el uso de perlas inhibidoras de la quinasa, denominadas perlas inhibidoras multiplex o quinabiadas, una estrategia proteómica química donde los inhibidores de la quinasa se inmovilizan en perlas y se utiliza la espectrometría de masas para identificar quinasas enriquecidas y sus interactores 10,11,12,13.
Hemos establecido un enfoque similar para estudiar la biología de la PPP. Esta técnica consiste en la captura por afinidad de subunidades catalíticas de PPP utilizando perlas con un inhibidor de PPP inmovilizado y no selectivo llamado microcistina-LR (MCLR) denominado perlas inhibidoras de la fosfatasa (PIBs)14,15. A diferencia de otros métodos que requieren el etiquetado endógeno o la expresión de subunidades exógenas de PPP que podrían alterar la actividad o localización de proteínas, PIB-MS permite el enriquecimiento de subunidades catalíticas endógenas de PPP, sus subunidades reguladoras y de andamiaje asociadas, y proteínas que interactúan (denominadas PPPome) de células y tejidos en un punto de tiempo dado o bajo condiciones de tratamiento específicas. MCLR inhibe PP1, PP2A, PP4-6, PPT y PPZ a concentraciones nanomolares, lo que hace que los PIB sean altamente efectivos para enriquecer el PPPome16. Este método se puede escalar para su uso en cualquier material de partida, desde células hasta muestras clínicas. Aquí, describimos en detalle el uso de PIBs y espectrometría de masas (PIB-MS) para capturar, identificar y cuantificar eficientemente el PPPome endógeno y sus estados de modificación.
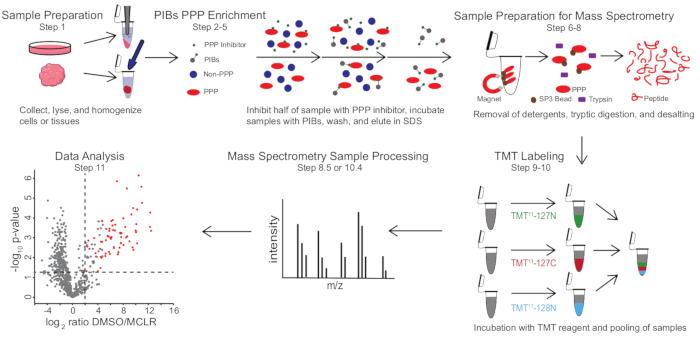
Figura 1: Resumen visual del protocolo PIB-MS. En un experimento PIB-MS, se pueden obtener muestras en varias formas, desde células hasta tumores. La muestra se recoge, se lisa y homogeneiza antes del enriquecimiento con PPP. Para enriquecer para las APP, el lisado se incuba con PIBs con o sin un inhibidor de PPP, como MCLR. Los PIB se lavan y las APP se eluyen en condiciones de desnaturalización. Las muestras se preparan para el análisis de espectrometría de masas mediante la eliminación de detergentes a través del enriquecimiento de proteínas SP3, la digestión tríptica y la desalinización. Las muestras pueden ser opcionalmente marcadas con TMT antes del análisis de espectrometría de masas. Haga clic aquí para ver una versión más grande de esta figura.
PIB-MS implica lisis y clarificación de células o tejidos, incubación del lisado con PIBs, elución y análisis del eluido a través de western blotting o enfoques basados en espectrometría de masas (Figura 1). La adición de MCLR libre se puede utilizar como un control para distinguir aglutinantes ESPECÍFICOS de PIB de interactores no específicos. Para la mayoría de las aplicaciones, se puede utilizar un enfoque sin etiquetas para identificar directamente las proteínas en los eluidos. En los casos en que se necesita una mayor precisión en la cuantificación o la identificación de especies de baja abundancia, se puede utilizar un procesamiento adicional con etiquetado de etiqueta de masa en tándem (TMT) para aumentar la cobertura y disminuir la entrada.
Protocol
Representative Results
Discussion
PIB-MS es un enfoque de proteómica química utilizado para perfilar cuantitativamente el PPPome de varias fuentes de muestra en un solo análisis. Se ha realizado mucho trabajo utilizando perlas inhibidoras de la quinasa para estudiar el quinasa y cómo cambia en el cáncer y otros estados de enfermedad 10,11,12,13. Sin embargo, el estudio del PPPome va a la zaga. Anticipamos que este enfoque …
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
A.N.K. reconoce el apoyo de NIH R33 CA225458 y R35 GM119455. Agradecemos a los laboratorios Kettenbach y Gerber por su útil discusión.
Materials
| Acetonitrile (ACN) | Honeywell | AH015-4 | CAUTION: ACN is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Anhydrous Acetonitrile | Sigma-Aldrich | 271004-100ML | CAUTION: ACN is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Benchtop centrifuge | Eppendorf | model no. 5424 | |
| Beta-glycerophosphoric acid, disodium salt pentahydrate | Acros Organics | 410991000 | |
| Centrifuge | Eppendorf | model no. 5810 R 15 amp version | |
| Distilled water | |||
| DMSO | Fisher Scientific | BP231-100 | |
| Dounce tissue grinder | Fisherbrand Pellet Pestles | 12-141-363 | |
| Empore solid phase extraction disk, C18 | CDS Analytical | 76333-132 | |
| Eppendorf tubes, 1.5 mL | Eppendorf | 22363204 | CRITICAL: Other tubes may leach polymer into sample, contaminating the analysis. |
| Eppendorf tubes, 2 mL | Eppendorf | 22363352 | CRITICAL: Other tubes may leach polymer into sample, contaminating the analysis. |
| Extraction plate manifold | Waters | WAT097944 | |
| Falcon tubes, 50 mL | VWR | 21008 | |
| Generic blunt end needle and plunger | |||
| Generic magnetic separation rack | |||
| HEPES | Sigma-Aldrich | H3375 | |
| Hydrogen chloride (HCl) | VWR Chemicals BDH | BDH3028 | CAUTION: HCl is corrosive; wear gloves and work in a chemical fume hood. |
| Hydroxylamine solution 50% (wt/vol) | Sigma-Aldrich | 467804 | |
| Incubator, 65 °C | VWR | model no. 1380FM | |
| Koptec Pure Ethanol, 200 Proof | Decon Labs | V1001 | |
| Methanol for HPLC (MeOH) | Sigma-Aldrich | 34860-4L-R | CAUTION: MeOH is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Microcystin LR (MCLR) | Cayman Chemical | 10007188 | CAUTION: MCLR is toxic; wear gloves when handling and avoid skin contact. |
| PBS, 1× without calcium and magnesium, pH 7.4 ± 0.1 | Corning | 21-040-CV | |
| pH test strips, such as MilliporeSigma MColorpHast pH test strips and indicator papers | Fisher Scientific | M1095310001 | |
| PIBs | For protocol for the generation of PIBs, see Moorhead et al., 2007. | ||
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23225 | |
| Pipette tips, 10 μL | Eppendorf | 22491504 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| Pipette tips, 1000 μL | Eppendorf | 22491555 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| Pipette tips, 200 μL | Eppendorf | 22491539 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| plastic syringe, 10 mL | BD | 309604 | |
| Protease inhibitor cocktail III | Research Products International | P50700-1 | |
| Q Exactive Plus Hybrid Quadrupole-Orbitrap Mass Spectrometer, Oribtrap Fusion, Orbitrap Fusion Lumos, or Orbitrap Eclipse Tribrid Mass Spectrometer | Thermo Scientific | ||
| Refrigerated benchtop centrifuge | Eppendorf | model no. 5424 R | |
| Rotator (Labquake Shaker Rotisserie) | Thermo Scientific | 13-687-12Q | 8 rpm rotation |
| Sample collection plate, 96- well, 1 mL | Waters | WAT058957 | |
| SDS | Fisher Scientific | BP1311-1 | |
| Sequencing grade modified trypsin | Promega | V511C | |
| Sodium azide | EMD Chemicals | SX0299-1 | CAUTION: Sodium azide is explosive and toxic; wear gloves, work in a chemical fume hood and avoid contact with metals. |
| Sodium chloride (NaCl) | Fisher Chemical | S27110 | |
| Sonicator (Branson digital sonifier) | model no. SFX 250 | ||
| SPE C18 desalting plate | Waters | 186001828BA | |
| SpeedBeads magnetic carboxylate modified particles (SP3 beads) | Cytiva | 6.51521E+13 | |
| Thermomixer | Eppendorf | model no. 5350 | |
| TMT10plex Isobaric Label Reagent Set plus TMT11-131C Label Reagent, 3 × 0.8 mg per tag | ThermoFisher | A37725 | |
| Trifluoroacetic acid (TFA) | Honeywell | T6508-25ML | CAUTION: TFA is corrosive and will irritate skin on contact. Wear gloves and eye protection, and work in a chemical fume hood. |
| Tris Base | Research Products International | T60040 | |
| Triton X-100 | Sigma-Aldrich | T9284 | |
| Vacuum centrifuge and vapor trap | Thermo Scientific | model nos. SpeedVac SPD120 and RVT5105 | |
| Vortexer (Vortex-Genie 2) | Scientific Industries | ||
| Water LC-MS | Honeywell | LC365-4 |
Riferimenti
- Nilsson, J. Protein phosphatases in the regulation of mitosis. Journal of Cell Biology. 218 (2), 395-409 (2019).
- Brautigan, D. L. Protein Ser/Thr phosphatases–the ugly ducklings of cell signalling. The FEBS Journal. 280 (2), 324-345 (2013).
- Brautigan, D. L., Shenolikar, S. Protein serine/threonine phosphatases: keys to unlocking regulators and substrates. Annual Review of Biochemistry. 87, 921-964 (2018).
- Janssens, V., Goris, J. Protein phosphatase 2A: A highly regulated family of serine/threonine phosphatases implicated in cell growth and signalling. Biochemical Journal. 353, 417-439 (2001).
- Virshup, D. M., Shenolikar, S. From promiscuity to precision: protein phosphatases get a makeover. Molecular Cell. 33 (5), 537-545 (2009).
- Bollen, M., Peti, W., Ragusa, M. J., Beullens, M. The extended PP1 toolkit: designed to create specificity. Trends in Biochemical Sciences. 35 (8), 450-458 (2010).
- Qian, J., Winkler, C., Bollen, M. 4D-networking by mitotic phosphatases. Current Opinion in Cell Biology. 25 (6), 697-703 (2013).
- Heroes, E., et al. The PP1 binding code: a molecular-lego strategy that governs specificity. The FEBS Journal. 280 (2), 584-595 (2013).
- Eichhorn, P. J., Creyghton, M. P., Bernards, R. Protein phosphatase 2A regulatory subunits and cancer. Biochimica et Biophysica Acta. 1795 (1), 1-15 (2009).
- Bantscheff, M., et al. Quantitative chemical proteomics reveals mechanisms of action of clinical ABL kinase inhibitors. Nature Biotechnology. 25 (9), 1035-1044 (2007).
- Klaeger, S., et al. Chemical proteomics reveals ferrochelatase as a common off-target of kinase inhibitors. ACS Chemical Biology. 11 (5), 1245-1254 (2016).
- Duncan, J. S., et al. Dynamic reprogramming of the kinome in response to targeted MEK inhibition in triple-negative breast cancer. Cell. 149 (2), 307-321 (2012).
- Cooper, M. J., et al. Application of multiplexed kinase inhibitor beads to study kinome adaptations in drug-resistant leukemia. PLoS ONE. 8 (6), 66755 (2013).
- Lyons, S. P., et al. A quantitative chemical proteomic strategy for profiling phosphoprotein phosphatases from yeast to humans. Molecular and Cellular Proteomics. 17 (12), 2448-2461 (2018).
- Nasa, I., et al. Quantitative kinase and phosphatase profiling reveal that CDK1 phosphorylates PP2Ac to promote mitotic entry. Science Signaling. 13 (648), (2020).
- Swingle, M., Ni, L., Honkanen, R. E. Small-molecule inhibitors of ser/thr protein phosphatases: specificity, use and common forms of abuse. Methods in Molecular Biology. 365, 23-38 (2007).
- Moorhead, G. B. G., Haystead, T. A. J., MacKintosh, C. Synthesis and use of the protein phosphatase affinity matrices microcystin-sepharose and microcystin-biotin-sepharose. Methods in Molecular Biology. 365, 39-45 (2007).
- Brauer, B. L., Wiredu, K., Mitchell, S., Moorhead, G. B., Gerber, S. A., Kettenbach, A. N. Affinity-based profiling of endogenous phosphoprotein phosphatases by mass spectrometry. Nature Protocols. 16 (10), 4919-4943 (2021).
- Hughes, C. S., Moggridge, S., Müller, T., Sorensen, P. H., Morin, G. B., Krijgsveld, J. Single-pot, solid-phase-enhanced sample preparation for proteomics experiments. Nature Protocols. 14 (1), 68-85 (2019).
- Rappsilber, J., Mann, M., Ishihama, Y. Protocol for micro-purification, enrichment, pre-fractionation and storage of peptides for proteomics using StageTips. Nature Protocols. 2 (8), 1896-1906 (2007).
- Zecha, J., et al. TMT labeling for the masses: A robust and cost-efficient, in-solution labeling approach. Molecular and Cellular Proteomics. 18 (7), 1468-1478 (2019).
- Elias, J. E., Gygi, S. P. Target-decoy search strategy for increased confidence in large-scale protein identifications by mass spectrometry. Nature Methods. 4 (3), 207-214 (2007).
- Eng, J. K., Jahan, T. A., Hoopmann, M. R. Comet: an open-source MS/MS sequence database search tool. Proteomics. 13 (1), 22-24 (2013).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nature Methods. 13 (9), 731-740 (2016).
- Yu, S. H., Ferretti, D., Schessner, J. P., Rudolph, J. D., Borner, G. H. H., Cox, J. Expanding the Perseus software for omics data analysis With custom plugins. Current Protocols in Bioinformatics. 71 (1), 1-29 (2020).

