Fosfoprotein fosfatazları ve bunların interaktörlerini tanımlamak için kütle spektrometrisine dayalı bir yaklaşım
Summary
Burada, endojen fosfoprotein fosfatazların ve bunların hücre ve dokulardan etkileşen proteinlerinin zenginleştirilmesi ve kütle spektrometrisine dayalı proteomiklerle tanımlanması ve nicelleştirilmesi için bir protokol sunuyoruz.
Abstract
Çoğu hücresel süreç dinamik protein fosforilasyonu ile düzenlenir. Proteinlerin dörtte üçünden fazlası fosforile edilir ve fosfoprotein fosfatazlar (PPP’ler) tüm hücresel serin / treonin defosforilasyonunun% 90’ından fazlasını koordine eder. Protein fosforilasyonunun serbestleştirilmesi, kanser ve nörodejenerasyon dahil olmak üzere çeşitli hastalıkların patofizyolojisinde rol oynamıştır. Yaygın aktivitelerine rağmen, PPP’leri kontrol eden moleküler mekanizmalar ve PPP’ler tarafından kontrol edilenler zayıf karakterizedir. Burada, fosfataz inhibitörü boncukları ve kütle spektrometresi (PIB-MS) olarak adlandırılan proteomik bir yaklaşım, PPP’leri, posttranslasyonel modifikasyonlarını ve interaktörlerini herhangi bir hücre hattı veya dokusunu kullanarak 12 saat kadar kısa bir sürede tanımlamak ve ölçmek için tanımlanmıştır. PIB-MS, endojen PPP’leri ve bunlarla ilişkili proteinleri (PPPome olarak adlandırılır) yakalamak ve zenginleştirmek için sefaroz boncukları üzerinde hareketsiz hale getirilmiş, seçici olmayan bir PPP inhibitörü olan mikrosistin-LR (MCLR) kullanır. Bu yöntem, PPP’lerin etiketli versiyonlarının eksojen ekspresyonunu veya spesifik antikorların kullanılmasını gerektirmez. PIB-MS, evrimsel olarak korunmuş PPP’leri incelemek ve mevcut defosforilasyon sinyalizasyonu anlayışımızı genişletmek için yenilikçi bir yol sunmaktadır.
Introduction
Protein fosforilasyonu, DNA hasarına verilen yanıt, büyüme faktörü sinyallemesi ve mitoz 1,2,3’ten geçiş dahil ancak bunlarla sınırlı olmamak üzere çoğu hücresel süreci kontrol eder. Memeli hücrelerinde, proteinlerin çoğunluğu bir veya daha fazla serin, treonin veya tirozin kalıntısında fosforile edilir, fosfoserlerin ve fosfotreoninler tüm fosforilasyon bölgelerinin yaklaşık% 98’ini oluşturur 2,3. Kinazlar hücresel sinyalizasyonda kapsamlı bir şekilde incelenmiş olsa da, PPP’lerin dinamik hücresel süreçlerin düzenlenmesindeki rolü hala ortaya çıkmaktadır.
Fosforilasyon dinamikleri, kinazlar ve fosfatazlar arasındaki dinamik etkileşim ile kontrol edilir. Memeli hücrelerinde, serin / treonin fosforilasyonunu katalize eden 400’den fazla protein kinaz vardır. Bu bölgelerin% 90’ından fazlası, PP1, PP2A, PP2B, PP4-7,PPT ve PPZ 2,3’ten oluşan küçük bir enzim ailesi olan fosfoprotein fosfatazlar (PPP’ler) tarafından defosforile edilir. PP1 ve PP2A, bir hücre 2,3,4 içindeki fosfoserin ve fosfotreonin defosforilasyonunun çoğundan sorumludur. Kinazlar ve fosfatazlar arasındaki kayda değer sayı farkı ve PPP katalitik alt birimlerinin in vitro özgüllüğünün olmaması, kinazların fosforilasyonun ana belirleyicisi olduğu inancına yol açmıştır 2,3. Bununla birlikte, birçok çalışma, fosfatazların multimerik holoenzimlerinoluşumu yoluyla substrat özgüllüğünü oluşturduğunu göstermiştir 5,6,7,8,9. Örneğin, PP1, katalitik bir alt birimden ve belirli bir zamanda, 150’den fazla düzenleyici alt birim 6,7,8’den birinden oluşan bir heterodimerdir. Tersine, PP2A, bir iskele (A), bir düzenleyici (B) ve bir katalitik (C) alt birimi 2,3,9’dan oluşan bir heterotrimerdir. PP2A düzenleyici alt birimlerinin (B55, B56, PR72 ve striatin) dört ayrı ailesi vardır, her biri birden fazla gen, ekleme varyantı ve lokalizasyon paternleri 2,3,9’dur. PPP’lerin multimerik doğası, kinazların ve PPP katalitik alt birimlerinin sayısındaki boşluğu doldurur. Bununla birlikte, PPP sinyallemesini incelemek için analitik zorluklar yaratır. PPP sinyalini kapsamlı bir şekilde analiz etmek için, bir hücre veya doku içindeki çeşitli holoenzimleri araştırmak çok önemlidir. İnsan kinomunun incelenmesinde, multipleks inhibitör boncuklar veya kinoboncuklar olarak adlandırılan kinaz inhibitörü boncukların kullanılmasında, kinaz inhibitörlerinin boncuklar üzerinde hareketsiz hale getirildiği ve zenginleştirilmiş kinazları ve bunların interaktörlerini tanımlamak için kütle spektrometrisinin kullanıldığı kimyasal bir proteomik strateji kullanılarak büyük ilerlemeler kaydedilmiştir10,11,12,13.
PPP biyolojisini incelemek için benzer bir yaklaşım oluşturduk. Bu teknik, fosfataz inhibitörü boncuklar (PIB’ler) olarak adlandırılan mikrosistin-LR (MCLR) adı verilen hareketsiz, seçici olmayan bir PPP inhibitörü ile boncuklar kullanılarak PPP katalitik alt birimlerinin afinite yakalamasını içerir14,15. Protein aktivitesini veya lokalizasyonunu değiştirebilecek eksojen PPP alt birimlerinin endojen etiketlemesini veya ekspresyonunu gerektiren diğer yöntemlerden farklı olarak, PIB-MS, endojen PPP katalitik alt birimlerinin, bunlarla ilişkili düzenleyici ve iskele alt birimlerinin ve belirli bir zaman noktasında veya belirli tedavi koşulları altında hücrelerden ve dokulardan etkileşime giren proteinlerin (PPPome olarak adlandırılır) zenginleştirilmesine izin verir. MCLR, nanomolar konsantrasyonlarda PP1, PP2A, PP4-6, PPT ve PPZ’yi inhibe ederek PIB’leri PPPome16 için zenginleştirmede oldukça etkili hale getirir. Bu yöntem, hücrelerden klinik örneklere kadar herhangi bir başlangıç materyalinde kullanılmak üzere ölçeklendirilebilir. Burada, endojen PPPome ve modifikasyon durumlarını verimli bir şekilde yakalamak, tanımlamak ve ölçmek için PIB’lerin ve kütle spektrometresinin (PIB-MS) kullanımını ayrıntılı olarak açıklıyoruz.
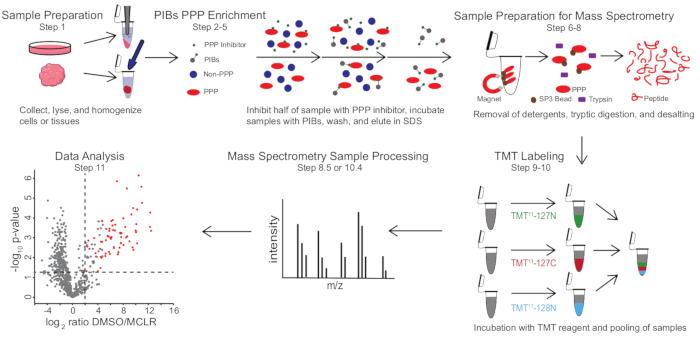
Şekil 1: PIB-MS protokolünün görsel özeti. Bir PIB-MS deneyinde, hücrelerden tümörlere kadar çeşitli şekillerde örnekler elde edilebilir. Numune, PPP zenginleştirmeden önce toplanır, lize edilir ve homojenize edilir. PPP’ler için zenginleştirmek için, lizat, MCLR gibi bir PPP inhibitörü olan veya olmayan PIB’lerle inkübe edilir. PIB’ler daha sonra yıkanır ve PPP’ler denatüre koşullarında salınır. Numuneler, SP3 protein zenginleştirme, triptik sindirim ve tuzdan arındırma yoluyla deterjanların uzaklaştırılması yoluyla kütle spektrometresi analizi için hazırlanır. Numuneler daha sonra kütle spektrometresi analizinden önce isteğe bağlı olarak TMT etiketli olabilir. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
PIB-MS, hücrelerin veya dokuların lizisini ve berraklaştırılmasını, lizatın PIB’lerle inkübasyonunu, elüsyonu ve batı lekelenmesi veya kütle spektrometrisine dayalı yaklaşımlarla elüatın analizini içerir (Şekil 1). Serbest MCLR’nin eklenmesi, spesifik PIB bağlayıcılarını spesifik olmayan interaktörlerden ayırt etmek için bir kontrol olarak kullanılabilir. Çoğu uygulama için, elüatlardaki proteinleri doğrudan tanımlamak için etiketsiz bir yaklaşım kullanılabilir. Nicelemede veya düşük bolluktaki türlerin tanımlanmasında daha fazla hassasiyetin gerekli olduğu durumlarda, kapsama alanını artırmak ve girişi azaltmak için tandem kütle etiketi (TMT) etiketleme ile daha fazla işlem kullanılabilir.
Protocol
Representative Results
Discussion
PIB-MS, PPPome’u çeşitli numune kaynaklarından tek bir analizde kantitatif olarak profillemek için kullanılan kimyasal bir proteomik yaklaşımdır. Kinomu incelemek için kinaz inhibitörü boncukları kullanarak ve kanser ve diğer hastalık durumlarında nasıl değiştiğini incelemek için çok fazla çalışma yapılmıştır10,11,12,13. Yine de, PPPome çalışması geride kalıyor….
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
A.N.K., NIH R33 CA225458 ve R35 GM119455’in desteğini kabul eder. Kettenbach ve Gerber laboratuvarlarına yararlı tartışmaları için teşekkür ederiz.
Materials
| Acetonitrile (ACN) | Honeywell | AH015-4 | CAUTION: ACN is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Anhydrous Acetonitrile | Sigma-Aldrich | 271004-100ML | CAUTION: ACN is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Benchtop centrifuge | Eppendorf | model no. 5424 | |
| Beta-glycerophosphoric acid, disodium salt pentahydrate | Acros Organics | 410991000 | |
| Centrifuge | Eppendorf | model no. 5810 R 15 amp version | |
| Distilled water | |||
| DMSO | Fisher Scientific | BP231-100 | |
| Dounce tissue grinder | Fisherbrand Pellet Pestles | 12-141-363 | |
| Empore solid phase extraction disk, C18 | CDS Analytical | 76333-132 | |
| Eppendorf tubes, 1.5 mL | Eppendorf | 22363204 | CRITICAL: Other tubes may leach polymer into sample, contaminating the analysis. |
| Eppendorf tubes, 2 mL | Eppendorf | 22363352 | CRITICAL: Other tubes may leach polymer into sample, contaminating the analysis. |
| Extraction plate manifold | Waters | WAT097944 | |
| Falcon tubes, 50 mL | VWR | 21008 | |
| Generic blunt end needle and plunger | |||
| Generic magnetic separation rack | |||
| HEPES | Sigma-Aldrich | H3375 | |
| Hydrogen chloride (HCl) | VWR Chemicals BDH | BDH3028 | CAUTION: HCl is corrosive; wear gloves and work in a chemical fume hood. |
| Hydroxylamine solution 50% (wt/vol) | Sigma-Aldrich | 467804 | |
| Incubator, 65 °C | VWR | model no. 1380FM | |
| Koptec Pure Ethanol, 200 Proof | Decon Labs | V1001 | |
| Methanol for HPLC (MeOH) | Sigma-Aldrich | 34860-4L-R | CAUTION: MeOH is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Microcystin LR (MCLR) | Cayman Chemical | 10007188 | CAUTION: MCLR is toxic; wear gloves when handling and avoid skin contact. |
| PBS, 1× without calcium and magnesium, pH 7.4 ± 0.1 | Corning | 21-040-CV | |
| pH test strips, such as MilliporeSigma MColorpHast pH test strips and indicator papers | Fisher Scientific | M1095310001 | |
| PIBs | For protocol for the generation of PIBs, see Moorhead et al., 2007. | ||
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23225 | |
| Pipette tips, 10 μL | Eppendorf | 22491504 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| Pipette tips, 1000 μL | Eppendorf | 22491555 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| Pipette tips, 200 μL | Eppendorf | 22491539 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| plastic syringe, 10 mL | BD | 309604 | |
| Protease inhibitor cocktail III | Research Products International | P50700-1 | |
| Q Exactive Plus Hybrid Quadrupole-Orbitrap Mass Spectrometer, Oribtrap Fusion, Orbitrap Fusion Lumos, or Orbitrap Eclipse Tribrid Mass Spectrometer | Thermo Scientific | ||
| Refrigerated benchtop centrifuge | Eppendorf | model no. 5424 R | |
| Rotator (Labquake Shaker Rotisserie) | Thermo Scientific | 13-687-12Q | 8 rpm rotation |
| Sample collection plate, 96- well, 1 mL | Waters | WAT058957 | |
| SDS | Fisher Scientific | BP1311-1 | |
| Sequencing grade modified trypsin | Promega | V511C | |
| Sodium azide | EMD Chemicals | SX0299-1 | CAUTION: Sodium azide is explosive and toxic; wear gloves, work in a chemical fume hood and avoid contact with metals. |
| Sodium chloride (NaCl) | Fisher Chemical | S27110 | |
| Sonicator (Branson digital sonifier) | model no. SFX 250 | ||
| SPE C18 desalting plate | Waters | 186001828BA | |
| SpeedBeads magnetic carboxylate modified particles (SP3 beads) | Cytiva | 6.51521E+13 | |
| Thermomixer | Eppendorf | model no. 5350 | |
| TMT10plex Isobaric Label Reagent Set plus TMT11-131C Label Reagent, 3 × 0.8 mg per tag | ThermoFisher | A37725 | |
| Trifluoroacetic acid (TFA) | Honeywell | T6508-25ML | CAUTION: TFA is corrosive and will irritate skin on contact. Wear gloves and eye protection, and work in a chemical fume hood. |
| Tris Base | Research Products International | T60040 | |
| Triton X-100 | Sigma-Aldrich | T9284 | |
| Vacuum centrifuge and vapor trap | Thermo Scientific | model nos. SpeedVac SPD120 and RVT5105 | |
| Vortexer (Vortex-Genie 2) | Scientific Industries | ||
| Water LC-MS | Honeywell | LC365-4 |
Riferimenti
- Nilsson, J. Protein phosphatases in the regulation of mitosis. Journal of Cell Biology. 218 (2), 395-409 (2019).
- Brautigan, D. L. Protein Ser/Thr phosphatases–the ugly ducklings of cell signalling. The FEBS Journal. 280 (2), 324-345 (2013).
- Brautigan, D. L., Shenolikar, S. Protein serine/threonine phosphatases: keys to unlocking regulators and substrates. Annual Review of Biochemistry. 87, 921-964 (2018).
- Janssens, V., Goris, J. Protein phosphatase 2A: A highly regulated family of serine/threonine phosphatases implicated in cell growth and signalling. Biochemical Journal. 353, 417-439 (2001).
- Virshup, D. M., Shenolikar, S. From promiscuity to precision: protein phosphatases get a makeover. Molecular Cell. 33 (5), 537-545 (2009).
- Bollen, M., Peti, W., Ragusa, M. J., Beullens, M. The extended PP1 toolkit: designed to create specificity. Trends in Biochemical Sciences. 35 (8), 450-458 (2010).
- Qian, J., Winkler, C., Bollen, M. 4D-networking by mitotic phosphatases. Current Opinion in Cell Biology. 25 (6), 697-703 (2013).
- Heroes, E., et al. The PP1 binding code: a molecular-lego strategy that governs specificity. The FEBS Journal. 280 (2), 584-595 (2013).
- Eichhorn, P. J., Creyghton, M. P., Bernards, R. Protein phosphatase 2A regulatory subunits and cancer. Biochimica et Biophysica Acta. 1795 (1), 1-15 (2009).
- Bantscheff, M., et al. Quantitative chemical proteomics reveals mechanisms of action of clinical ABL kinase inhibitors. Nature Biotechnology. 25 (9), 1035-1044 (2007).
- Klaeger, S., et al. Chemical proteomics reveals ferrochelatase as a common off-target of kinase inhibitors. ACS Chemical Biology. 11 (5), 1245-1254 (2016).
- Duncan, J. S., et al. Dynamic reprogramming of the kinome in response to targeted MEK inhibition in triple-negative breast cancer. Cell. 149 (2), 307-321 (2012).
- Cooper, M. J., et al. Application of multiplexed kinase inhibitor beads to study kinome adaptations in drug-resistant leukemia. PLoS ONE. 8 (6), 66755 (2013).
- Lyons, S. P., et al. A quantitative chemical proteomic strategy for profiling phosphoprotein phosphatases from yeast to humans. Molecular and Cellular Proteomics. 17 (12), 2448-2461 (2018).
- Nasa, I., et al. Quantitative kinase and phosphatase profiling reveal that CDK1 phosphorylates PP2Ac to promote mitotic entry. Science Signaling. 13 (648), (2020).
- Swingle, M., Ni, L., Honkanen, R. E. Small-molecule inhibitors of ser/thr protein phosphatases: specificity, use and common forms of abuse. Methods in Molecular Biology. 365, 23-38 (2007).
- Moorhead, G. B. G., Haystead, T. A. J., MacKintosh, C. Synthesis and use of the protein phosphatase affinity matrices microcystin-sepharose and microcystin-biotin-sepharose. Methods in Molecular Biology. 365, 39-45 (2007).
- Brauer, B. L., Wiredu, K., Mitchell, S., Moorhead, G. B., Gerber, S. A., Kettenbach, A. N. Affinity-based profiling of endogenous phosphoprotein phosphatases by mass spectrometry. Nature Protocols. 16 (10), 4919-4943 (2021).
- Hughes, C. S., Moggridge, S., Müller, T., Sorensen, P. H., Morin, G. B., Krijgsveld, J. Single-pot, solid-phase-enhanced sample preparation for proteomics experiments. Nature Protocols. 14 (1), 68-85 (2019).
- Rappsilber, J., Mann, M., Ishihama, Y. Protocol for micro-purification, enrichment, pre-fractionation and storage of peptides for proteomics using StageTips. Nature Protocols. 2 (8), 1896-1906 (2007).
- Zecha, J., et al. TMT labeling for the masses: A robust and cost-efficient, in-solution labeling approach. Molecular and Cellular Proteomics. 18 (7), 1468-1478 (2019).
- Elias, J. E., Gygi, S. P. Target-decoy search strategy for increased confidence in large-scale protein identifications by mass spectrometry. Nature Methods. 4 (3), 207-214 (2007).
- Eng, J. K., Jahan, T. A., Hoopmann, M. R. Comet: an open-source MS/MS sequence database search tool. Proteomics. 13 (1), 22-24 (2013).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nature Methods. 13 (9), 731-740 (2016).
- Yu, S. H., Ferretti, D., Schessner, J. P., Rudolph, J. D., Borner, G. H. H., Cox, J. Expanding the Perseus software for omics data analysis With custom plugins. Current Protocols in Bioinformatics. 71 (1), 1-29 (2020).

