Isolierung, Charakterisierung und Gesamt-DNA-Extraktion zur Identifizierung endophytischer Pilze in mykoheterotrophen Pflanzen
Summary
Der vorliegende Artikel zielt darauf ab, detaillierte und adäquate Protokolle für die Isolierung pflanzenassoziierter endophytischer Pilze, die Langzeitkonservierung von Isolaten, die morphologische Charakterisierung und die Gesamt-DNA-Extraktion für die anschließende molekulare Identifizierung und metagenomische Analyse bereitzustellen.
Abstract
Mykoheterotrophe Pflanzen stellen eine der extremsten Formen der Mykorrhizaabhängigkeit dar, da sie ihre autotrophe Kapazität vollständig verloren haben. Die Pilze, mit denen diese Pflanzen eng verbunden sind, sind für sie so wichtig wie jede andere lebenswichtige Ressource. Daher sind einige der relevantesten Techniken zur Untersuchung mykoheterotropher Arten diejenigen, die die Untersuchung assoziierter Pilze ermöglichen, insbesondere solcher, die Wurzeln und unterirdische Organe bewohnen. In diesem Zusammenhang werden häufig Techniken zur Identifizierung kulturabhängiger und kulturunabhängiger endophytischer Pilze eingesetzt. Die Isolierung von Pilzendophyten bietet die Möglichkeit, sie morphologisch zu identifizieren, ihre Diversität zu analysieren und Inokula für Anwendungen in der symbiotischen Keimung von Orchideensamen zu erhalten. Es ist jedoch bekannt, dass es eine große Vielfalt von nicht kultivierbaren Pilzen gibt, die Pflanzengewebe bewohnen. Kulturunabhängige molekulare Identifizierungstechniken bieten somit eine breitere Abdeckung der Artenvielfalt und -häufigkeit. Ziel dieses Beitrags ist es, die methodische Unterstützung zu geben, die notwendig ist, um zwei Untersuchungsverfahren zu starten: ein kulturabhängiges und ein unabhängiges. In Bezug auf das kulturabhängige Protokoll werden die Prozesse der Entnahme und Pflege von Pflanzenproben von den Entnahmestellen bis hin zu Laboreinrichtungen detailliert beschrieben, zusammen mit der Isolierung filamentöser Pilze aus unterirdischen und luftigen Organen mykoheterotropher Pflanzen, der Aufbewahrung einer Sammlung von Isolaten, der morphologischen Charakterisierung von Hyphen durch Objektträgerkulturmethoden und der molekularen Identifizierung von Pilzen durch vollständige DNA-Extraktion. Zu den detaillierten Verfahren, die kulturunabhängige Methoden umfassen, gehören die Entnahme von Pflanzenproben für metagenomische Analysen und die Extraktion von Gesamt-DNA aus achlorophyllösen Pflanzenorganen mit einem kommerziellen Kit. Schließlich werden auch Kontinuitätsprotokolle (z. B. Polymerase-Kettenreaktion [PCR], Sequenzierung) für Analysen vorgeschlagen und Techniken vorgestellt.
Introduction
Endophytische Pilze sind per Definition solche, die das Innere von Pflanzenorganen und -geweben in unauffälligen Infektionen besiedeln (d. h. ohne ihrem Wirt Schaden zuzufügen)1,2. Diese Pilze können neutral oder vorteilhaft mit Wirtspflanzen interagieren, können Resistenzen gegen Krankheitserreger und ungünstige Umweltbedingungen verleihen und zur Synthese nützlicher Verbindungen für die Pflanze beitragen (z. B. Wachstumsfaktoren und andere Phytohormone)1,3. Mykorrhiza-Endophyten sind Pilze, die Mykorrhiza-Assoziationen mit der Pflanze eingehen und am Nährstofftransfer beteiligtsind 4. Bei Orchidaceae ist die Interaktion mit Mykorrhiza-Endophyten grundlegend für die Samenkeimung bei der überwiegenden Mehrheit der Arten und die Etablierung von Keimlingen bei allen Pflanzen der Familie5. In solchen Kontexten stellen mykoheterotrophe Orchideen einen Fall von völliger Abhängigkeit von ihren Mykorrhizapartnern dar, da sie während ihres gesamten Lebenszyklus auf die Übertragung von Mineralstoffen und Kohlenstoffverbindungen durch diese Pilze angewiesen sind6. Daher ist die Isolierung und Identifizierung assoziierender Pilze eine grundlegende Grundlage für die Untersuchung mykoheterotropher Lebensstrategien. Darüber hinaus ist wenig über die Rolle von Pilzendophyten in mykoheterotrophen Pflanzen oder gar über die tatsächliche Diversität dieser Pilze bekannt 7,8.
Die Untersuchung endophytischer Pilze kann mit verschiedenen Techniken durchgeführt werden, die traditionell als kulturunabhängig oder -abhängig beschrieben werden, zum Beispiel: (a) direkte Beobachtung, (b) Pilzisolierung und morphologische und/oder molekulare Identifizierung und (c) Gesamt-DNA-Extraktion von Pflanzengeweben und molekulare Identifizierung9. In direkter Beobachtung (a) können endophytische Pilze noch im Inneren von Pflanzenzellen und -geweben mittels Licht- oder Elektronenmikroskopie untersucht werden9, da verschiedene Mikroskopieprotokolle von Pena-Passos et al.10 detailliert beschrieben werden. Mit Hilfe von Isolationsmethoden (b) können pilzliche Endophyten nach ihren Kolonien, Hyphen und der Morphologie der Fortpflanzungs- oder Resistenzstruktur charakterisiert werden. Darüber hinaus ist es über Isolationstechniken möglich, die molekulare Identifizierung von Isolaten durch DNA-Extraktion, Amplifikation molekularer Identifizierungssequenzen (Barcodes oder Fingerabdrücke) und Sequenzierung durchzuführen11. Die letztgenannte Technik (c) ermöglicht die molekulare Identifizierung endophytischer Pilze mittels DNA-Extraktion im Inneren von Pflanzengeweben (Metabarcoding), gefolgt von der Bibliotheksvorbereitung und -sequenzierung12.
Darüber hinaus können Pilzisolate in symbiotischen Keimversuchen unter Verwendung von Samen von autotrophen oder mykoheterotrophen Orchideen eingesetzt werden. Ein Beispiel für eine solche Anwendung ist die von Sisti et al.13 durchgeführte Untersuchung, die die Keimung und die Anfangsstadien der Protokormentwicklung bei Pogoniopsis schenckii, einer mykoheterotrophen Orchidee, in Verbindung mit einigen ihrer Isolate, die nicht-mykorrhiza-endophytische Pilze enthalten, beschreibt. Das angewandte symbiotische Keimungsprotokoll wird detailliert beschrieben und in einem Video von Pena-Passos et al.10 vorgestellt. Die Isolierung von Pilzen in Assoziation mit verschiedenen Pflanzenorganen ermöglicht vielfältige Untersuchungsschwerpunkte hinsichtlich der Natur von Pflanzen-Pilz-Interaktionen (z.B. um entweder ökologische oder physiologische Aspekte der Assoziation zu verstehen, sowie Untersuchungen zum Nährstofftransfer von Pilzen auf die Pflanze)9.
Die in Abschnitt 1 vorgestellten Methoden basieren auf einer Sammlung von unterirdischen Organproben, da diese Organe die größten Schwierigkeiten bei der Entnahme aufweisen und von großem Interesse sind, da sie von Mykorrhiza-Endophyten besiedelt werden. Beide enthaltenen Protokolle (Schritte 1.1 und 1.2) können jedoch auf andere mykoheterotrophe Pflanzenorgane (z. B. Rhizome, Blütenstängel und Früchte) angewendet werden. Die in Schritt 1.1 beschriebene Entnahmemethodik ist für die Isolierung endophytischer Pilze (Abschnitt 2) zur morphologischen Charakterisierung (Abschnitte 4 und 5) und/oder die Gesamt-DNA-Extraktion zur Identifizierung von Isolaten (Abschnitt 6) vorgesehen. Andererseits ist die in Schritt 1.2 beschriebene Entnahmemethodik ausschließlich der Gesamt-DNA-Extraktion von Pflanzengeweben für Metabarcoding-Techniken zugeordnet (Abschnitt 7). In Abschnitt 3 werden vier Methoden zur Lagerung und Konservierung von Fadenpilzen vorgestellt, zwei für die kurzfristige Lagerung (3-6 Monate) und die anderen beiden für die langfristige Lagerung (>1 Jahr). Die morphologische Charakterisierung (Abschnitte 4 und 5) kann mit der molekularen Identifizierung verbunden werden, um sie zu untermauern und wichtige Informationen über die Makro- und Mikromorphologie von Pilzen zu liefern. Abbildung 1 fasst die im Folgenden beschriebenen kollektiven Methoden zusammen.
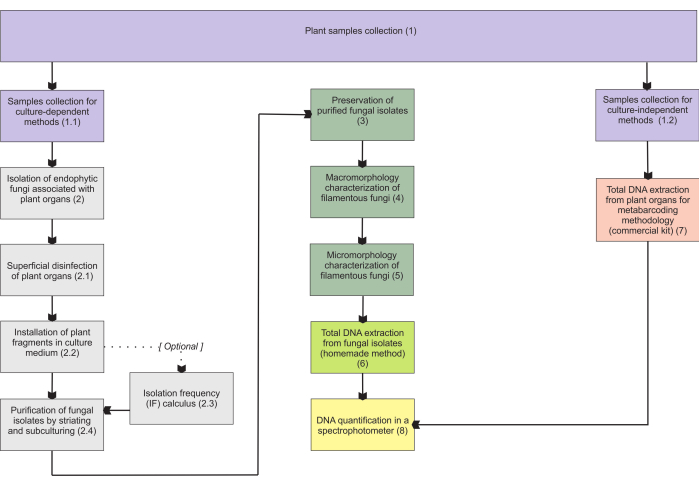
Abbildung 1: Schematische Zusammenfassung der vorgestellten Methoden. Pflanzensammlung und Pilzisolierung, Konservierung und molekulare Identifizierung durch kulturabhängige und -unabhängige Methoden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Protocol
Representative Results
Discussion
Die oberflächliche Entwesung von Pflanzenproben ist eine der kritischsten Phasen des vorgestellten Protokolls. Keine Kontamination des PDA-Geschirrs mit Tropfen aus dem letzten Waschgang ist sehr wünschenswert. Bakterien werden häufig als Verunreinigungen in den Isolierschalen beobachtet, in der Regel mehr als luftgetragene sporulierende Pilze, wenn man bedenkt, dass endophytische Bakterien auch in Pflanzengeweben häufig vorkommen 3,11. Daher ist die Zugabe v…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Wir danken für die Finanzierung durch FAPESP (2015/26479-6) und CNPq (447453/2014-9). JLSM bedankt sich bei CNPq für Produktivitätszuschüsse (303664/2020-7). MPP bedankt sich bei Capes (Master-Stipendium, Prozess 88887.600591/2021-00) und CNPq.
Materials
| Adhesive tape | (from any company, for adhesive tape mount in micromorphological analyses) | ||
| Ampicillin | Sigma-Aldrich | A5354 | (for installation of plant fragments; other antibiotics may be used – check step 2.2.1) |
| Autoclave | (from any company, for materials sterilization in many steps) | ||
| Bacteriological agar | Sigma-Aldrich | A1296 | (for many steps) |
| C1, C2, C3, C4, C5, and C6 solutions | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Centrifuge | Merck/Eppendorf | 5810 G | (for total DNA extraction from fungal isolates) |
| Centrifuge tubes | Merck | CLS430828 | (for samples collection) |
| Chloroform | Sigma-Aldrich | C2432 | (for total DNA extraction from fungal isolates) |
| Congo red | Supelco | 75768 | (for hyphae staining) |
| Cryotubes | Merck | BR114831 | (for many steps) |
| Ethanol | Supelco | 100983 | It will be necessary to carry out the appropriate dilutions (for many steps) |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | 3609 | (for total DNA extraction from fungal isolates) |
| Filter paper | Merck | WHA10010155 | (for many steps) |
| Glass test tubes | Merck | CLS7082516 | (for cryopreservation in unhulled rice grains) |
| Glass wool | Supelco | 20411 | (for cryopreservation in unhulled rice grains) |
| Glucose | Sigma-Aldrich | G8270 | Or dextrose (for cryopreservation in vermiculite) |
| Glycerol | Sigma-Aldrich | G5516 | Or glycerin (for cryopreservation in vermiculite, for preparing LPCB) |
| Isopropanol | Sigma-Aldrich | 563935 | (for total DNA extraction from fungal isolates) |
| Lactic acid | Sigma-Aldrich | 252476 | (for preparing LPCB – hyphae staining) |
| Lactophenol blue solution (LPCB) | Sigma-Aldrich | 61335 | (for hyphae staining) |
| Laminar flow hood | (class I, from any company, for many steps) | ||
| Light microscope | (from any company, for hyphae observation) | ||
| MB Spin Columns | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Methyl blue (cotton blue) | Sigma-Aldrich | M5528 | (for preparing LPCB – hyphae staining) |
| Microcentrifuge tube (1.5 mL) | Merck | HS4323 | (for total DNA extraction from fungal isolates) |
| Microcentrifuge tube (2 mL) | Merck | BR780546 | (for many steps) |
| Mineral oil | (for preservation of fungal isolates) | ||
| Paper bags | Average size 150 mm x 200 mm (for samples collection) | ||
| Petri dish (Glass, 120 mm x 20 mm) | Merck/Pyrex | SLW1480/10D | (autoclavable, for fungi slide culture, prefer higher ones) |
| Petri dish (Glass, 50 mm x 17 mm) | Merck/Aldrich | Z740618 | (for purification of fungal isolates); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Petri dish (Glass, 80 mm x 15 mm) | Merck/Brand | BR455732 | (for installation of plant fragments); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Phenol | Sigma-Aldrich | P1037 | (for total DNA extraction from fungal isolates, for preparing LPCB) |
| Porcelain mortar | Sigma-Aldrich | Z247464 | (for total DNA extraction from fungal isolates) |
| Porcelain pestle | Sigma-Aldrich | Z247502 | (for total DNA extraction from fungal isolates) |
| Potato dextrose agar (PDA) | Millipore | P2182 | (for many steps) |
| PowerBead tubes | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Rapid mounting medium (Entellan) | Sigma-Aldrich | 1.0796 | (for fungi slide culture) |
| Silica gel | Supelco | 717185 | (for cryopreservation in unhulled rice grains) |
| Sodium chloride (NaCl) | Sigma-Aldrich | S9888 | (for total DNA extraction from fungal isolates) |
| Sodium dodecyl sulfate (SDS) | Sigma-Aldrich | L3771 | Lauryl sulfate sodium salt (for total DNA extraction from fungal isolates) |
| Sodium hypochlorite (w/ 2% active chlorine) | (commercial product, for superficial desinfestation) | ||
| Soil DNA extraction kit (DNeasy PowerSoil kit) | Qiagen | 12888-50 | (for total DNA extraction from plant organs) |
| Spectrophotometer – Nanodrop 2000/2000c | ThermoFisher Scientific | ND2000CLAPTOP | (for total DNA extraction from plant organs) |
| Stereomicroscope | (=dissecting microscope, from any company, for macromorphological analyses) | ||
| Tetracycline | Sigma-Aldrich | T7660 | (for installation of plant fragments) |
| Thermoblock | Merck/Eppendorf | EP5362000035 | (or from other companies) |
| Tissue homogenizer and cell lyzer | SPEX SamplePrep | 2010 Geno/Grinder – Automated Tissue Homogenizer and Cell Lyzer (for total DNA extraction from plant organs) | |
| Toluidine blue O | Sigma-Aldrich/Harleco | 364-M | (for hyphae staining) |
| Trehalose | Sigma-Aldrich | T9531 | (for cryopreservation in vermiculite) |
| Tris Base Solution (Tris) | Sigma-Aldrich | T1699 | (for total DNA extraction from fungal isolates) |
| Unhulled rice grains | (for cryopreservation) | ||
| U-shaped glass rod | (or an adaptation – check step 5.4.1, for fungi slide culture) | ||
| Vermiculite | Fine granulometry (for cryopreservation in vermiculite) | ||
| Vortexer | Sigma-Aldrich/BenchMixer | BMSBV1000 | (for total DNA extraction from fungal isolates) |
| Yeast extract | Sigma-Aldrich | Y1625 | (for cryopreservation in vermiculite) |
Riferimenti
- de Azevedo, J. L. Endophytic microorganisms. Ecologia Microbiana. , 117-137 (1998).
- Stone, J. K., Bacon, C. W., White, J. F. An overview of endophytic microbes: endophytism defined. Microbial Endophytes. , 17-44 (2000).
- Schulz, B., Boyle, C. What are Endophytes. Microbial Root Endophytes. , 1-13 (2006).
- Smith, S. E., Read, D. J. . Mycorrhizal Symbiosis. , (2008).
- Rasmussen, H. N., Dixon, K. W., Jersáková, J., Těšitelová, T. Germination and seedling establishment in orchids: a complex of requirements. Annals of Botany. 116 (3), 391-402 (2015).
- Rasmussen, H. N., Rasmussen, F. N. Orchid mycorrhiza: implications of a mycophagous life style. Oikos. 118 (3), 334-345 (2009).
- Ma, X., Kang, J., Nontachaiyapoom, S., Wen, T., Hyde, K. D. Non-mycorrhizal endophytic fungi from orchids. Current Science. 109 (1), 72-87 (2015).
- Favre-Godal, Q., Gourguillon, L., Lordel-Madeleine, S., Gindro, K., Choisy, P. Orchids and their mycorrhizal fungi: an insufficiently explored relationship. Mycorrhiza. 30 (1), 5-22 (2020).
- Sun, X., Guo, L. -. D. Endophytic fungal diversity: review of traditional and molecular techniques. Mycology. 3 (1), 65-76 (2012).
- Pena-Passos, M., Sisti, L. S., Mayer, J. L. S. Microscopy techniques for interpreting fungal colonization in mycoheterotrophic plants tissues and symbiotic germination of seeds. Journal of Visualized Experiments. (183), e63777 (2022).
- Araújo, W. L., et al. . Endophytic microorganisms: Theoretical and Practical Aspects of Isolation and Characterization. 1st ed. 1, 257 (2014).
- de Souza, R. S. C., et al. Unlocking the bacterial and fungal communities assemblages of sugarcane microbiome. Scientific Reports. 6, 28774 (2016).
- Sisti, L. S., et al. The role of non-mycorrhizal fungi in germination of the mycoheterotrophic orchid Pogoniopsis schenckii Cogn. Frontiers in Plant Science. 10, 1589 (2019).
- Araújo, W. L., et al. Variability and interactions between endophytic bacteria and fungi isolated from leaf tissues of citrus rootstocks. Canadian Journal of Microbiology. 47 (3), 229-236 (2001).
- Castellani, A. Further researches on the long viability and growth of many pathogenic fungi and some bacteria in sterile distilled water. Mycopathologia. 20 (1-2), 1-6 (1963).
- Currah, R. S., Zelmer, C. D., Hambleton, S., Richardson, K. A. Fungi from orchid mycorrhizas. Orchid Biology: Reviews and Perspectives, VII. , 117-170 (1997).
- Freitas, E. F. S., et al. Diversity of mycorrhizal Tulasnella associated with epiphytic and rupicolous orchids from the Brazilian Atlantic Forest, including four new species. Scientific Reports. 10 (1), 7069 (2020).
- Sato, M., Inaba, S., Noguchi, M., Nakagiri, A. Vermiculite as a culture substrate greatly improves the viability of frozen cultures of ectomycorrhizal basidiomycetes. Fungal Biology. 124 (8), 742-751 (2020).
- Pereira, O. L., Kasuya, M. C. M., Borges, A. C., Araújo, E. F. D. Morphological and molecular characterization of mycorrhizal fungi isolated from neotropical orchids in Brazil. Canadian Journal of Botany. 83 (1), 54-65 (2005).
- Riddell, R. W. Permanent stained mycological preparations obtained by slide culture. Mycologia. 42 (2), 265-270 (1950).
- Walsh, T. J., Hayden, R. T., Larone, D. H. . Larone’s Medically Important Fungi: A Guide to Identification. , (2018).
- Microscopy: Chemical Reagents. British Mycological Society Available from: https://www.britmycolsoc.org.uk/field_mycology/microscopy/reagents (2022)
- Senanayake, I. C., et al. Morphological approaches in studying fungi: Collection, examination, isolation, sporulation and preservation. Mycosphere. 11 (1), 2678-2754 (2020).
- Slifkin, M., Cumbie, R. Congo red as a fluorochrome for the rapid detection of fungi. Journal of Clinical Microbiology. 26 (5), 827-830 (1988).
- Raeder, U., Broda, P. Rapid preparation of DNA from filamentous fungi. Letters in Applied Microbiology. 1 (1), 17-20 (1985).
- Martins, M. K., et al. Molecular characterization of endophytic microorganisms. Endophytic microorganisms: theoretical and practical aspects of isolation and characterization. 1st edition. , 189-211 (2014).
- Rayner, R. W. A Mycological Colour Chart. Commonwealth Mycological Institute. , (1970).
- Kornerup, A., Wanscher, J. H. . Methuen Handbook of Colour. Methuen handbook of colour. , (1967).
- Ridgway, R. . Color Standards and Color Nomenclature. , (1912).
- McGinnis, M. R. . Laboratory Handbook of Medical Mycology. , (2012).
- Webster, J., Weber, R. . Introduction to Fungi. , (2007).
- Sridharan, G., Shankar, A. A. Toluidine blue: A review of its chemistry and clinical utility. Journal of Oral and Maxillofacial Pathology. 16 (2), 251-255 (2012).
- Smith, D., Onions, A. H. S. A comparison of some preservation techniques for fungi. Transactions of the British Mycological Society. 81 (3), 535-540 (1983).
- Ryan, M. J., Smith, D., Jeffries, P. A decision-based key to determine the most appropriate protocol for the preservation of fungi. World Journal of Microbiology and Biotechnology. 16 (2), 183-186 (2000).
- Lalaymia, I., Cranenbrouck, S., Declerck, S. Maintenance and preservation of ectomycorrhizal and arbuscular mycorrhizal fungi. Mycorrhiza. 24 (5), 323-337 (2014).
- Zettler, L. W., Corey, L. L. Orchid mycorrhizal fungi: isolation and identification techniques. Orchid Propagation: From Laboratories to Greenhouses-Methods and Protocols. , 27-59 (2018).
- Yu, S., Wang, Y., Li, X., Yu, F., Li, W. The factors affecting the reproducibility of micro-volume DNA mass quantification in Nanodrop 2000 spectrophotometer. Optik. 145, 555-560 (2017).
- Martos, F., et al. Independent recruitment of saprotrophic fungi as mycorrhizal partners by tropical achlorophyllous orchids. New Phytologist. 184 (3), 668-681 (2009).
- Schoch, C. L., et al. Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for Fungi. Proceedings of the National Academy of Sciences. 109 (16), 6241-6246 (2012).
- White, T. J., Bruns, T., Lee, S., Taylor, J. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. PCR Protocols: A Guide to Methods and Applications. 18 (1), 315-322 (1990).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proceedings of the National Academy of Sciences. 74 (12), 5463-5467 (1977).
- Ranjard, L., et al. Characterization of bacterial and fungal soil communities by automated ribosomal intergenic spacer analysis fingerprints: biological and methodological variability. Applied and Environmental Microbiology. 67 (10), 4479-4487 (2001).
- Metzker, M. L. Sequencing technologies-the next generation. Nature Reviews Genetics. 11 (1), 31-46 (2010).

