Polymerasekjedereaksjon og dot-blot-hybridisering for Leptospira-deteksjon i vannprøver
Summary
I denne studien ble en dot-blot-applikasjon designet for å oppdage Leptospira fra de tre hovedkladene i vannprøver. Denne metoden tillater identifisering av minimale DNA-mengder spesifikt målrettet av en digoksigenin-merket sonde, lett detekteres av et anti-digoksigeninantistoff. Denne tilnærmingen er et verdifullt og tilfredsstillende verktøy for screeningformål.
Abstract
Dot-blot er en enkel, rask, sensitiv og allsidig teknikk som muliggjør identifisering av minimale mengder DNA spesifikt målrettet ved sondehybridisering i nærvær av bærer-DNA. Den er basert på overføring av en kjent mengde DNA til en inert fast støtte, slik som en nylonmembran, ved bruk av dot-blot-apparatet og uten elektroforetisk separasjon. Nylonmembraner har fordelen av høy nukleinsyrebindingskapasitet (400 μg / cm2), høy styrke, og er positivt eller nøytralt ladet. Sonden som brukes er et svært spesifikt ssDNA-fragment på 18 til 20 baser som lenge er merket med digoksigenin (DIG). Sonden vil konjugere med Leptospira DNA. Når sonden har hybridisert med mål-DNA, detekteres den av et anti-digoksigeninantistoff, slik at den enkelt kan detekteres gjennom utslippene som avsløres i en røntgenfilm. Prikkene med en utslipp vil tilsvare DNA-fragmentene av interesse. Denne metoden benytter sondens ikke-isotopiske merking, som kan ha en svært lang halveringstid. Ulempen med denne standard immunetiketten er en lavere følsomhet enn isotopiske prober. Likevel reduseres det ved kobling av polymerasekjedereaksjon (PCR) og dot-blot-analyser. Denne tilnærmingen muliggjør berikelse av målsekvensen og dens deteksjon. I tillegg kan den brukes som en kvantitativ applikasjon sammenlignet med en seriell fortynning av en velkjent standard. En dot-blot-applikasjon for å oppdage Leptospira fra de tre hovedkladene i vannprøver presenteres her. Denne metoden kan brukes på store mengder vann når de har blitt konsentrert ved sentrifugering for å gi bevis på tilstedeværelsen av leptospiralt DNA. Dette er et verdifullt og tilfredsstillende verktøy for generelle screeningformål, og kan brukes til andre ikke-dyrkbare bakterier som kan være tilstede i vann, noe som øker forståelsen av økosystemet.
Introduction
Leptospirose hos mennesker stammer hovedsakelig fra miljøkilder 1,2. Tilstedeværelsen av Leptospira i innsjøer, elver og bekker er en indikator på leptospiroseoverføring blant dyreliv og husdyr og produksjonsdyr som til slutt kan komme i kontakt med disse vannmassene 1,3,4. Videre har Leptospira blitt identifisert i ikke-naturlige kilder, inkludert kloakk, stillestående og vann fra springen 5,6.
Leptospira er en verdensomspennende distribuert bakterie 7,8, og miljøets rolle i bevaring og overføring har blitt godt anerkjent. Leptospira kan overleve i drikkevann under variabel pH og mineraler9, og i naturlige vannmasser1. Den kan også overleve i lange perioder i destillert vann10, og under konstant pH (7,8) kan den overleve opptil 152 dager11. Videre kan Leptospira samhandle i bakterielle konsortier for å overleve tøffe forhold12,13. Det kan være en del av biofilm i ferskvann med Azospirillum og Sphingomonas og er til og med i stand til å vokse og tåle temperaturer over 49 ° C14,15. Det kan også formere seg i vannmettet jord og forbli levedyktig i opptil 379 dager16, og bevare sin evne til å forårsake sykdommen så lenge som et år17,18. Imidlertid er lite kjent om økologien i vannlegemer og hvordan den er fordelt i dem.
Siden oppdagelsen var studien av slekten Leptospira basert på serologiske tester. Det var ikke før det nåværende århundre at molekylære teknikker ble mer utbredt i studiet av denne spirochaete. Dot-blot har knapt blitt brukt til identifikasjon ved hjelp av (1) en isotopisk sonde basert på 16S rRNA og på en inter-enkel sekvensrepetisjon (ISSR) 19,20, (2) som en nanogoldbasert immunoassay for human leptospirose anvendt på urin21, eller (3) som en antistoffbasert analyse for storfe urinprøver22. Teknikken falt ut av bruk fordi den opprinnelig var basert på isotopiske prober. Det er imidlertid en velkjent teknikk som, kombinert med PCR, gir forbedrede resultater, og det anses som trygt på grunn av bruk av ikke-isotopiske prober. PCR spiller en avgjørende rolle i anrikningen av Leptospira DNA ved å amplifisere et spesifikt DNA-fragment som kan finnes i spormengder i en prøve. Under hver PCR-syklus dobles mengden av det målrettede DNA-fragmentet i reaksjonen. På slutten av reaksjonen har amplikonen blitt multiplisert med en faktor på mer enn en million23. Produktet forsterket av PCR, ofte ikke synlig i agaroseelektroforese, blir synlig gjennom spesifikk hybridisering med en DIG-merket sonde i dot-blot 24,25,26.
Dot-blot-teknikken er enkel, robust og egnet for mange prøver, noe som gjør den tilgjengelig for laboratorier med begrensede ressurser. Det har blitt brukt i en rekke bakteriestudier, inkludert (1) orale bakterier27, (2) andre prøvetyper som mat og avføring28, og (3) identifisering av unculturable bakterier29, ofte i samsvar med andre molekylære teknikker. Blant fordelene som tilbys av dot-blot-teknikken er: (1) Membranen har en høy bindingskapasitet, i stand til å binde over 200 μg / cm2 nukleinsyrer og opptil 400 μg / cm2; (2) Dot-blot-resultater kan tolkes visuelt uten å kreve spesialutstyr, og (3) de kan enkelt lagres i årevis ved romtemperatur (RT).
Slekten Leptospira har blitt klassifisert i patogene, mellomliggende og saprofytiske klader30,31. Skillet mellom disse kladene kan oppnås basert på spesifikke gener som lipL41, lipL32 og 16S rRNA. LipL32 er tilstede i de patogene kladene og utviser høy sensitivitet i ulike serologiske og molekylære verktøy, mens den er fraværende i saprofyttarter21. Housekeeping-genet lipL41 er kjent for sitt stabile uttrykk og brukes i molekylære teknikker32, mens 16S rRNA-genet brukes til klassifisering.
Denne metoden kan brukes på store mengder vann når de har blitt konsentrert ved sentrifugering. Det tillater vurdering av ulike punkter og dybder i en vannkropp for å oppdage tilstedeværelsen av leptospiralt DNA og kladen som den tilhører. Dette verktøyet er verdifullt for både økologiske og generelle screeningformål, og kan også brukes til å oppdage andre ikke-dyrkbare bakterier som kan være tilstede i vann.
I tillegg er PCR- og dot-blot-analyser teknisk og økonomisk rimelige for et bredt spekter av laboratorier, selv de som mangler sofistikert eller dyrt utstyr. Denne studien tar sikte på å anvende digoksigeninbasert dot-blot for identifisering av de tre Leptospira-klader i vannprøver samlet fra naturlige vannmasser.
Bakterielle stammer
Tolv Leptospira serovarer (Autumnalis, Bataviae, Bratislava, Canicola, Celledoni, Grippothyphosa, Hardjoprajitno, Icterohaemorrhagiae, Pomona, Pyrogenes, Tarassovi, og Wolffi) ble inkludert i denne studien. Disse serovarene er en del av samlingen ved Institutt for mikrobiologi og immunologi, Fakultet for veterinærmedisin og zooteknikk, National Autonomous University of Mexico, og de brukes for tiden i mikroagglutineringstesten (LAR).
Alle Leptospira serovarer ble dyrket i EMJH, og deres DNA ble ekstrahert ved hjelp av et kommersielt DNA-ekstraksjonssett (se materialtabell). En genomisk DNA-blanding av de tolv serovarene ble brukt som en positiv kontroll for den patogene kladen Leptospira . Som positiv kontroll av mellomkladen Leptospira ble genomisk DNA fra Leptospira fainei serovar Hurstbridge-stamme BUT6 inkludert, og som positiv kontroll for Leptospira-saprofyttkladen ble også genomisk DNA fra Leptospira biflexa serovar Patoc-stammen Patoc I, inkludert.
Negative kontroller besto av et tomt plasmid, DNA fra ikke-relaterte bakterier (Ureaplasma urealyticum, Staphylococcus aureus, Brucella abortus, Salmonella typhimurium, Shigella boydii, Klebsiella pneumoniae, Acinetobacter baumannii og Escherichia coli) og PCR-gradig vann, som fungerte som ikke-malkontroll.
Vannprøver
Tolv prøvetakingsprøver ble samlet inn ved hjelp av en stratifisert-tilfeldig prøvetakingsmetode fra Cuemanco Biological and Aquaculture Research Center (CIBAC) (19° 16′ 54″ N 99° 6′ 11″ W). Disse prøvene ble tatt på tre dybder: overfladisk, 10 og 30 cm (figur 1A, B). Vanninnsamlingsprosedyrene påvirket ingen truede eller beskyttede arter. Hver prøve ble samlet i et sterilt 15 ml mikrosentrifugerør. For å samle prøven ble hvert rør forsiktig nedsenket i vannet, fylt på den valgte dybden og deretter forseglet. Prøvene ble oppbevart ved 22 °C og straks transportert til laboratoriet for behandling.
Hver prøve ble konsentrert ved sentrifugering i sterile 1,5 ml mikrosentrifugerør ved 8000 x g i 20 minutter ved romtemperatur. Dette trinnet ble gjentatt til alle prøvene ble konsentrert i ett rør, som deretter ble brukt til DNA-ekstraksjon (figur 1C).
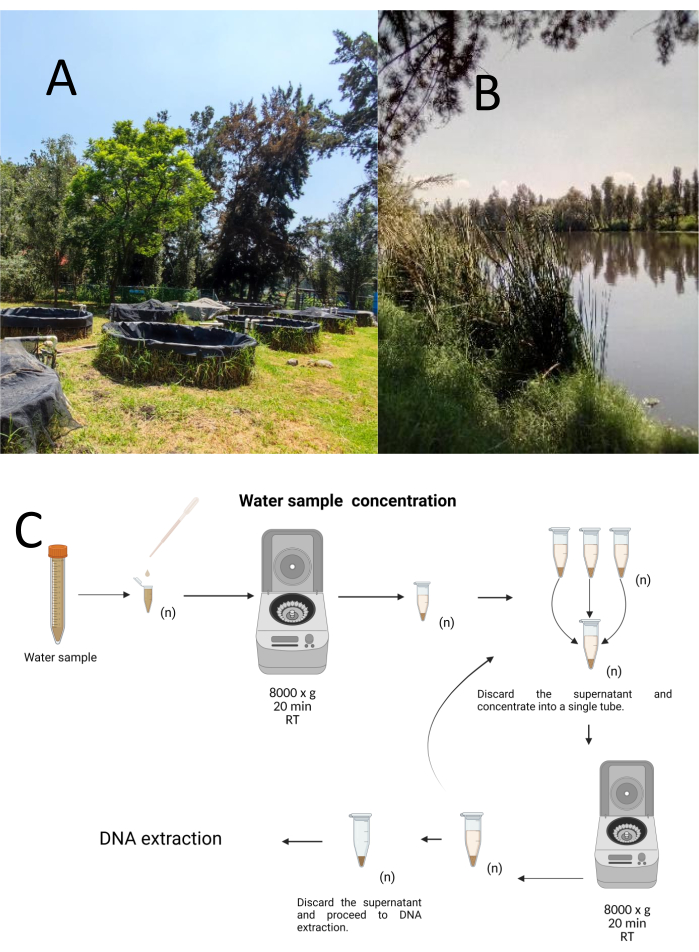
Figur 1: Konsentrasjon av vannprøver ved sentrifugering. (A) Vannprøvedammer, og (B) Naturlige bekker. (C) Sentrifugeringsbasert vannprøvebehandling i gjentatte trinn så mange ganger som nødvendig (n). Klikk her for å se en større versjon av denne figuren.
DNA-ekstraksjon
Totalt DNA ble isolert ved hjelp av et kommersielt genomisk DNA-sett i henhold til produsentens instruksjoner (se materialtabell). DNA-ekstraksjoner ble eluert i 20 μL elueringsbuffer, og DNA-konsentrasjon ble bestemt av et UV-spektrofotometer ved 260-280 nm, og lagret ved 4 °C til bruk.
PCR-amplifikasjon
PCR-målene var 16S rRNA-, lipL41- og lipL32-gener, som identifiserer DNA fra slekten Leptospira og tillater skillet mellom de tre kladene: patogen, saprofytisk og mellomliggende. Både primere og sondedesign var basert på tidligere arbeider av Ahmed et al., Azali et al., Bourhy et al., Weiss et al. og Branger et al.33,34,35,36,37. Sekvensen av hver sonde, primer og forsterkede fragment er beskrevet i tabell 1, og deres tilpasning til referansesekvenser er gitt i tilleggsfil 1, tilleggsfil 2, tilleggsfil 3, tilleggsfil 4 og tilleggsfil 5. PCR-reagensene og termosyklusforholdene er beskrevet i protokollavsnittet.
Forsterkningsprodukter ble visualisert ved elektroforetisk separasjon på en 1% agarosegel i TAE (40 mM Tris-base, 20 mM eddiksyre og 1 mM EDTA; pH 8,3), ved 60 V i 45 minutter med ethidiumbromiddeteksjon, som vist i tilleggsfigur 1. Genomisk DNA oppnådd fra hver serovar ble brukt med konsentrasjoner fra 6 x 106 til 1 x 104 genomiske ekvivalente kopier (GEq) i hver PCR-reaksjon, basert på genomstørrelsen til L. interrogans (4, 691, 184 bp) 38 for patogen Leptospira, genomstørrelsen til L. biflexa (3, 956, 088 bp) 39 for saprofytisk Leptospira, og genomstørrelsen til L. fainei serovar Hurstbridge-stamme BUT6 (4, 267, 324 bp) med tiltredelsesnummer AKWZ00000000.2.
Sensitiviteten til sondene ble vurdert med DNA fra hver patogen serovar, L. biflexa serovar Patoc stamme Patoc I og L. fainei serovar Hurstbridge stamme BUT6 i hvert eksperiment. For å vurdere spesifisiteten til PCR- og dot-blot-hybridiseringsanalysen ble DNA fra ikke-relaterte bakterier inkludert.
Tabell 1: PCR-primere og prober for å amplifisere produkter for å identifisere de patogene, saprofyte og mellomliggende klader av Leptospira. Klikk her for å laste ned denne tabellen.
Dot-blot hybridiseringsanalyse
Teknikken kalles dot-blot fordi hullene der DNA-prøven er plassert har en prikkform, og når de suges for å festes på plass ved vakuumsuging, får de denne formen. Denne teknikken ble utviklet av Kafatos et al.40. Teknikken tillater semi-kvantifisering av Leptospira i hver PCR-positiv prøve. Protokollen består av en denaturering med NaOH 0,4 M ved romtemperatur, prøver med Leptospira DNA fra 30 ng til 0,05 ng, tilsvarende 6 x 106 til 1 x 104 leptospirer, blir blottet på en nylonmembran med et 96-brønns dot-blot-apparat. Etter immobilisering er DNA bundet til membranen ved eksponering for 120 mJ UV-lys. Hver DNA-sonde er konjugert med digoksigenin-11 dUTP ved et terminalt transferasekatalysetrinn i 3′-enden (Digoxigenin er et plantesteroid oppnådd fra Digitalis purpurea, brukt som reporter41). Etter den strenge hybridiseringen av den merkede DNA-sonden (50 pmol) ved den spesifikke temperaturen på mål-DNA, visualiseres DNA-hybridene ved kjemiluminescensreaksjonen med det alkaliske fosfataseantistoffet mot digoksigenin kovalent konjugert med dets substrat CSPD. Luminescensen fanges opp ved eksponering for røntgenfilm (figur 2).
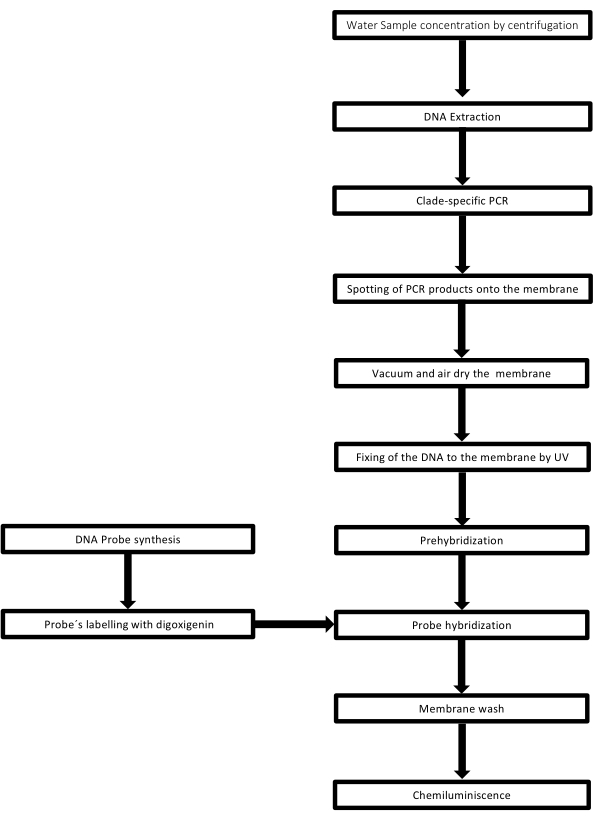
Figur 2: Trinn i prosedyren for PCR-dot-blot-analysen. Klikk her for å se en større versjon av denne figuren.
Protocol
Representative Results
Discussion
De kritiske trinnene i dot-blot-teknikken inkluderer (1) DNA-immobilisering, (2) blokkering av de frie bindingsstedene på membranen med ikke-homologt DNA, (3) komplementariteten mellom sonden og målfragmentet under glødningsbetingelser, (4) fjerning av den uhybridiserte sonden og (5) påvisning av reportermolekylet41.
PCR-Dot-blot har visse begrensninger, for eksempel at teknikken ikke gir informasjon om størrelsen på det hybridiserte fragmentet37…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Vi står i gjeld til Leptospira-samlingen ved Institutt for mikrobiologi og immunologi, Fakultet for veterinærmedisin og zooteknikk, National Autonomous University of Mexico. Vi er takknemlige for den sjenerøse donasjonen av referansen Leptospira-stammer ; Leptospira fainei serovar Hurstbridge stamme BUT6 og Leptospira biflexa serovar Patoc stamme Patoc I til Dr. Alejandro de la Peña Moctezuma. Vi takker Dr. José Antonio Ocampo Cervantes, CIBAC-koordinator, og personellet for deres logistiske støtte. EDT var under Terminal Project-programmet for bachelorstudenter ved Metropolitan Autonomous University-Campus Cuajimalpa. Vi anerkjenner den Biorender.com programvaren for opprettelsen av figur 1 og 3 til 9.
Materials
| REAGENTS | |||
| Purelink DNA extraction kit | Invitrogen | K182002 | |
| Gotaq Flexi DNA Polimerase (End-Point PCR Taq polymerase kit) | Promega | M3001 | |
| Whatman filter paper, grade 1, | Merk | WHA1001325 | |
| Nylon Membranes, positively charged Roll 30cm x 3 m | Roche | 11417240001 | |
| Anti-Digoxigenin-AP, Fab fragments Sheep Polyclonal Primary-antibody | Roche | 11093274910 | |
| Medium Base EMJH | Difco | S1368JAA | |
| Leptospira Enrichment EMJH | Difco | BD 279510 | |
| Blocking Reagent | Roche | 11096176001 | |
| CSPD ready to use Disodium 3-(4-methoxyspiro {1,2-dioxetane-3,2′-(5′-chloro) tricyclo [3.3.1.13,7] decan}8-4-yl) phenyl phosphate | Merk | 11755633001 | |
| Deoxyribonucleic acid from herring sperm | Sigma Aldrich | D3159 | |
| Developer Carestream | Carestream Health Inc | GBX5158621 | |
| Digoxigenin-11-ddUTP | Roche | 11363905910 | |
| EDTA, Disodium Salt (Dihydrate) | Promega | H5032 | |
| Ficoll 400 | Sigma Aldrich | F8016 | |
| Fixer Carestream | Carestream Health Inc | GBX 5158605 | |
| Lauryl sulfate Sodium Salt (Sodium dodecyl sulfate; SDS) C12H2504SNa | Sigma Aldrich | L5750 | |
| N- Lauroylsarcosine sodium salt CH3(CH2)10CON(CH3) CH2COONa | Sigma Aldrich | L-9150 | It is an anionic surfactant |
| Polivinylpyrrolidone (PVP-40) | Sigma Aldrich | PVP40 | |
| Polyethylene glycol Sorbitan monolaurate (Tween 20) | Sigma Aldrich | 9005-64-5 | |
| Sodium Chloride (NaCl) | Sigma Aldrich | 7647-14-5 | |
| Sodium dodecyl sulfate (SDS) | Sigma Aldrich | 151-21-3 | |
| Sodium hydroxide (NaOH) | Sigma Aldrich | 1310-73-2 | |
| Sodium phosphate dibasic (NaH2PO4) | Sigma-Aldrich | 7558-79-4 | |
| Terminal transferase, recombinant | Roche | 3289869103 | |
| Tris hydrochloride (Tris HCl) | Sigma-Aldrich | 1185-53-1 | |
| SSPE 20X | Sigma-Aldrich | S2015-1L | It can be Home-made following Supplementary File 6 |
| Primers | Sigma-Aldrich | On demand | Follow table 1 |
| Probes | Sigma-Aldrich | On demand | Follow table 1 |
| Equipment | |||
| Nanodrop™ One Spectrophotometer | Thermo-Scientific | ND-ONE-W | |
| Refrigerated microcentrifuge Sigma 1-14K, suitable for centrifugation of 1.5 ml microcentrifuge tubes at 14,000 rpm | Sigma-Aldrich | 1-14K | |
| Disinfected adjustable pipettes, range 2-20 µl, 20-200 µl | Gilson | SKU:F167360 | |
| Disposable 1.5 ml microcentrifuge tubes (autoclaved) | Axygen | MCT-150-SP | |
| Disposable 600 µl microcentrifuge tubes (autoclaved) | Axygen | 3208 | |
| Disposable Pipette tips 1-10 µl | Axygen | T-300 | |
| Disposable Pipette tips 1-200 µl | Axygen | TR-222-Y | |
| Dot-Blot apparatus Bio-Dot | BIORAD | 1706545 | |
| Portable Hergom Suction | Hergom | 7E-A | |
| Scientific Light Box (Visible-light PH90-115V) | Hoefer | PH90-115V | |
| UV Crosslinker | Hoefer | UVC-500 | |
| Thermo Hybaid PCR Express Thermocycler | Hybaid | HBPX110 | |
| Radiographic cassette with IP Plate14 X 17 | Fuji |
Riferimenti
- Bierque, E., Thibeaux, R., Girault, D., Soupé-Gilbert, M. E., Goarant, C. A systematic review of Leptospira in water and soil environments. PLOS One. 15 (1), e0227055 (2020).
- Haake, D. A., Levett, P. N. Leptospirosis in humans. Current Topics in Microbiology and Immunology. 387, 65-97 (2015).
- Tripathy, D. N., Hanson, L. E. Leptospires from water sources at Dixon Springs Agricultural Center. Journal of Wildlife Diseases. 9 (3), 209-212 (1973).
- Smith, D. J., Self, H. R. Observations on the survival of Leptospira australis A in soil and water. The Journal of Hygiene. 53 (4), 436-444 (1955).
- Karpagam, K. B., Ganesh, B. Leptospirosis: a neglected tropical zoonotic infection of public health importance-an updated review. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 39 (5), 835-846 (2020).
- Casanovas-Massana, A., et al. Spatial and temporal dynamics of pathogenic Leptospira in surface waters from the urban slum environment. Water Research. 130, 176-184 (2018).
- Costa, F., et al. Global morbidity and mortality of Leptospirosis: A systematic review. PLOS Neglected Tropical Diseases. 9 (9), e0003898 (2015).
- Mwachui, M. A., Crump, L., Hartskeerl, R., Zinsstag, J., Hattendorf, J. Environmental and behavioural determinants of Leptospirosis transmission: A systematic review. PLOS Neglected Tropical Diseases. 9 (9), e0003843 (2015).
- Andre-Fontaine, G., Aviat, F., Thorin, C. Waterborne Leptospirosis: Survival and preservation of the virulence of pathogenic Leptospira spp. in fresh water. Current Microbiology. 71 (1), 136-142 (2015).
- Trueba, G., Zapata, S., Madrid, K., Cullen, P., Haake, D. Cell aggregation: A mechanism of pathogenic Leptospira to survive in freshwater. International Microbiology: the Official Journal of the Spanish Society for Microbiology. 7 (1), 35-40 (2004).
- Smith, C. E., Turner, L. H. The effect of pH on the survival of leptospires in water. Bulletin of the World Health Organization. 24 (1), 35-43 (1961).
- Barragan, V. A., et al. Interactions of Leptospira with environmental bacteria from surface water. Current Microbiology. 62 (6), 1802-1806 (2011).
- Abdoelrachman, R. Comparative investigations into the influence of the presence of bacteria on the life of pathogenic and apathogenic leptospirae. Antonie van Leeuwenhoek. 13 (1), 21-32 (1947).
- Singh, R., et al. Microbial diversity of biofilms in dental unit water systems. Applied and Environmental Microbiology. 69 (6), 3412-3420 (2003).
- Kumar, K. V., Lall, C., Raj, R. V., Vedhagiri, K., Vijayachari, P. Coexistence and survival of pathogenic leptospires by formation of biofilm with Azospirillum. FEMS Microbiology Ecology. 91 (6), 051 (2015).
- Yanagihara, Y., et al. Leptospira Is an environmental bacterium that grows in waterlogged soil. Microbiology Spectrum. 10 (2), 0215721 (2022).
- Gillespie, R. W., Ryno, J. Epidemiology of leptospirosis. American Journal of Public Health and Nation’s Health. 53 (6), 950-955 (1963).
- Bierque, E., et al. Leptospira interrogans retains direct virulence after long starvation in water. Current Microbiology. 77 (10), 3035-3043 (2020).
- Zhang, Y., Dai, B. Marking and detection of DNA of leptospires in the dot-blot and situ hybridization with digoxigenin-labeled probes. Journal of West China University of Medical Sciences. 23 (4), 353-435 (1992).
- Mérien, F., Amouriaux, P., Perolat, P., Baranton, G., Saint Girons, I. Polymerase chain reaction for detection of Leptospira spp. in clinical samples. Journal of Clinical Microbiology. 30 (9), 2219-2224 (1992).
- Veerapandian, R., et al. Silver enhanced nano-gold dot-blot immunoassay for leptospirosis. Journal of Microbiological Methods. 156, 20-22 (2019).
- Junpen, S., et al. Evaluation of a monoclonal antibody-based dot-blot ELISA for detection of Leptospira spp in bovine urine samples. American Journal of Veterinary Research. 66 (5), 762-766 (2005).
- Ishmael, F. T., Stellato, C. Principles and applications of polymerase chain reaction: basic science for the practicing physician. Annals of Allergy, Asthma & Immunology: Official Publication of the American College of Allergy, Asthma, & Immunology. 101 (4), 437-443 (2008).
- Boerner, B., Weigelt, W., Buhk, H. J., Castrucci, G., Ludwig, H. A sensitive and specific PCR/Southern blot assay for detection of bovine herpesvirus 4 in calves infected experimentally. Journal of Virological Methods. 83 (1-2), 169-180 (1999).
- Curry, E., Pratt, S. L., Kelley, D. E., Lapin, D. R., Gibbons, J. R. Use of a Combined duplex PCR/Dot-blot assay for more sensitive genetic characterization. Biochemistry Insights. 1, 35-39 (2008).
- Pilatti, M. M., Ferreira, S. d. e. A., de Melo, M. N., de Andrade, A. S. Comparison of PCR methods for diagnosis of canine visceral leishmaniasis in conjunctival swab samples. Research in Veterinary Science. 87 (2), 255-257 (2009).
- Conrads, G., et al. PCR reaction and dot-blot hybridization to monitor the distribution of oral pathogens within plaque samples of periodontally healthy individuals. Journal of Periodontology. 67 (10), 994-1003 (1996).
- Langa, S., et al. Differentiation of Enterococcus faecium from Lactobacillus delbrueckii subsp. bulgaricus and Streptococcus thermophilus strains by PCR and dot-blot hybridisation. International Journal of Food Microbiology. 88 (2-3), 197-200 (2003).
- Francesca, C., Lucilla, I., Marco, F., Giuseppe, C., Marisa, M. Identification of the unculturable bacteria Candidatus arthromitus in the intestinal content of trouts using dot-blot and Southern blot techniques. Veterinary Microbiology. 156 (3-4), 389-394 (2012).
- Arent, Z., Pardyak, L., Dubniewicz, K., Plachno, B. J., Kotula-Balak, M. Leptospira taxonomy: then and now. Medycyna Weterynaryjna. 78 (10), 489-496 (2022).
- Thibeaux, R., et al. Biodiversity of environmental Leptospira: Improving identification and revisiting the diagnosis. Frontiers in Microbiology. 9, 816 (2018).
- Carrillo-Casas, E. M., Hernández-Castro, R., Suárez-Güemes, F., de la Peña-Moctezuma, A. Selection of the internal control gene for real-time quantitative RT-PCR assays in temperature treated Leptospira. Current Microbiology. 56 (6), 539-546 (2008).
- Azali, M. A., Yean Yean, C., Harun, A., Aminuddin Baki A, N. N., Ismail, N. Molecular characterization of Leptospira spp. in environmental samples from North-Eastern Malaysia revealed a pathogenic strain, Leptospira alstonii. Journal of Tropical Medicine. 2016, 2060241 (2016).
- Ahmed, N., et al. Multilocus sequence typing method for identification and genotypic classification of pathogenic Leptospira species. Annals of Clinical Microbiology and Antimicrobials. 5, 28 (2006).
- Bourhy, P., Collet, L., Brisse, S., Picardeau, M. Leptospira mayottensis sp. nov., a pathogenic species of the genus Leptospira isolated from humans. International Journal of Systematic and Evolutionary Microbiology. 64, 4061-4067 (2014).
- Weiss, S., et al. An extended Multilocus Sequence Typing (MLST) scheme for rapid direct typing of Leptospira from clinical samples. PLOS Neglected Tropical Diseases. 10 (9), e0004996 (2016).
- Branger, C., et al. Polymerase chain reaction assay specific for pathogenic Leptospira based on the gene hap1 encoding the hemolysis-associated protein-1. FEMS Microbiology Letters. 243 (2), 437-445 (2005).
- Ren, S. X., et al. Unique physiological and pathogenic features of Leptospira interrogans revealed by whole-genome sequencing. Nature. 422 (6934), 888-893 (2003).
- Picardeau, M., et al. Genome sequence of the saprophyte Leptospira biflexa provides insights into the evolution of Leptospira and the pathogenesis of leptospirosis. PLOS One. 3 (2), e1607 (2008).
- Kafatos, F. C., Jones, C. W., Efstratiadis, A. Determination of nucleic acid sequence homologies and relative concentrations by a dot hybridization procedure. Nucleic Acids Research. 7 (6), 1541-1552 (1979).
- Bhat, A. I., Rao, G. P. Dot-blot hybridization technique. Characterization of Plant Viruses. , 303-321 (2020).
- Yadav, J. P., Batra, K., Singh, Y., Singh, M. Comparative evaluation of indirect-ELISA and Dot-blot assay for serodetection of Mycoplasma gallisepticum and Mycoplasma synoviae antibodies in poultry. Journal of Microbiological Methods. 189, 106317 (2021).
- Malinen, E., Kassinen, A., Rinttilä, T., Palva, A. Comparison of real-time PCR with SYBR Green I or 5′-nuclease assays and dot-blot hybridization with rDNA-targeted oligonucleotide probes in quantification of selected faecal bacteria. Microbiology. 149, 269-277 (2003).
- Wyss, C., et al. Treponema lecithinolyticum sp. nov., a small saccharolytic spirochaete with phospholipase A and C activities associated with periodontal diseases. International Journal of Systematic Bacteriology. 49, 1329-1339 (1999).
- Shah, J. S., I, D. C., Ward, S., Harris, N. S., Ramasamy, R. Development of a sensitive PCR-dot-blot assay to supplement serological tests for diagnosing Lyme disease. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 37 (4), 701-709 (2018).
- Niu, C., Wang, S., Lu, C. Development and evaluation of a dot-blot assay for rapid determination of invasion-associated gene ibeA directly in fresh bacteria cultures of E. coli. Folia microbiologica. 57 (6), 557-561 (2012).
- Wetherall, B. L., McDonald, P. J., Johnson, A. M. Detection of Campylobacter pylori DNA by hybridization with non-radioactive probes in comparison with a 32P-labeled probe. Journal of Medical Microbiology. 26 (4), 257-263 (1988).
- Kolk, A. H., et al. Detection of Mycobacterium tuberculosis in clinical samples by using polymerase chain reaction and a nonradioactive detection system. Journal of Clinical Microbiology. 30 (10), 2567-2575 (1992).
- Scherer, L. C., et al. PCR colorimetric dot-blot assay and clinical pretest probability for diagnosis of Pulmonary Tuberculosis in smear-negative patients. BMC Public Health. 7, 356 (2007).
- Armbruster, D. A., Pry, T. Limit of blank, limit of detection and limit of quantitation. The Clinical Biochemist Reviews. 29, S49-S52 (2008).
- Zhang, Y., Dai, B. Detection of Leptospira by dot-blot hybridization with photobiotin- and 32P-labeled DNA. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (2), 130-132 (1992).
- Terpstra, W. J., Schoone, G. J., ter Schegget, J. Detection of leptospiral DNA by nucleic acid hybridization with 32P- and biotin-labeled probes. Journal of Medical Microbiology. 22 (1), 23-28 (1986).
- Shukla, J., Tuteja, U., Batra, H. V. DNA probes for identification of leptospires and disease diagnosis. The Southeast Asian Journal of Tropical Medicine and Public Health. 35 (2), 346-352 (2004).
- Jiang, N., Jin, B., Dai, B., Zhang, Y. Identification of pathogenic and nonpathogenic leptospires by recombinant probes. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 26 (1), 1-5 (1995).
- Fach, P., Trap, D., Guillou, J. P. Biotinylated probes to detect Leptospira interrogans on dot-blot hybridization or by in situ hybridization. Letters in Applied Microbiology. 12 (5), 171-176 (1991).
- Huang, N., Dai, B. Assay of genomic DNA homology among strains of different virulent leptospira by DNA hybridization. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (2), 122-125 (1992).
- Dong, X., Dai, B., Chai, J. Homology study of leptospires by molecular hybridization. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (1), 1-4 (1992).
- Komminoth, P. Digoxigenin as an alternative probe labeling for in situ hybridization. Diagnostic Molecular Pathology: The American Journal of Surgical Pathology, part B. 1 (2), 142-150 (1992).
- Saengjaruk, P., et al. Diagnosis of human leptospirosis by monoclonal antibody-based antigen detection in urine. Journal of Clinical Microbiology. 40 (2), 480-489 (2002).
- Okuda, M., et al. Enzyme-linked immunosorbent assay for the detection of canine Leptospira antibodies using recombinant OmpL1 protein. The Journal of Veterinary Medical Science. 67 (3), 249-254 (2005).
- Suwimonteerabutr, J., et al. Evaluation of a monoclonal antibody-based dot-blot ELISA for detection of Leptospira spp in bovine urine samples. American Journal of Veterinary Research. 66 (5), 762-766 (2005).
- Kanagavel, M., et al. Peptide-specific monoclonal antibodies of Leptospiral LigA for acute diagnosis of leptospirosis. Scientific reports. 7 (1), 3250 (2017).
- Levett, P. N. Leptospirosis. Clinical Microbiology Reviews. 14 (2), 296-326 (2001).
- Monahan, A. M., Callanan, J. J., Nally, J. E. Proteomic analysis of Leptospira interrogans shed in urine of chronically infected hosts. Infection and Immunity. 76 (11), 4952-4958 (2008).
- Rojas, P., et al. Detection and quantification of leptospires in urine of dogs: a maintenance host for the zoonotic disease leptospirosis. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 29 (10), 1305-1309 (2010).

