Generering af transkriptionsreguleringsvisning af transkriptomiske funktioner til forudsigelsesopgave og mørk biomarkørdetektion på små datasæt
Summary
Her introducerer vi en protokol til konvertering af transkriptomiske data til en mqTrans-visning, der muliggør identifikation af mørke biomarkører. Selvom de ikke udtrykkes differentielt i konventionelle transkriptomiske analyser, udviser disse biomarkører differentiel ekspression i mqTrans-visningen. Tilgangen fungerer som en komplementær teknik til traditionelle metoder og afslører tidligere oversete biomarkører.
Abstract
Transkriptom repræsenterer ekspressionsniveauerne for mange gener i en prøve og har været meget udbredt i biologisk forskning og klinisk praksis. Forskere fokuserede normalt på transkriptomiske biomarkører med differentielle repræsentationer mellem en fænotypegruppe og en kontrolgruppe af prøver. Denne undersøgelse præsenterede en multitask graph-attention network (GAT) læringsramme for at lære de komplekse intergene interaktioner mellem referenceprøverne. En demonstrativ referencemodel blev forududdannet på de sunde prøver (HealthModel), som direkte kunne bruges til at generere den modelbaserede kvantitative transkriptionsreguleringsvisning (mqTrans) af de uafhængige testtranskriptomer. Den genererede mqTrans-visning af transkriptomer blev demonstreret ved forudsigelsesopgaver og mørk biomarkørdetektion. Det opfundne udtryk “mørk biomarkør” stammede fra dens definition af, at en mørk biomarkør viste differentiel repræsentation i mqTrans-visningen, men intet differentielt udtryk i dets oprindelige ekspressionsniveau. En mørk biomarkør blev altid overset i traditionelle biomarkørdetektionsstudier på grund af fraværet af differentiel ekspression. Kildekoden og manualen til rørledningen HealthModelPipe kan downloades fra http://www.healthinformaticslab.org/supp/resources.php.
Introduction
Transkriptom består af ekspressionerne af alle generne i en prøve og kan profileres af teknologier med høj kapacitet som microarray og RNA-seq1. Ekspressionsniveauerne for et gen i et datasæt kaldes en transkriptomisk funktion, og differentiel repræsentation af en transkriptomisk funktion mellem fænotypen og kontrolgrupperne definerer dette gen som en biomarkør for denne fænotype 2,3. Transkriptomiske biomarkører er blevet anvendt i vid udstrækning i undersøgelserne af sygdomsdiagnose4, biologisk mekanisme5 og overlevelsesanalyse 6,7 osv.
Genaktivitetsmønstre i det sunde væv bærer afgørende information om livet 8,9. Disse mønstre giver uvurderlig indsigt og fungerer som ideelle referencer til forståelse af de komplekse udviklingsbaner for godartede lidelser10,11 og dødelige sygdomme12. Gener interagerer med hinanden, og transkriptomer repræsenterer de endelige ekspressionsniveauer efter deres komplicerede interaktioner. Sådanne mønstre formuleres som transkriptionsreguleringsnetværk13 og metabolismenetværk14 osv. Ekspressionerne af messenger-RNA’er (mRNA’er) kan transkriptionelt reguleres af transkriptionsfaktorer (TF’er) og lange intergene ikke-kodende RNA’er (lincRNA’er)15,16,17. Konventionel differentialekspressionsanalyse ignorerede sådanne komplekse geninteraktioner med antagelsen om uafhængighed mellem funktioner18,19.
Nylige fremskridt inden for grafneurale netværk (GNN’er) viser ekstraordinært potentiale i at udtrække vigtig information fra OMIC-baserede data til kræftstudier20, f.eks. identifikation af co-ekspressionsmoduler21. GNN’ernes medfødte kapacitet gør dem ideelle til modellering af de indviklede forhold og afhængigheder mellem gener22,23.
Biomedicinske undersøgelser fokuserer ofte på nøjagtigt at forudsige en fænotype mod kontrolgruppen. Sådanne opgaver er almindeligvis formuleret som binære klassifikationer 24,25,26. Her er de to klasseetiketter typisk kodet som 1 og 0, sandt og falsk, eller endda positivt og negativt27.
Denne undersøgelse havde til formål at tilvejebringe en brugervenlig protokol til generering af transkriptionsreguleringsvisningen (mqTrans) af et transkriptomdatasæt baseret på den forududdannede grafopmærksomhedsnetværk (GAT) referencemodel. Multitask-GAT-rammen fra et tidligere offentliggjort værk26 blev brugt til at transformere transkriptomiske funktioner til mqTrans-funktionerne. Et stort datasæt af sunde transkriptomer fra University of California, Santa Cruz (UCSC) Xena platform28 blev brugt til at prætræne referencemodellen (HealthModel), som kvantitativt målte transkriptionsreglerne fra regulatoriske faktorer (TF’er og lincRNA’er) til mål-mRNA’erne. Den genererede mqTrans-visning kan bruges til at opbygge forudsigelsesmodeller og registrere mørke biomarkører. Denne protokol bruger patientdatasættet for kolonadenocarcinom (COAD) fra The Cancer Genome Atlas (TCGA) database29 som et illustrativt eksempel. I denne sammenhæng kategoriseres patienter i trin I eller II som negative prøver, mens de i trin III eller IV betragtes som positive prøver. Fordelingen af mørke og traditionelle biomarkører på tværs af de 26 TCGA-kræfttyper sammenlignes også.
Beskrivelse af HealthModel-pipelinen
Den metode, der anvendes i denne protokol, er baseret på den tidligere offentliggjorte ramme26 som skitseret i figur 1. Til at begynde med skal brugerne forberede inputdatasættet, indføre det i den foreslåede HealthModel-pipeline og hente mqTrans-funktioner. Detaljerede instruktioner til dataforberedelse findes i afsnit 2 i protokolafsnittet. Derefter har brugerne mulighed for at kombinere mqTrans-funktioner med de originale transkriptomiske funktioner eller kun fortsætte med de genererede mqTrans-funktioner. Det producerede datasæt underkastes derefter en proces til valg af funktioner, hvor brugerne har fleksibiliteten til at vælge deres foretrukne værdi for k i k-fold krydsvalidering til klassificering. Den primære evalueringsmetrik, der anvendes i denne protokol, er nøjagtighed.
HealthModel26 kategoriserer de transkriptomiske egenskaber i tre forskellige grupper: TF (transkriptionsfaktor), lincRNA (langt intergent ikke-kodende RNA) og mRNA (messenger-RNA). TF-funktionerne er defineret ud fra de annoteringer, der er tilgængelige i Human Protein Atlas 30,31. Dette arbejde udnytter annotationerne af lincRNA’er fra GTEx-datasættet32. Gener, der tilhører veje på tredje niveau i KEGG-databasen33, betragtes som mRNA-funktioner. Det er værd at bemærke, at hvis en mRNA-funktion udviser regulatoriske roller for et målgen som dokumenteret i TRRUST-databasen34, omklassificeres det til TF-klassen.
Denne protokol genererer også manuelt de to eksempelfiler for gen-id’erne for regulatoriske faktorer (regulatory_geneIDs.csv) og mål-mRNA (target_geneIDs.csv). Den parvise afstandsmatrix blandt de regulatoriske egenskaber (TF’er og lincRNA’er) beregnes ved hjælp af Pearson-korrelationskoefficienterne og grupperes af den populære værktøjsvægtede gen-co-ekspressionsnetværksanalyse (WGCNA)36 (adjacent_matrix.csv). Brugere kan direkte bruge HealthModel-pipelinen sammen med disse eksempelkonfigurationsfiler til at generere mqTrans-visningen af et transkriptomisk datasæt.
Tekniske detaljer om HealthModel
HealthModel repræsenterer de indviklede forhold mellem TF’er og lincRNA’er som en graf, hvor inputfunktionerne tjener som hjørnerne betegnet med V og en inter-vertex kantmatrix udpeget som E. Hver prøve er kendetegnet ved K-regulatoriske træk, symboliseret som VK×1. Specifikt omfattede datasættet 425 TF’er og 375 lincRNA’er, hvilket resulterede i en prøvedimensionalitet på K = 425 + 375 = 800. For at etablere kantmatrixen E anvendte dette arbejde det populære værktøj WGCNA35. Den parvise vægt, der forbinder to hjørner repræsenteret som 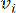 og
og 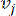 , bestemmes af Pearson-korrelationskoefficienten. Det genregulerende netværk udviser en skalafri topologi36, der er kendetegnet ved tilstedeværelsen af hubgener med centrale funktionelle roller. Vi beregner korrelationen mellem to funktioner eller hjørner
, bestemmes af Pearson-korrelationskoefficienten. Det genregulerende netværk udviser en skalafri topologi36, der er kendetegnet ved tilstedeværelsen af hubgener med centrale funktionelle roller. Vi beregner korrelationen mellem to funktioner eller hjørner 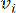 og
og 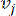 ved hjælp af det topologiske overlapningsmål (TOM) som følger:
ved hjælp af det topologiske overlapningsmål (TOM) som følger:
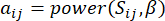 (1)
(1)
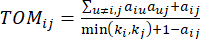 (2)
(2)
Den bløde tærskel β beregnes ved hjælp af funktionen ‘pickSoft Threshold’ fra WGCNA-pakken. Effekteksponentialfunktionen aij anvendes, hvor 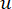 repræsenterer et gen eksklusive i og j og
repræsenterer et gen eksklusive i og j og 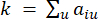 repræsenterer toppunktforbindelsen. WGCNA grupperer ekspressionsprofilerne for de transkriptomiske træk i flere moduler ved hjælp af et almindeligt anvendt ulighedsmål (
repræsenterer toppunktforbindelsen. WGCNA grupperer ekspressionsprofilerne for de transkriptomiske træk i flere moduler ved hjælp af et almindeligt anvendt ulighedsmål (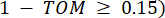 37.
37.
HealthModel-rammen blev oprindeligt designet som en multitask-læringsarkitektur26. Denne protokol bruger kun modelfortræningsopgaven til konstruktion af den transkriptomiske mqTrans-visning. Brugeren kan vælge at forfine den forudtrænede HealthModel yderligere under opmærksomhedsnetværket for multitask-grafer med yderligere opgavespecifikke transkriptomiske prøver.
Tekniske detaljer om valg og klassificering af funktioner
Funktionsvalgspuljen implementerer elleve FS-algoritmer (feature selection). Blandt dem er tre filterbaserede FS-algoritmer: valg af K bedste funktioner ved hjælp af den maksimale informationskoefficient (SK_mic), valg af K-funktioner baseret på FPR for MIC (SK_fpr) og valg af K-funktioner med den højeste falske opdagelsesrate for MIC (SK_fdr). Derudover vurderer tre træbaserede FS-algoritmer individuelle funktioner ved hjælp af et beslutningstræ med Gini-indekset (DT_gini), adaptive boostede beslutningstræer (AdaBoost) og tilfældig skov (RF_fs). Puljen indeholder også to indpakningsmetoder: Rekursiv funktionseliminering med lineær støttevektorklassifikator (RFE_SVC) og eliminering af rekursiv funktion med den logistiske regressionsklassifikator (RFE_LR). Endelig er to integreringsalgoritmer inkluderet: lineær SVC-klassifikator med de højest rangerede L1-funktionsvigtighedsværdier (lSVC_L1) og logistisk regressionsklassifikator med de højest rangerede L1-funktionsvigtighedsværdier (LR_L1).
Klassifikatorpuljen anvender syv forskellige klassifikatorer til at opbygge klassificeringsmodeller. Disse klassifikatorer omfatter lineær støttevektormaskine (SVC), Gaussisk naiv Bayes (GNB), logistisk regressionsklassifikator (LR), k-nærmeste nabo, med k indstillet til 5 som standard (KNN), XGBoost, tilfældig skov (RF) og beslutningstræ (DT).
Den tilfældige opdeling af datasættet i toget: testundersæt kan indstilles på kommandolinjen. Det demonstrerede eksempel bruger forholdet mellem tog: test = 8: 2.
Protocol
Representative Results
Discussion
Afsnit 2 (Brug den forudtrænede HealthModel til at generere mqTrans-funktionerne) i protokollen er det mest kritiske trin i denne protokol. Efter forberedelse af det beregningsmæssige arbejdsmiljø i afsnit 1 genererer afsnit 2 mqTrans-visningen af et transkriptomisk datasæt baseret på den forududdannede store referencemodel. Afsnit 3 er et demonstrativt eksempel på valg af de genererede mqTrans-funktioner til biomarkørdetektioner og forudsigelsesopgaver. Brugerne kan udføre andre transkriptomiske analyser på det…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Dette arbejde blev støttet af Senior og Junior Technological Innovation Team (20210509055RQ), Guizhou Provincial Science and Technology Projects (ZK2023-297), Science and Technology Foundation of Health Commission of Guizhou Province (gzwkj2023-565), Science and Technology Project of Education Department of Jilin Province (JJKH20220245KJ og JJKH20220226SK), National Natural Science Foundation of China (U19A2061), Jilin Provincial Key Laboratory of Big Data Intelligent Computing (20180622002JC) og grundforskningsfondene for de centrale universiteter, JLU. Vi udtrykker vores dybeste påskønnelse til anmeldelsesredaktøren og de tre anonyme korrekturlæsere for deres konstruktive kritik, som har været medvirkende til væsentligt at forbedre strengheden og klarheden i denne protokol.
Materials
| Anaconda | Anaconda | version 2020.11 | Python programming platform |
| Computer | N/A | N/A | Any general-purpose computers satisfy the requirement |
| GPU card | N/A | N/A | Any general-purpose GPU cards with the CUDA computing library |
| pytorch | Pytorch | version 1.13.1 | Software |
| torch-geometric | Pytorch | version 2.2.0 | Software |
Riferimenti
- Mutz, K. -. O., Heilkenbrinker, A., Lönne, M., Walter, J. -. G., Stahl, F. Transcriptome analysis using next-generation sequencing. Curr Opin in Biotechnol. 24 (1), 22-30 (2013).
- Meng, G., Tang, W., Huang, E., Li, Z., Feng, H. A comprehensive assessment of cell type-specific differential expression methods in bulk data. Brief Bioinform. 24 (1), 516 (2023).
- Iqbal, N., Kumar, P. Integrated COVID-19 Predictor: Differential expression analysis to reveal potential biomarkers and prediction of coronavirus using RNA-Seq profile data. Comput Biol Med. 147, 105684 (2022).
- Ravichandran, S., et al. VB(10), a new blood biomarker for differential diagnosis and recovery monitoring of acute viral and bacterial infections. EBioMedicine. 67, 103352 (2021).
- Lv, J., et al. Targeting FABP4 in elderly mice rejuvenates liver metabolism and ameliorates aging-associated metabolic disorders. Metabolism. 142, 155528 (2023).
- Cruz, J. A., Wishart, D. S. Applications of machine learning in cancer prediction and prognosis. Cancer Inform. 2, 59-77 (2007).
- Cox, D. R. . Analysis of Survival Data. , (2018).
- Newman, A. M., et al. Robust enumeration of cell subsets from tissue expression profiles. Nat Methods. 12 (5), 453-457 (2015).
- Ramilowski, J. A., et al. A draft network of ligand-receptor-mediated multicellular signalling in human. Nat Commun. 6 (1), 7866 (2015).
- Xu, Y., et al. MiR-145 detection in urinary extracellular vesicles increase diagnostic efficiency of prostate cancer based on hydrostatic filtration dialysis method. Prostate. 77 (10), 1167-1175 (2017).
- Wang, Y., et al. Profiles of differential expression of circulating microRNAs in hepatitis B virus-positive small hepatocellular carcinoma. Cancer Biomark. 15 (2), 171-180 (2015).
- Hu, S., et al. Transcriptional response profiles of paired tumor-normal samples offer novel. Oncotarget. 8 (25), 41334-41347 (2017).
- Xu, H., Luo, D., Zhang, F. DcWRKY75 promotes ethylene induced petal senescence in carnation (Dianthus caryophyllus L). Plant J. 108 (5), 1473-1492 (2021).
- Niu, H., et al. Dynamic role of Scd1 gene during mouse oocyte growth and maturation. Int J Biol Macromol. 247, 125307 (2023).
- Aznaourova, M., et al. Single-cell RNA sequencing uncovers the nuclear decoy lincRNA PIRAT as a regulator of systemic monocyte immunity during COVID-19. Proc Natl Acad Sci U S A. 119 (36), 2120680119 (2022).
- Prakash, A., Banerjee, M. An interpretable block-attention network for identifying regulatory feature interactions. Brief Bioinform. 24 (4), (2023).
- Zhai, Y., et al. Single-cell RNA sequencing integrated with bulk RNA sequencing analysis reveals diagnostic and prognostic signatures and immunoinfiltration in gastric cancer. Comput Biol Med. 163, 107239 (2023).
- Duan, L., et al. Dynamic changes in spatiotemporal transcriptome reveal maternal immune dysregulation of autism spectrum disorder. Comput Biol Med. 151, 106334 (2022).
- Zolotareva, O., et al. Flimma: a federated and privacy-aware tool for differential gene expression analysis). Genome Biol. 22 (1), 338 (2021).
- Su, R., Zhu, Y., Zou, Q., Wei, L. Distant metastasis identification based on optimized graph representation of gene. Brief Bioinform. 23 (1), (2022).
- Xing, X., et al. Multi-level attention graph neural network based on co-expression gene modules for disease diagnosis and prognosis. Bioinformatics. 38 (8), 2178-2186 (2022).
- Bongini, P., Pancino, N., Scarselli, F., Bianchini, M. . BioGNN: How Graph Neural Networks Can Solve Biological Problems. Artificial Intelligence and Machine Learning for Healthcare: Vol. 1: Image and Data Analytics. , (2022).
- Muzio, G., O’Bray, L., Borgwardt, K. Biological network analysis with deep learning. Brief Bioinform. 22 (2), 1515-1530 (2021).
- Luo, H., et al. Multi-omics integration for disease prediction via multi-level graph attention network and adaptive fusion. bioRxiv. , (2023).
- Feng, X., et al. Selecting multiple biomarker subsets with similarly effective binary classification performances. J Vis Exp. (140), e57738 (2018).
- Duan, M., et al. Orchestrating information across tissues via a novel multitask GAT framework to improve quantitative gene regulation relation modeling for survival analysis. Brief Bioinform. 24 (4), (2023).
- Chicco, D., Starovoitov, V., Jurman, G. The benefits of the Matthews correlation Coefficient (MCC) over the diagnostic odds ratio (DOR) in binary classification assessment. IEEE Access. 9, 47112-47124 (2021).
- Goldman, M. J., et al. Visualizing and interpreting cancer genomics data via the Xena platform. Nat Biotechnol. 38 (6), 675-678 (2020).
- Liu, J., et al. An integrated TCGA pan-cancer clinical data resource to drive high-quality survival outcome analytics. Cell. 173 (2), 400-416 (2018).
- Uhlen, M., et al. Towards a knowledge-based human protein atlas. Nat Biotechnol. 28 (12), 1248-1250 (2010).
- Hernaez, M., Blatti, C., Gevaert, O. Comparison of single and module-based methods for modeling gene regulatory. Bioinformatics. 36 (2), 558-567 (2020).
- Consortium, G. The genotype-tissue expression (GTEx) project. Nat Genet. 45 (6), 580-585 (2013).
- Kanehisa, M., et al. KEGG for taxonomy-based analysis of pathways and genomes. Nucleic Acids Res. 51, D587-D592 (2023).
- Han, H., et al. TRRUST v2: an expanded reference database of human and mouse transcriptional. Nucleic Acids Res. 46, D380-D386 (2018).
- Langfelder, P., Horvath, S. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics. 9, 559 (2008).
- Sulaimanov, N., et al. Inferring gene expression networks with hubs using a degree weighted Lasso. Bioinformatics. 35 (6), 987-994 (2019).
- Kogelman, L. J. A., Kadarmideen, H. N. Weighted Interaction SNP Hub (WISH) network method for building genetic networks. BMC Syst Biol. 8, 5 (2014).
- Duan, M., et al. Pan-cancer identification of the relationship of metabolism-related differentially expressed transcription regulation with non-differentially expressed target genes via a gated recurrent unit network. Comput Biol Med. 148, 105883 (2022).
- Duan, M., et al. Detection and independent validation of model-based quantitative transcriptional regulation relationships altered in lung cancers. Front Bioeng Biotechnol. 8, 582 (2020).
- Fiorini, N., Lipman, D. J., Lu, Z. Towards PubMed 2.0. eLife. 6, 28801 (2017).
- Liu, J., et al. Maternal microbiome regulation prevents early allergic airway diseases in mouse offspring. Pediatr Allergy Immunol. 31 (8), 962-973 (2020).
- Childs, E. J., et al. Association of common susceptibility variants of pancreatic cancer in higher-risk patients: A PACGENE study. Cancer Epidemiol Biomarkers Prev. 25 (7), 1185-1191 (2016).
- Wang, C., et al. Thailandepsins: bacterial products with potent histone deacetylase inhibitory activities and broad-spectrum antiproliferative activities. J Nat Prod. 74 (10), 2031-2038 (2011).
- Lv, X., et al. Transcriptional dysregulations of seven non-differentially expressed genes as biomarkers of metastatic colon cancer. Genes (Basel). 14 (6), 1138 (2023).
- Li, X., et al. Undifferentially expressed CXXC5 as a transcriptionally regulatory biomarker of breast cancer. Advanced Biology. , (2023).
- Yuan, W., et al. The N6-methyladenosine reader protein YTHDC2 promotes gastric cancer progression via enhancing YAP mRNA translation. Transl Oncol. 16, 101308 (2022).
- Tanabe, A., et al. RNA helicase YTHDC2 promotes cancer metastasis via the enhancement of the efficiency by which HIF-1α mRNA is translated. Cancer Lett. 376 (1), 34-42 (2016).

