Generera transkriptionsregleringsvyn för transkriptomiska funktioner för prediktionsuppgift och detektering av mörka biomarkörer på små datamängder
Summary
Här introducerar vi ett protokoll för att konvertera transkriptomiska data till en mqTrans-vy, vilket möjliggör identifiering av mörka biomarkörer. Även om dessa biomarkörer inte uttrycks differentiellt i konventionella transkriptomiska analyser, uppvisar de differentiellt uttryck i mqTrans-vyn. Tillvägagångssättet fungerar som en kompletterande teknik till traditionella metoder och avslöjar tidigare förbisedda biomarkörer.
Abstract
Transkriptom representerar uttrycksnivåerna för många gener i ett prov och har använts i stor utsträckning inom biologisk forskning och klinisk praxis. Forskare fokuserade vanligtvis på transkriptomiska biomarkörer med differentiella representationer mellan en fenotypgrupp och en kontrollgrupp av prover. Denna studie presenterade ett multitask graph-attention network (GAT) inlärningsramverk för att lära sig de komplexa intergena interaktionerna mellan referensproverna. En demonstrativ referensmodell tränades i förväg på de friska proverna (HealthModel), som direkt kunde användas för att generera den modellbaserade kvantitativa transkriptionsregleringen (mqTrans) av de oberoende testtranskriptomen. Den genererade mqTrans-vyn av transkriptom demonstrerades genom prediktionsuppgifter och detektion av mörka biomarkörer. Den myntade termen “mörk biomarkör” härrörde från dess definition att en mörk biomarkör visade differentiell representation i mqTrans-vyn men inget differentiellt uttryck i sin ursprungliga uttrycksnivå. En mörk biomarkör har alltid förbisetts i traditionella biomarkördetektionsstudier på grund av frånvaron av differentiellt uttryck. Källkoden och manualen för pipelinen HealthModelPipe kan laddas ner från http://www.healthinformaticslab.org/supp/resources.php.
Introduction
Transkriptom består av uttrycken av alla gener i ett prov och kan profileras med hjälp av tekniker med hög genomströmning som mikroarray och RNA-seq1. Uttrycksnivåerna för en gen i en datauppsättning kallas en transkriptomisk egenskap, och den differentiella representationen av en transkriptomisk egenskap mellan fenotypen och kontrollgruppen definierar denna gen som en biomarkör för denna fenotyp 2,3. Transkriptomiska biomarkörer har använts i stor utsträckning i undersökningar av sjukdomsdiagnos4, biologisk mekanism5 och överlevnadsanalys 6,7, etc.
Genaktivitetsmönster i de friska vävnaderna bär på avgörande information om livet 8,9. Dessa mönster ger ovärderliga insikter och fungerar som idealiska referenser för att förstå de komplexa utvecklingsbanorna för godartade sjukdomar10,11 och dödliga sjukdomar12. Gener interagerar med varandra, och transkriptom representerar de slutliga uttrycksnivåerna efter deras komplicerade interaktioner. Sådana mönster är formulerade som transkriptionellt regleringsnätverk13 och metabolismnätverk14, etc. Uttrycken av budbärar-RNA (mRNA) kan transkriptionellt regleras av transkriptionsfaktorer (TF) och långa intergena icke-kodande RNA (lincRNA)15,16,17. Konventionell differentiell uttrycksanalys ignorerade sådana komplexa geninteraktioner med antagandet om oberoende mellan egenskaper18,19.
De senaste framstegen inom grafneurala nätverk (GNN) visar en extraordinär potential när det gäller att extrahera viktig information från OMIC-baserade data för cancerstudier20, t.ex. genom att identifiera kouttrycksmoduler21. GNN:s medfödda kapacitet gör dem idealiska för att modellera de intrikata relationerna och beroendena mellan gener22,23.
Biomedicinska studier fokuserar ofta på att exakt förutsäga en fenotyp mot kontrollgruppen. Sådana uppgifter formuleras vanligtvis som binära klassificeringar 24,25,26. Här kodas de två klassetiketterna vanligtvis som 1 och 0, sant och falskt, eller till och med positivt och negativt27.
Denna studie syftade till att tillhandahålla ett lättanvänt protokoll för att generera transkriptionsregleringen (mqTrans) av en transkriptomdatauppsättning baserad på den förtränade referensmodellen för graf-uppmärksamhetsnätverk (GAT). Multitask-GAT-ramverket från ett tidigare publicerat arbete26 användes för att transformera transkriptomiska funktioner till mqTrans-funktionerna. En stor datamängd av friska transkriptom från University of California, Santa Cruz (UCSC) Xena-plattform28 användes för att förträna referensmodellen (HealthModel), som kvantitativt mätte transkriptionsreglerna från de regulatoriska faktorerna (TF och lincRNA) till mål-mRNA. Den genererade mqTrans-vyn kan användas för att bygga prediktionsmodeller och upptäcka mörka biomarkörer. Detta protokoll använder patientdatasetet colon adenocarcinom (COAD) från databasen The Cancer Genome Atlas (TCGA)29 som ett illustrativt exempel. I detta sammanhang kategoriseras patienter i stadium I eller II som negativa prover, medan de i stadium III eller IV betraktas som positiva prover. Fördelningen av mörka och traditionella biomarkörer mellan de 26 TCGA-cancertyperna jämförs också.
Beskrivning av HealthModel-pipelinen
Den metod som används i detta protokoll bygger på det tidigare offentliggjorda ramverket26, som beskrivs i figur 1. Till att börja med måste användarna förbereda indatauppsättningen, mata in den i den föreslagna HealthModel-pipelinen och hämta mqTrans-funktioner. Detaljerade instruktioner för förberedelse av data finns i avsnitt 2 i protokollavsnittet. Därefter har användarna möjlighet att kombinera mqTrans-funktioner med de ursprungliga transkriptomiska funktionerna eller endast fortsätta med de genererade mqTrans-funktionerna. Den producerade datauppsättningen utsätts sedan för en funktionsvalsprocess, där användarna har flexibiliteten att välja önskat värde för k i k-faldig korsvalidering för klassificering. Det primära utvärderingsmåttet som används i detta protokoll är noggrannhet.
HealthModel26 kategoriserar de transkriptomiska egenskaperna i tre distinkta grupper: TF (transkriptionsfaktor), lincRNA (långt intergent icke-kodande RNA) och mRNA (budbärar-RNA). TF-egenskaperna definieras baserat på de annoteringar som finns tillgängliga i Human Protein Atlas30,31. Detta arbete använder annoteringar av lincRNA från GTEx-datasetet32. Gener som tillhör den tredje nivåns vägar i KEGG-databasen33 betraktas som mRNA-egenskaper. Det är värt att notera att om en mRNA-funktion uppvisar reglerande roller för en målgen som dokumenterats i TRRUST-databasen34, omklassificeras den till TF-klassen.
Detta protokoll genererar också manuellt de två exempelfilerna för gen-ID:n för regulatoriska faktorer (regulatory_geneIDs.csv) och mål-mRNA (target_geneIDs.csv). Den parvisa avståndsmatrisen mellan de regulatoriska egenskaperna (TF och lincRNA) beräknas med hjälp av Pearsons korrelationskoefficienter och klustras med hjälp av den populära verktygsviktade nätverksanalysen för genuttryck (WGCNA)36 (adjacent_matrix.csv). Användare kan direkt använda HealthModel-pipelinen tillsammans med dessa exempelkonfigurationsfiler för att generera mqTrans-vyn för en transkriptomisk datauppsättning.
Teknisk information om HealthModel
HealthModel representerar de intrikata relationerna mellan TF och lincRNA som en graf, där indatafunktionerna fungerar som hörnen som betecknas med V och en gränsmatris mellan hörnen som betecknas som E. Varje prov kännetecknas av K-reglerande egenskaper, symboliserade som VK×1. Specifikt omfattade datauppsättningen 425 TF och 375 lincRNA, vilket resulterade i en provdimensionalitet på K = 425 + 375 = 800. För att fastställa kantmatrisen E användes det populära verktyget WGCNA35 i detta arbete. Den parvisa vikten som länkar två hörn representerade som 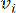 och
och 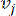 , bestäms av Pearsons korrelationskoefficient. Det genreglerande nätverket uppvisar en skalfri topologi36, kännetecknad av närvaron av navgener med centrala funktionella roller. Vi beräknar korrelationen mellan två objekt eller hörn
, bestäms av Pearsons korrelationskoefficient. Det genreglerande nätverket uppvisar en skalfri topologi36, kännetecknad av närvaron av navgener med centrala funktionella roller. Vi beräknar korrelationen mellan två objekt eller hörn 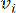 , och
, och 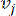 , med hjälp av det topologiska överlappningsmåttet (TOM) enligt följande:
, med hjälp av det topologiska överlappningsmåttet (TOM) enligt följande:
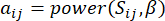 (1)
(1)
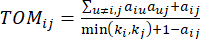 (2)
(2)
Det mjuka tröskelvärdet β beräknas med hjälp av funktionen “pickSoft Threshold” från WGCNA-paketet. Den exponentiella potensfunktionen aij tillämpas, där 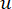 representerar en gen exklusive i och j, och
representerar en gen exklusive i och j, och 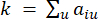 representerar vertexkonnektiviteten. WGCNA klustrar uttrycksprofilerna för de transkriptomiska funktionerna i flera moduler med hjälp av ett vanligt olikhetsmått (
representerar vertexkonnektiviteten. WGCNA klustrar uttrycksprofilerna för de transkriptomiska funktionerna i flera moduler med hjälp av ett vanligt olikhetsmått (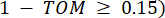 37.
37.
HealthModel-ramverket utformades ursprungligen som en arkitektur för multitasking-inlärning26. Det här protokollet använder endast modellens förträningsuppgift för konstruktionen av den transkriptomiska mqTrans-vyn. Användaren kan välja att ytterligare förfina den förtränade HealthModel under multitask-grafens uppmärksamhetsnätverk med ytterligare uppgiftsspecifika transkriptomiska exempel.
Tekniska detaljer om val och klassificering av funktioner
Funktionsvalspoolen implementerar elva FS-algoritmer (feature selection). Bland dem är tre filterbaserade FS-algoritmer: välja K bästa funktioner med hjälp av maximal informationskoefficient (SK_mic), välja K-funktioner baserat på FPR för MIC (SK_fpr) och välja K-funktioner med den högsta falska upptäcktsfrekvensen för MIC (SK_fdr). Dessutom utvärderar tre trädbaserade FS-algoritmer enskilda funktioner med hjälp av ett beslutsträd med Gini-index (DT_gini), adaptiva förstärkta beslutsträd (AdaBoost) och slumpmässig skog (RF_fs). Poolen innehåller också två omslutningsmetoder: Eliminering av rekursiva funktioner med den linjära stödvektorklassificeraren (RFE_SVC) och eliminering av rekursiva funktioner med den logistiska regressionsklassificeraren (RFE_LR). Slutligen ingår två inbäddningsalgoritmer: linjär SVC-klassificerare med de högst rankade L1-funktionsprioritetsvärdena (lSVC_L1) och logistisk regressionsklassificerare med de högst rankade L1-funktionsprioritetsvärdena (LR_L1).
Klassificerarpoolen använder sju olika klassificerare för att skapa klassificeringsmodeller. Dessa klassificerare består av linjär stödvektormaskin (SVC), Gaussisk Naïve Bayes (GNB), logistisk regressionsklassificerare (LR), k-närmaste granne, med k inställt på 5 som standard (KNN), XGBoost, slumpmässig skog (RF) och beslutsträd (DT).
Den slumpmässiga uppdelningen av datauppsättningen i train: testdelmängder kan anges på kommandoraden. I det demonstrerade exemplet används förhållandet mellan train: test = 8: 2.
Protocol
Representative Results
Discussion
Avsnitt 2 (Använd den förtränade HealthModel för att generera mqTrans-funktionerna) i protokollet är det mest kritiska steget i det här protokollet. Efter att ha förberett beräkningsarbetsmiljön i avsnitt 1 genererar avsnitt 2 mqTrans-vyn av en transkriptomisk datauppsättning baserad på den förtränade stora referensmodellen. Avsnitt 3 är ett demonstrativt exempel på hur man väljer de genererade mqTrans-funktionerna för biomarkördetektion och prediktionsuppgifter. Användarna kan utföra andra transkript…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Detta arbete stöddes av Senior and Junior Technological Innovation Team (20210509055RQ), Guizhou Provincial Science and Technology Projects (ZK2023-297), Science and Technology Foundation of Health Commission of Guizhou Province (gzwkj2023-565), Science and Technology Project of Education Department of Jilin Province (JJKH20220245KJ and JJKH20220226SK), National Natural Science Foundation of China (U19A2061), Jilin Provincial Key Laboratory of Big Data Intelligent Computing (20180622002JC) och Grundforskningsfonderna för de centrala universiteten, JLU. Vi vill framföra vår uppriktiga uppskattning till recensionsredaktören och de tre anonyma granskarna för deras konstruktiva kritik, som har varit avgörande för att avsevärt förbättra noggrannheten och tydligheten i detta protokoll.
Materials
| Anaconda | Anaconda | version 2020.11 | Python programming platform |
| Computer | N/A | N/A | Any general-purpose computers satisfy the requirement |
| GPU card | N/A | N/A | Any general-purpose GPU cards with the CUDA computing library |
| pytorch | Pytorch | version 1.13.1 | Software |
| torch-geometric | Pytorch | version 2.2.0 | Software |
Riferimenti
- Mutz, K. -. O., Heilkenbrinker, A., Lönne, M., Walter, J. -. G., Stahl, F. Transcriptome analysis using next-generation sequencing. Curr Opin in Biotechnol. 24 (1), 22-30 (2013).
- Meng, G., Tang, W., Huang, E., Li, Z., Feng, H. A comprehensive assessment of cell type-specific differential expression methods in bulk data. Brief Bioinform. 24 (1), 516 (2023).
- Iqbal, N., Kumar, P. Integrated COVID-19 Predictor: Differential expression analysis to reveal potential biomarkers and prediction of coronavirus using RNA-Seq profile data. Comput Biol Med. 147, 105684 (2022).
- Ravichandran, S., et al. VB(10), a new blood biomarker for differential diagnosis and recovery monitoring of acute viral and bacterial infections. EBioMedicine. 67, 103352 (2021).
- Lv, J., et al. Targeting FABP4 in elderly mice rejuvenates liver metabolism and ameliorates aging-associated metabolic disorders. Metabolism. 142, 155528 (2023).
- Cruz, J. A., Wishart, D. S. Applications of machine learning in cancer prediction and prognosis. Cancer Inform. 2, 59-77 (2007).
- Cox, D. R. . Analysis of Survival Data. , (2018).
- Newman, A. M., et al. Robust enumeration of cell subsets from tissue expression profiles. Nat Methods. 12 (5), 453-457 (2015).
- Ramilowski, J. A., et al. A draft network of ligand-receptor-mediated multicellular signalling in human. Nat Commun. 6 (1), 7866 (2015).
- Xu, Y., et al. MiR-145 detection in urinary extracellular vesicles increase diagnostic efficiency of prostate cancer based on hydrostatic filtration dialysis method. Prostate. 77 (10), 1167-1175 (2017).
- Wang, Y., et al. Profiles of differential expression of circulating microRNAs in hepatitis B virus-positive small hepatocellular carcinoma. Cancer Biomark. 15 (2), 171-180 (2015).
- Hu, S., et al. Transcriptional response profiles of paired tumor-normal samples offer novel. Oncotarget. 8 (25), 41334-41347 (2017).
- Xu, H., Luo, D., Zhang, F. DcWRKY75 promotes ethylene induced petal senescence in carnation (Dianthus caryophyllus L). Plant J. 108 (5), 1473-1492 (2021).
- Niu, H., et al. Dynamic role of Scd1 gene during mouse oocyte growth and maturation. Int J Biol Macromol. 247, 125307 (2023).
- Aznaourova, M., et al. Single-cell RNA sequencing uncovers the nuclear decoy lincRNA PIRAT as a regulator of systemic monocyte immunity during COVID-19. Proc Natl Acad Sci U S A. 119 (36), 2120680119 (2022).
- Prakash, A., Banerjee, M. An interpretable block-attention network for identifying regulatory feature interactions. Brief Bioinform. 24 (4), (2023).
- Zhai, Y., et al. Single-cell RNA sequencing integrated with bulk RNA sequencing analysis reveals diagnostic and prognostic signatures and immunoinfiltration in gastric cancer. Comput Biol Med. 163, 107239 (2023).
- Duan, L., et al. Dynamic changes in spatiotemporal transcriptome reveal maternal immune dysregulation of autism spectrum disorder. Comput Biol Med. 151, 106334 (2022).
- Zolotareva, O., et al. Flimma: a federated and privacy-aware tool for differential gene expression analysis). Genome Biol. 22 (1), 338 (2021).
- Su, R., Zhu, Y., Zou, Q., Wei, L. Distant metastasis identification based on optimized graph representation of gene. Brief Bioinform. 23 (1), (2022).
- Xing, X., et al. Multi-level attention graph neural network based on co-expression gene modules for disease diagnosis and prognosis. Bioinformatics. 38 (8), 2178-2186 (2022).
- Bongini, P., Pancino, N., Scarselli, F., Bianchini, M. . BioGNN: How Graph Neural Networks Can Solve Biological Problems. Artificial Intelligence and Machine Learning for Healthcare: Vol. 1: Image and Data Analytics. , (2022).
- Muzio, G., O’Bray, L., Borgwardt, K. Biological network analysis with deep learning. Brief Bioinform. 22 (2), 1515-1530 (2021).
- Luo, H., et al. Multi-omics integration for disease prediction via multi-level graph attention network and adaptive fusion. bioRxiv. , (2023).
- Feng, X., et al. Selecting multiple biomarker subsets with similarly effective binary classification performances. J Vis Exp. (140), e57738 (2018).
- Duan, M., et al. Orchestrating information across tissues via a novel multitask GAT framework to improve quantitative gene regulation relation modeling for survival analysis. Brief Bioinform. 24 (4), (2023).
- Chicco, D., Starovoitov, V., Jurman, G. The benefits of the Matthews correlation Coefficient (MCC) over the diagnostic odds ratio (DOR) in binary classification assessment. IEEE Access. 9, 47112-47124 (2021).
- Goldman, M. J., et al. Visualizing and interpreting cancer genomics data via the Xena platform. Nat Biotechnol. 38 (6), 675-678 (2020).
- Liu, J., et al. An integrated TCGA pan-cancer clinical data resource to drive high-quality survival outcome analytics. Cell. 173 (2), 400-416 (2018).
- Uhlen, M., et al. Towards a knowledge-based human protein atlas. Nat Biotechnol. 28 (12), 1248-1250 (2010).
- Hernaez, M., Blatti, C., Gevaert, O. Comparison of single and module-based methods for modeling gene regulatory. Bioinformatics. 36 (2), 558-567 (2020).
- Consortium, G. The genotype-tissue expression (GTEx) project. Nat Genet. 45 (6), 580-585 (2013).
- Kanehisa, M., et al. KEGG for taxonomy-based analysis of pathways and genomes. Nucleic Acids Res. 51, D587-D592 (2023).
- Han, H., et al. TRRUST v2: an expanded reference database of human and mouse transcriptional. Nucleic Acids Res. 46, D380-D386 (2018).
- Langfelder, P., Horvath, S. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics. 9, 559 (2008).
- Sulaimanov, N., et al. Inferring gene expression networks with hubs using a degree weighted Lasso. Bioinformatics. 35 (6), 987-994 (2019).
- Kogelman, L. J. A., Kadarmideen, H. N. Weighted Interaction SNP Hub (WISH) network method for building genetic networks. BMC Syst Biol. 8, 5 (2014).
- Duan, M., et al. Pan-cancer identification of the relationship of metabolism-related differentially expressed transcription regulation with non-differentially expressed target genes via a gated recurrent unit network. Comput Biol Med. 148, 105883 (2022).
- Duan, M., et al. Detection and independent validation of model-based quantitative transcriptional regulation relationships altered in lung cancers. Front Bioeng Biotechnol. 8, 582 (2020).
- Fiorini, N., Lipman, D. J., Lu, Z. Towards PubMed 2.0. eLife. 6, 28801 (2017).
- Liu, J., et al. Maternal microbiome regulation prevents early allergic airway diseases in mouse offspring. Pediatr Allergy Immunol. 31 (8), 962-973 (2020).
- Childs, E. J., et al. Association of common susceptibility variants of pancreatic cancer in higher-risk patients: A PACGENE study. Cancer Epidemiol Biomarkers Prev. 25 (7), 1185-1191 (2016).
- Wang, C., et al. Thailandepsins: bacterial products with potent histone deacetylase inhibitory activities and broad-spectrum antiproliferative activities. J Nat Prod. 74 (10), 2031-2038 (2011).
- Lv, X., et al. Transcriptional dysregulations of seven non-differentially expressed genes as biomarkers of metastatic colon cancer. Genes (Basel). 14 (6), 1138 (2023).
- Li, X., et al. Undifferentially expressed CXXC5 as a transcriptionally regulatory biomarker of breast cancer. Advanced Biology. , (2023).
- Yuan, W., et al. The N6-methyladenosine reader protein YTHDC2 promotes gastric cancer progression via enhancing YAP mRNA translation. Transl Oncol. 16, 101308 (2022).
- Tanabe, A., et al. RNA helicase YTHDC2 promotes cancer metastasis via the enhancement of the efficiency by which HIF-1α mRNA is translated. Cancer Lett. 376 (1), 34-42 (2016).

