Konstruktion av metaboliska nätverk ur jämvikt i nano- och mikrometerstora vesiklar
Summary
Vi presenterar ett protokoll för att rekonstituera membranproteiner och kapsla in enzymer och andra vattenlösliga komponenter i lipidvesiklar av submikrometer- och mikrometerstorlek.
Abstract
Vi presenterar en metod för att inkorporera komplexa proteinnätverk i vesiklar, som involverar integrerade membranproteiner, enzymer och fluorescensbaserade sensorer, med hjälp av renade komponenter. Denna metod är relevant för design och konstruktion av bioreaktorer och för studier av komplexa nätverk för metaboliska reaktioner utanför jämvikt. Vi börjar med att rekonstruera (multipla) membranproteiner till stora unilamellära vesiklar (LUVs) enligt ett tidigare utvecklat protokoll. Vi kapslar sedan in en blandning av renade enzymer, metaboliter och fluorescensbaserade sensorer (fluorescerande proteiner eller färgämnen) via freeze-thaw-extrudering och avlägsnar icke-inkorporerade komponenter genom centrifugering och/eller storleksuteslutningskromatografi. Prestandan hos de metaboliska nätverken mäts i realtid genom att övervaka ATP/ADP-förhållandet, metabolitkoncentrationen, internt pH eller andra parametrar genom fluorescensavläsning. Våra membranproteininnehållande vesiklar med en diameter på 100-400 nm kan omvandlas till jätte-unilamellära vesiklar (GUV), med hjälp av befintliga men optimerade procedurer. Metoden gör det möjligt att inkludera lösliga komponenter (enzymer, metaboliter, sensorer) i mikrometerstora vesiklar, vilket ökar volymen av bioreaktorerna med storleksordningar. Det metaboliska nätverket som innehåller GUV:er fångas i mikrofluidiska enheter för analys med optisk mikroskopi.
Introduction
Området bottom-up syntetisk biologi fokuserar på att konstruera (minimala) celler 1,2 och metaboliska bioreaktorer för bioteknologiska 3,4 eller biomedicinska ändamål 5,6,7,8. Konstruktionen av syntetiska celler ger en unik plattform som gör det möjligt för forskare att studera (membran)proteiner under väldefinierade förhållanden som efterliknar dem i naturliga miljöer, vilket möjliggör upptäckt av emergenta egenskaper och dolda biokemiska funktioner hos proteiner och reaktionsnätverk9. Som ett mellansteg mot en autonomt fungerande syntetisk cell utvecklas moduler som fångar upp väsentliga egenskaper hos levande celler såsom metabolisk energibevarande, protein- och lipidsyntes och homeostas. Sådana moduler förbättrar inte bara vår förståelse av livet utan har också potentiella tillämpningar inom områdena medicin8 och bioteknik10.
Transmembranproteiner är kärnan i praktiskt taget alla metaboliska nätverk eftersom de transporterar molekyler in eller ut ur cellen, signalerar och svarar på miljöns kvalitet och spelar många biosyntetiska roller. Således kräver konstruktionen av metaboliska moduler i syntetiska celler i de flesta fall rekonstituering av integrerade och/eller perifera membranproteiner till ett membrandubbelskikt som består av specifika lipider och hög integritet (låg permeabilitet). Hanteringen av dessa membranproteiner är utmanande och kräver specifik kunskap och experimentella färdigheter.
Flera metoder har utvecklats för att rekonstruera membranproteiner i fosfolipidvesiklar, oftast i syfte att studera funktionen11,12, reglering13, kinetiska egenskaper14,15, lipidberoende15,16 och/eller stabilitet17 hos ett specifikt protein. Dessa metoder innefattar snabb utspädning av tvättmedelslösligt protein till vattenhaltigt medium i närvaro av lipider18, avlägsnande av tvättmedel genom inkubation av tvättmedelslösligt protein med detergentdestabiliserade lipidvesiklar och absorption av tvättmedlet eller tvättmedlen på polystyrenpärlor19, eller avlägsnande av rengöringsmedel genom dialys eller storleksuteslutningskromatografi20. Organiska lösningsmedel har använts för att bilda lipidvesiklar, till exempel genom bildandet av olje-vatten-interfaser21, men majoriteten av integrerade membranproteiner inaktiveras när de utsätts för sådana lösningsmedel.
I vårt laboratorium rekonstituerar vi mestadels membranproteiner med tvättmedelsabsorptionsmetoden för att bilda stora unilamellära vesiklar (LUV)19. Denna metod gör det möjligt att samtidigt rekonstituera flera membranproteiner och kapsla in enzymer, metaboliter och sonder22,23 i vesikellumen. De membranproteininnehållande LUV:erna kan omvandlas till jätte-unilamellära vesiklar (GUV) med/utan inkapsling av vattenlösliga komponenter, med hjälp av antingen elektroformation24 eller gelassisterad svullnad25 och specifika förhållanden för att bevara integriteten hos membranproteinerna26.
Denna artikel presenterar ett protokoll för rekonstituering i LUV:er av ett metaboliskt nätverk som är ur jämvikt och som regenererar ATP genom nedbrytning av L-arginin till L-ornitin27. Bildningen av ATP är kopplad till produktionen av glycerol-3-fosfat (G3P), en viktig byggsten för fosfolipidsyntes22,28. Den metaboliska vägen består av två integrerade membranproteiner, en arginin/ornitin (ArcD) och en G3P/Pi-antiporter (GlpT). Dessutom krävs tre lösliga enzymer (ArcA, ArcB, ArcC) för återvinning av ATP, och GlpK används för att omvandla glycerol till glycerol-3-fosfat, med hjälp av ATP från nedbrytningen av L-arginin, se figur 1 för en schematisk översikt över vägen. Detta protokoll representerar en bra utgångspunkt för den framtida konstruktionen av ännu mer komplexa reaktionsnätverk – för syntes av lipider eller proteiner eller delning av celler. Lipidsammansättningen i vesiklarna stöder aktiviteten hos en mängd olika integrerade membranproteiner och har optimerats för transport av olika molekyler in i eller ut ur vesiklarna 27,29,30.
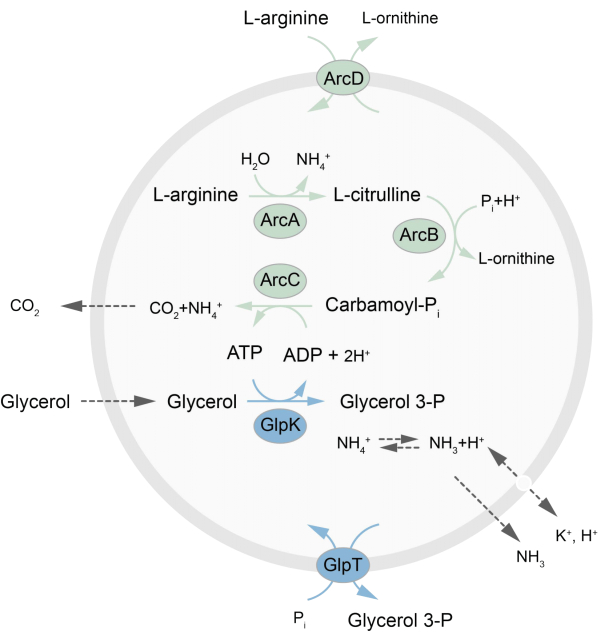
Figur 1: Översikt över vägen för ATP-produktion och syntes och utsöndring av glycerol-3-fosfat. Klicka här för att se en större version av denna figur.
Kort sagt, renade membranproteiner (solubiliserade i dodecyl-β-D-maltosid, DDM) tillsätts till förformade lipidvesiklar som har destabiliserats med Triton X-100, vilket gör det möjligt att föra in proteinerna i membranet. Tvättmedelsmolekylerna avlägsnas därefter (långsamt) genom tillsats av aktiverade polystyrenpärlor, vilket resulterar i bildandet av väl förseglade proteoliposomer. Lösliga komponenter kan sedan tillsättas till vesiklarna och kapslas in via frys-upptiningscykler, vilket fångar molekylerna i processen för membranfusion. De erhållna vesiklarna är mycket heterogena och många är multilamellära vesiklar. De extruderas sedan genom ett polykarbonatfilter med en porstorlek på 400, 200 eller 100 nm, vilket ger mer jämnt dimensionerade vesiklar; Ju mindre porstorlek, desto mer homogena och unilamellära vesiklar men till priset av en mindre inre volym. Icke-inkorporerade proteiner och små molekyler avlägsnas från den externa lösningen genom storleksuteslutningskromatografi. ProteoLUV:erna kan omvandlas till vesiklar i mikrometerstorlek genom gelassisterad svullnad, och dessa proteoGUV:er samlas sedan in och fångas i ett mikrofluidiskt chip för mikroskopisk karakterisering och manipulation. Figur 2 visar en schematisk översikt över det fullständiga protokollet.
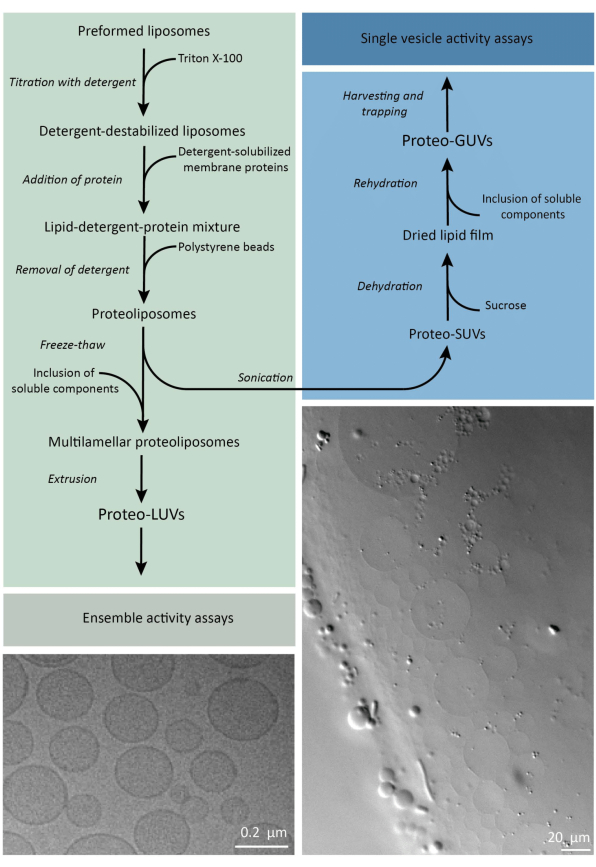
Figur 2: Översikt över protokollet för rekonstituering av membranproteiner och inkapsling av enzymer och vattenlösliga komponenter i lipidvesiklar av submikrometer (LUV) och mikrometerstorlek (GUV). Klicka här för att se en större version av denna figur.
Rekonstituerings- och inkapslingsprotokollen fungerar bra och proteinernas funktionalitet bibehålls, men proteoLUV:erna och proteoGUV:erna är heterogena i storlek. Mikrofluidiska metoder 31,32 tillåter bildandet av mikrometerstora vesiklar som är mer homogena i storlek, men funktionell rekonstitution av membranproteiner är i allmänhet inte möjlig eftersom kvarvarande lösningsmedel i dubbellagret inaktiverar proteinerna. ProteoLUV:erna varierar i storlek från 100 till 400 nm, och vid låga koncentrationer av enzymer kan inkapslingen leda till vesiklar med ofullständiga metaboliska vägar (stokastiska effekter; se figur 3). LUV:er är idealiska för att konstruera specifika metaboliska moduler, som visas här för produktion av ATP och byggstenar som G3P. Sådana proteoLUV:er kan potentiellt kapslas in i GUV:er och fungera som organellliknande fack för värdvesiklarna.
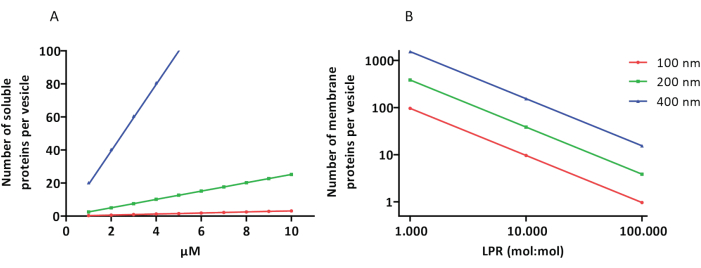
Figur 3: Antal molekyler per vesikel med en diameter på 100, 200 eller 400 nm. (A) När de inkapslade proteinerna (enzymer, sonder) ligger i intervallet 1-10 μM. (B) Beredningen görs vid 1 till 1 000, 1 till 10 000 och 1 till 100 000 membranproteiner per lipid (mol/mol). Vi gör antagandet att molekyler är inkapslade vid de angivna koncentrationerna och inkorporeras i membranet vid dessa protein-till-lipid-förhållanden. För vissa enzymer har vi sett att de binder till membran, vilket kan öka deras skenbara koncentration i vesiklarna. Förkortning: LPR = Lipid-Protein-Ratio Klicka här för att se en större version av denna figur.
Protocol
Representative Results
Discussion
Vi presenterar ett protokoll för syntes av (membran)protein som innehåller lipidvesiklar med submikrometerstorlek (proteoLUVs), och omvandlingen av proteoLUVs till giant-unilamellära vesiklar (proteoGUVs). Protokollet bör vara tillämpligt för rekonstituering av andra membranproteiner 13,19,30,40 och inkapsling av andra metaboliska nätverk än de L-argininnedbrytnings- och glycerol-3-fosfatsyntesvägar som presenteras här.</sup…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Författarna tackar Aditya Iyer för kloningen av pBAD-PercevalHR-genen och Gea Schuurman-Wolters för att ha hjälpt till med proteinproduktion och rening. Forskningen finansierades av NWO Gravitation-programmet “Building a Synthetic Cell” (BaSyC).
Materials
| Agarose | Sigma Aldrich | A9414-25g | |
| Amicon cut-off filter | Sigma Aldrich | Milipore centrifugal filter units Amicon Ultra | |
| BioBeads | BioRad | 152-3920 | |
| CHCl3 | Macron Fine Chemicals | MFCD00000826 | |
| D(+)-Glucose | Formedium | – | |
| D(+)-Sucrose | Formedium | – | |
| DDM | Glycon | D97002 -C | |
| Diethyl Ether | Biosolve | 52805 | |
| DMSO | Sigma-Aldrich | 276855-100ml | |
| DOPC | Avanti | 850375P-1g | |
| DOPE | Avanti | 850725P-1g | |
| DOPG | Avanti | 840475P-1g | |
| DTT | Formedium | DTT005 | |
| EtOH | J.T.Baker Avantor | MFCD00003568 | |
| Extruder | Avestin Inc | LF-1 | |
| Fluorimeter | Jasco | Spectrofluorometer FP-8300 | |
| Glycerol | BOOM | 51171608 | |
| Gravity flow column | Bio-Rad | 732-1010 | |
| Hamilton syringe 100 µL | Hamilton | 7656-01 | |
| Hamilton syringe 1000 µL | Hamilton | 81320 | |
| Handheld LCP dispenser | Art Robbins Instruments | 620-411-00 | |
| Handheld Sonicator | Hielscher Ultrasound Technology | UP50H | |
| HCl | BOOM | x76021889.1000 | |
| Imidazole | Roth | X998.4-250g | |
| K2HPO4 | Supelco | 1.05099.1000 | |
| KCl | BOOM | 76028270.1 | |
| KH2PO4 | Supelco | 1.04873.1000 | |
| Kimwipe | Kimtech Science | 7552 | |
| Large Falcon tube centrifuge | Eppendorf | Centrifuge 5810 R | |
| L-Arginine | Sigma-Aldrich | A5006-100G | |
| Light microscope | Leica | DM LS2 | |
| L-Ornithine | Roth | T204.1 | |
| LSM Laser Scanning Confocal Microscope | Zeiss | LSM 710 ConfoCor 3 | |
| MgCl2 | Sigma-Aldrich | M2670-1KG | |
| Microfluidic chip | Homemade | PDMS based | DOI: https://doi.org/10.1039/C8LC01275J |
| Na-ADP | Sigma-Aldrich | A2754-1G | |
| NaCl | Supelco | 1.06404.1000 | |
| Nanodrop Spectrometer | Isogen Life Science | ND-1000 spectrophotometer NanoDrop | |
| NaOH | Supelco | 1.06498.1000 | |
| Needles for GUVs | Henke-Ject | 14-14575 | 27 G x 3/4'' 0.4 x 20 mm |
| Needles for microfluidics | Henke-Ject | 14-15538 | 18 G x 1 1/2'' 1.2 x 40 mm |
| Ni2+ Sepharose | Cytiva | 17526802 | |
| Nigericin | Sigma-Aldrich | N7143-5MG | |
| Nutator | VWR | 83007-210 | |
| Osmolality meter | Gonotec Salmenkipp | Osmomat 3000 basic freezing point osmometer | |
| Plasmacleaner | Plasma Etch | PE-Avenger | |
| Polycarbonate filter | Cytiva Whatman | Nuclepor Track-Etch Membrane Product: 10417104 | 0.4 µm |
| Polycarbonate ultracentrifuge tube | Beckman Coulter | 355647 | |
| Pyranine | Acros Organics | H1529-1G | |
| Quartz cuvette (black) | Hellma Analytics | 108B-10-40 | |
| Sephadex G-75 resin | GE Healthcare | 17-0050-01 | |
| Sonicator | Sonics Sonics & Materials INC | Sonics vibra cell | |
| Syringe filter | Sarstedt | Filtropur S plus 0.2 | 0.2 µm |
| Syringe pump | Harvard Apparatus | A-42467 | |
| Tabletop centrifuge | Eppendorf | centrifuge 5418 | |
| Teflon spacer | Homemade | Teflon based | 45 x 26 x 1.5 or 45 x 26 x 3 or 20 x 20 x 3 mm |
| Tris | PanReac AppliChem | A1086.1000 | |
| Triton X-100 | Sigma Aldrich | T8787-100 ml | |
| Ultracentrifuge | Beckman Coulter | Optima Max-E | |
| UV lamp | Spectroline | ENB-280C/FE | |
| UV/VIS Spectrometer | Jasco | V730 spectrophotometer | |
| Valinomycin | Sigma-Aldrich | V0627-10MG | |
| Widefield fluorescence microscope | Zeiss | AxioObserver | |
| β-Casein | Sigma Aldrich | C5890-500g |
Riferimenti
- Hirschi, S., Ward, T. R., Meier, W. P., Müller, D. J., Fotiadis, D. Synthetic biology: bottom-up assembly of molecular systems. Chem Rev. 122 (21), 16294-16328 (2022).
- Ivanov, I., et al. Bottom-up synthesis of artificial cells: recent highlights and future challenges. Annu Rev Chem Biomol. Eng. 12 (1), 287-308 (2021).
- Clomburg, J. M., Crumbley, A. M., Gonzalez, R. Industrial biomanufacturing: The future of chemical production. Science. 355 (6320), (2017).
- Shi, T., Han, P., You, C., Zhang, Y. -. H. P. J. An in vitro synthetic biology platform for emerging industrial biomanufacturing: Bottom-up pathway design. Synth Syst Biotechnol. 3 (3), 186-195 (2018).
- Wang, A., et al. Liver-target and glucose-responsive polymersomes toward mimicking endogenous insulin secretion with improved hepatic glucose utilization. Adv Funct Mater. 30 (13), 1910168 (2020).
- Kanter, G., et al. Cell-free production of scFv fusion proteins: an efficient approach for personalized lymphoma vaccines. Blood. 109 (8), 3393-3399 (2007).
- Zeltins, A. Construction and characterization of virus-like particles: a review. Mol Biotechnol. 53 (1), 92-107 (2013).
- Jain, K. K. Synthetic biology and personalized medicine. Med Princ Pract. 22 (3), 209-219 (2013).
- Schwille, P., Frohn, B. P. Hidden protein functions and what they may teach us. Trends Cell Biol. 32 (2), 102-109 (2022).
- Sachsenmeier, P. Industry 5.0-The relevance and implications of bionics and synthetic biology. Ingegneria. 2 (2), 225-229 (2016).
- Schmidt, D., Jiang, Q. -. X., MacKinnon, R. Phospholipids and the origin of cationic gating charges in voltage sensors. Nature. 444 (7120), 775-779 (2006).
- Godoy-Hernandez, A., et al. Rapid and highly stable membrane reconstitution by LAiR enables the study of physiological integral membrane protein functions. ACS Cent Sci. 9 (3), 494-507 (2023).
- Sikkema, H. R., et al. Gating by ionic strength and safety check by cyclic-di-AMP in the ABC transporter OpuA. Sci Adv. 6 (47), 7697 (2020).
- Foucaud, C., Poolman, B. Lactose transport system of Streptococcus thermophilus. Functional reconstitution of the protein and characterization of the kinetic mechanism of transport. J Biol Chem. 267 (31), 22087-22094 (1992).
- Yoneda, J. S., Sebinelli, H. G., Itri, R., Ciancaglini, P. Overview on solubilization and lipid reconstitution of Na,K-ATPase: enzyme kinetic and biophysical characterization. Biophys Rev. 12 (1), 49-64 (2020).
- Simidjiev, I., et al. Self-assembly of large, ordered lamellae from non-bilayer lipids and integral membrane proteins in vitro. Proc Natl Acad Sci. 97 (4), 1473-1476 (2000).
- Harris, N. J., Booth, P. J. Folding and stability of membrane transport proteins in vitro. Biochim Biophys Acta BBA – Biomembr. 1818 (4), 1055-1066 (2012).
- Jackson, M. L., Litman, B. J. Rhodopsin-egg phosphatidylcholine reconstitution by an octyl glucoside dilution procedure. Biochim Biophys Acta BBA – Biomembr. 812 (2), 369-376 (1985).
- Geertsma, E. R., Nik Mahmood, N. A. B., Schuurman-Wolters, G. K., Poolman, B. Membrane reconstitution of ABC transporters and assays of translocator function. Nat Protoc. 3 (2), 256-266 (2008).
- Rigaud, J. -. L., Pitard, B., Levy, D. Reconstitution of membrane proteins into liposomes: application to energy-transducing membrane proteins. Biochim Biophys Acta BBA – Bioenerg. 1231 (3), 223-246 (1995).
- Szoka, F., Papahadjopoulos, D. Procedure for preparation of liposomes with large internal aqueous space and high capture by reverse-phase evaporation. Proc Natl Acad Sci. 75 (9), 4194-4198 (1978).
- . Synthetic Organelles for Energy Conservation and Delivery of Building Blocks for Lipid Biosynthesis Available from: https://www.researchsquare.com/article/rs-3385355/v1 (2023)
- Lee, K. Y., et al. Photosynthetic artificial organelles sustain and control ATP-dependent reactions in a protocellular system. Nat Biotechnol. 36 (6), 530-535 (2018).
- Méléard, P., Bagatolli, L. A., Pott, T. Giant unilamellar vesicle electroformation. Methods in Enzymology. , 161-176 (2009).
- Garten, M., Aimon, S., Bassereau, P., Toombes, G. E. S. Reconstitution of a transmembrane protein, the voltage-gated ion channel, KvAP, into giant unilamellar vesicles for microscopy and patch clamp studies. J. Vis. Exp. (95), e52281 (2015).
- Doeven, M. K., et al. lateral mobility and function of membrane proteins incorporated into giant unilamellar vesicles. Biophys J. 88 (2), 1134-1142 (2005).
- Pols, T., et al. A synthetic metabolic network for physicochemical homeostasis. Nat Commun. 10 (1), 4239 (2019).
- Bailoni, E., Poolman, B. ATP recycling fuels sustainable glycerol 3-phosphate formation in synthetic cells fed by dynamic dialysis. ACS Synth Biol. 11 (7), 2348-2360 (2022).
- Van Der Heide, T. On the osmotic signal and osmosensing mechanism of an ABC transport system for glycine betaine. EMBO J. 20 (24), 7022-7032 (2001).
- Van’T Klooster, J. S., et al. Membrane lipid requirements of the lysine transporter Lyp1 from Saccharomyces cerevisiae. J Mol Biol. 432 (14), 4023-4031 (2020).
- Lou, G., Anderluzzi, G., Woods, S., Roberts, C. W., Perrie, Y. A novel microfluidic-based approach to formulate size-tuneable large unilamellar cationic liposomes: Formulation, cellular uptake and biodistribution investigations. Eur J Pharm Biopharm. 143, 51-60 (2019).
- Weiss, M., et al. Sequential bottom-up assembly of mechanically stabilized synthetic cells by microfluidics. Nat Mater. 17 (1), 89-96 (2018).
- Pols, T., Singh, S., Deelman-Driessen, C., Gaastra, B. F., Poolman, B. Enzymology of the pathway for ATP production by arginine breakdown. FEBS J. 288 (1), 293-309 (2021).
- Yandrapalli, N., Robinson, T. Ultra-high capacity microfluidic trapping of giant vesicles for high-throughput membrane studies. Lab Chip. 19 (4), 626-633 (2019).
- Elias, M., et al. Microfluidic characterization of biomimetic membrane mechanics with an on-chip micropipette. Micro Nano Eng. 8, 100064 (2020).
- Robinson, T., Kuhn, P., Eyer, K., Dittrich, P. S. Microfluidic trapping of giant unilamellar vesicles to study transport through a membrane pore. Biomicrofluidics. 7 (4), 044105 (2013).
- Cooper, A., Girish, V., Subramaniam, A. B. Osmotic Pressure Enables High-Yield Assembly of Giant Vesicles in Solutions of Physiological Ionic Strengths. Langmuir. 39 (15), 5579-5590 (2023).
- Tantama, M., Martínez-François, J. R., Mongeon, R., Yellen, G. Imaging energy status in live cells with a fluorescent biosensor of the intracellular ATP-to-ADP ratio. Nat Commun. 4 (1), 2550 (2013).
- Setyawati, I., et al. In vitro reconstitution of dynamically interacting integral membrane subunits of energy-coupling factor transporters. eLife. 9, e64389 (2020).
- Oropeza-Guzman, E., Ríos-Ramírez, M., Ruiz-Suárez, J. C. Leveraging the coffee ring effect for a defect-free electroformation of giant unilamellar vesicles. Langmuir. 35 (50), 16528-16535 (2019).
- Estes, D. J., Mayer, M. Electroformation of giant liposomes from spin-coated films of lipids. Colloids Surf B Biointerfaces. 42 (2), 115-123 (2005).

