नैनो- और माइक्रोमीटर के आकार के पुटिकाओं में आउट-ऑफ-इक्विलिब्रियम मेटाबोलिक नेटवर्क का निर्माण
Summary
हम झिल्ली प्रोटीन के पुनर्गठन और उप-माइक्रोमीटर और माइक्रोमीटर आकार के लिपिड पुटिकाओं में एंजाइमों और अन्य पानी में घुलनशील घटकों को एनकैप्सुलेट करने के लिए एक प्रोटोकॉल प्रस्तुत करते हैं।
Abstract
हम पुटिकाओं जटिल प्रोटीन नेटवर्क में शामिल करने के लिए एक विधि प्रस्तुत करते हैं, जिसमें शुद्ध घटकों का उपयोग करके अभिन्न झिल्ली प्रोटीन, एंजाइम और प्रतिदीप्ति आधारित सेंसर शामिल होते हैं। यह विधि बायोरिएक्टरों के डिजाइन और निर्माण और जटिल आउट-ऑफ-संतुलन चयापचय प्रतिक्रिया नेटवर्क के अध्ययन के लिए प्रासंगिक है। हम पहले से विकसित प्रोटोकॉल के अनुसार बड़े यूनिलामेलर पुटिकाओं (एलयूवी) में (कई) झिल्ली प्रोटीन का पुनर्गठन करके शुरू करते हैं। फिर हम फ्रीज-पिघलना-एक्सट्रूज़न के माध्यम से शुद्ध एंजाइमों, चयापचयों और प्रतिदीप्ति आधारित सेंसर (फ्लोरोसेंट प्रोटीन या रंजक) के मिश्रण को समाहित करते हैं और सेंट्रीफ्यूजेशन और/या आकार-बहिष्करण क्रोमैटोग्राफी द्वारा गैर-निगमित घटकों को हटाते हैं। चयापचय नेटवर्क के प्रदर्शन को प्रतिदीप्ति रीडआउट द्वारा एटीपी/एडीपी अनुपात, मेटाबोलाइट एकाग्रता, आंतरिक पीएच, या अन्य मापदंडों की निगरानी करके वास्तविक समय में मापा जाता है। 100-400 एनएम व्यास के हमारे झिल्ली प्रोटीन युक्त पुटिकाओं को मौजूदा लेकिन अनुकूलित प्रक्रियाओं का उपयोग करके विशाल-यूनिलामेलर पुटिकाओं (जीयूवी) में परिवर्तित किया जा सकता है। दृष्टिकोण माइक्रोमीटर-आकार के पुटिकाओं में घुलनशील घटकों (एंजाइम, मेटाबोलाइट्स, सेंसर) को शामिल करने में सक्षम बनाता है, इस प्रकार परिमाण के आदेशों द्वारा बायोरिएक्टर की मात्रा को बढ़ाता है। जीयूवी युक्त चयापचय नेटवर्क ऑप्टिकल माइक्रोस्कोपी द्वारा विश्लेषण के लिए माइक्रोफ्लुइडिक उपकरणों में फंस गए हैं।
Introduction
नीचे-ऊपर सिंथेटिक जीव विज्ञान के क्षेत्र (न्यूनतम) कोशिकाओं 1,2 और जैव प्रौद्योगिकी 3,4 या जैव चिकित्सा प्रयोजनों 5,6,7,8 के लिए चयापचय bioreactors के निर्माण पर केंद्रित है. सिंथेटिक कोशिकाओं का निर्माण एक अनूठा मंच प्रदान करता है जो शोधकर्ताओं को देशी वातावरण की नकल करने वाली अच्छी तरह से परिभाषित स्थितियों में प्रोटीन का अध्ययन करने की अनुमति देता है, जिससे प्रोटीन औरप्रतिक्रिया नेटवर्क 9 के आकस्मिक गुणों और छुपा जैव रासायनिक कार्यों की खोज को सक्षम किया जा सकता है। एक स्वायत्त रूप से काम करने वाले सिंथेटिक सेल की दिशा में एक मध्यवर्ती कदम के रूप में, मॉड्यूल विकसित किए जाते हैं जो चयापचय ऊर्जा संरक्षण, प्रोटीन और लिपिड संश्लेषण, और होमियोस्टेसिस जैसे जीवित कोशिकाओं की आवश्यक विशेषताओं को पकड़ते हैं। इस तरह के मॉड्यूल न केवल जीवन की हमारी समझ को बढ़ाने के लिए लेकिन यह भी चिकित्सा8 और जैव प्रौद्योगिकी10 के क्षेत्र में संभावित अनुप्रयोगों है.
ट्रांसमेम्ब्रेन प्रोटीन वस्तुतः किसी भी चयापचय नेटवर्क के केंद्र में होते हैं क्योंकि वे अणुओं को कोशिका के अंदर या बाहर ले जाते हैं, संकेत देते हैं, और पर्यावरण की गुणवत्ता का जवाब देते हैं, और कई बायोसिंथेटिक भूमिकाएं निभाते हैं। इस प्रकार, सिंथेटिक कोशिकाओं में चयापचय मॉड्यूल की इंजीनियरिंग के लिए ज्यादातर मामलों में विशिष्ट लिपिड और उच्च अखंडता (कम पारगम्यता) से बना झिल्ली बाईलेयर में अभिन्न और / या परिधीय झिल्ली प्रोटीन के पुनर्गठन की आवश्यकता होती है। इन झिल्ली प्रोटीन की हैंडलिंग चुनौतीपूर्ण है और विशिष्ट ज्ञान और प्रयोगात्मक कौशल की आवश्यकता है.
फॉस्फोलिपिड पुटिकाओं के भीतर झिल्ली प्रोटीन का पुनर्गठन करने के लिए कई तरीकों का विकास किया गया है, अक्सर समारोह11,12, विनियमन13, गतिज गुण14,15, लिपिड निर्भरता15,16, और/या स्थिरता17 एक विशिष्ट प्रोटीन के अध्ययन के उद्देश्य से। इन विधियों लिपिड18 की उपस्थिति में जलीय मीडिया में डिटर्जेंट घुलनशील प्रोटीन के तेजी से कमजोर पड़ने शामिल, डिटर्जेंट-अस्थिर लिपिड पुटिकाओं और डिटर्जेंट के अवशोषण के साथ डिटर्जेंट-घुलनशील प्रोटीन सेते द्वारा डिटर्जेंट को हटाने(रों) polystyrene मोती19 पर, या डायलिसिस या आकार बहिष्करण क्रोमैटोग्राफी द्वारा डिटर्जेंट को हटाने20. कार्बनिक सॉल्वैंट्स लिपिड पुटिकाओं के रूप में इस्तेमाल किया गया है, उदाहरण के लिए, तेल-पानी interphases21 के गठन के माध्यम से, लेकिन अभिन्न झिल्ली प्रोटीन के बहुमत निष्क्रिय कर रहे हैं जब इस तरह के सॉल्वैंट्स के संपर्क में हैं.
हमारी प्रयोगशाला में, हम ज्यादातर बड़े-यूनिलामेलर पुटिकाओं (एलयूवी)19बनाने के लिए डिटर्जेंट-अवशोषण विधि द्वारा झिल्ली प्रोटीन का पुनर्गठन करते हैं। यह विधि कई झिल्ली प्रोटीन के सह-पुनर्गठन और एंजाइमों, चयापचयों और जांच22,23 के पुटिका लुमेन में एनकैप्सुलेशन की अनुमति देती है। झिल्ली प्रोटीन युक्त LUVs विशाल unilamellar vesicles (GUVs) के साथ / पानी में घुलनशील घटकों के encapsulation के बिना, या तो electroformation24 या जेल सहायता प्राप्त सूजन25 और झिल्ली प्रोटीन26 की अखंडता को संरक्षित करने के लिए विशिष्ट शर्तों का उपयोग कर में परिवर्तित किया जा सकता है.
यह पत्र एक आउट-ऑफ-इक्विलिब्रियम मेटाबोलिक नेटवर्क के एलयूवी में पुनर्गठन के लिए एक प्रोटोकॉल प्रस्तुत करता है जो एल-आर्जिनिन के टूटने के माध्यम से एटीपी को एल-ऑर्निथिन27 में पुन: उत्पन्न करता है। एटीपी का गठन ग्लिसरॉल-3-फॉस्फेट (जी 3 पी) के उत्पादन के लिए युग्मित है, फॉस्फोलिपिड संश्लेषण 22,28के लिए एक महत्वपूर्ण बिल्डिंग ब्लॉक। चयापचय मार्ग में दो अभिन्न झिल्ली प्रोटीन, एक आर्जिनिन / ऑर्निथिन (एआरसीडी) और एक जी 3 पी / पीआई एंटीपोर्टर (जीएलपीटी) होते हैं। इसके अलावा, एटीपी के पुनर्चक्रण के लिए तीन घुलनशील एंजाइम (ArcA, ArcB, ArcC) की आवश्यकता होती है, और GlpK का उपयोग ग्लिसरॉल को ग्लिसरॉल 3-फॉस्फेट में बदलने के लिए किया जाता है, L-arginine के टूटने से ATP का उपयोग करके, मार्ग के योजनाबद्ध अवलोकन के लिए चित्र 1 देखें। यह प्रोटोकॉल लिपिड या प्रोटीन के संश्लेषण या कोशिकाओं के विभाजन के लिए और भी जटिल प्रतिक्रिया नेटवर्क के भविष्य के निर्माण के लिए एक अच्छा प्रारंभिक बिंदु का प्रतिनिधित्व करता है। पुटिकाओं की लिपिड संरचना अभिन्न झिल्ली प्रोटीन की एक विस्तृत विविधता की गतिविधि का समर्थन करती है और पुटिकाओं 27,29,30में या बाहर विविध अणुओं के परिवहन के लिए अनुकूलित किया गया है।
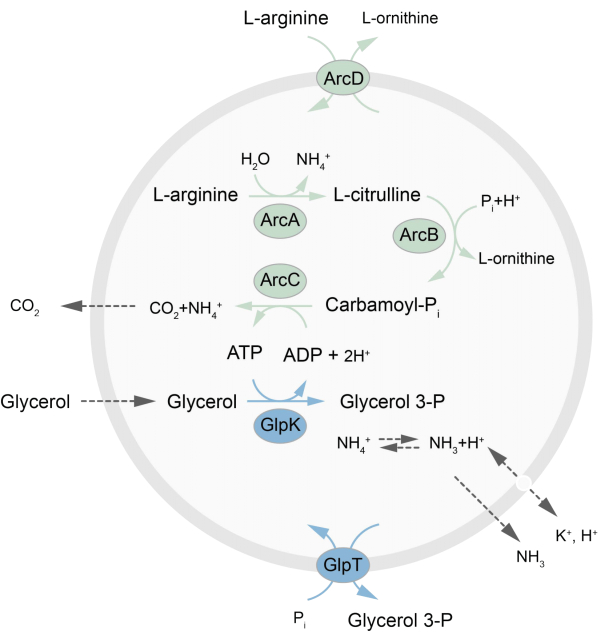
चित्रा 1: एटीपी उत्पादन और ग्लिसरॉल 3-फॉस्फेट संश्लेषण और उत्सर्जन के लिए मार्ग का अवलोकन। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें।
संक्षेप में, शुद्ध झिल्ली प्रोटीन (डोडेसिल-β-डी-माल्टोसाइड, डीडीएम में घुलनशील) को पूर्वनिर्मित लिपिड पुटिकाओं में जोड़ा जाता है जिन्हें ट्राइटन एक्स -100 के साथ अस्थिर किया गया है, जो झिल्ली में प्रोटीन के सम्मिलन की अनुमति देता है। डिटर्जेंट अणुओं को बाद में (धीरे-धीरे) सक्रिय पॉलीस्टायर्न मोतियों के अतिरिक्त हटा दिया जाता है, जिसके परिणामस्वरूप अच्छी तरह से सील प्रोटीओलिपोसोम का निर्माण होता है। घुलनशील घटकों को तब पुटिकाओं में जोड़ा जा सकता है और फ्रीज-पिघलना चक्रों के माध्यम से समझाया जा सकता है, जो झिल्ली संलयन की प्रक्रिया में अणुओं को फँसाता है। प्राप्त पुटिकाएं अत्यधिक विषम होती हैं और कई मल्टीलामेलर होती हैं। फिर उन्हें 400, 200, या 100 एनएम के छिद्र आकार के साथ एक पॉली कार्बोनेट फिल्टर के माध्यम से निकाला जाता है, जो अधिक समान आकार के पुटिकाओं का उत्पादन करता है; छिद्र का आकार जितना छोटा होता है, पुटिकाओं का आकार उतना ही अधिक सजातीय और एकमिलामेलर होता है, लेकिन एक छोटे आंतरिक आयतन की कीमत पर। गैर-निगमित प्रोटीन और छोटे अणुओं को आकार-बहिष्करण क्रोमैटोग्राफी द्वारा बाहरी समाधान से हटा दिया जाता है। प्रोटीओएलयूवी को जेल-असिस्टेड सूजन द्वारा माइक्रोमीटर आकार के पुटिकाओं में परिवर्तित किया जा सकता है, और इन प्रोटीओजीयूवी को तब सूक्ष्म लक्षण वर्णन और हेरफेर के लिए माइक्रोफ्लुइडिक चिप में एकत्र और फंसाया जाता है। चित्रा 2 पूर्ण प्रोटोकॉल का एक योजनाबद्ध अवलोकन दिखाता है।
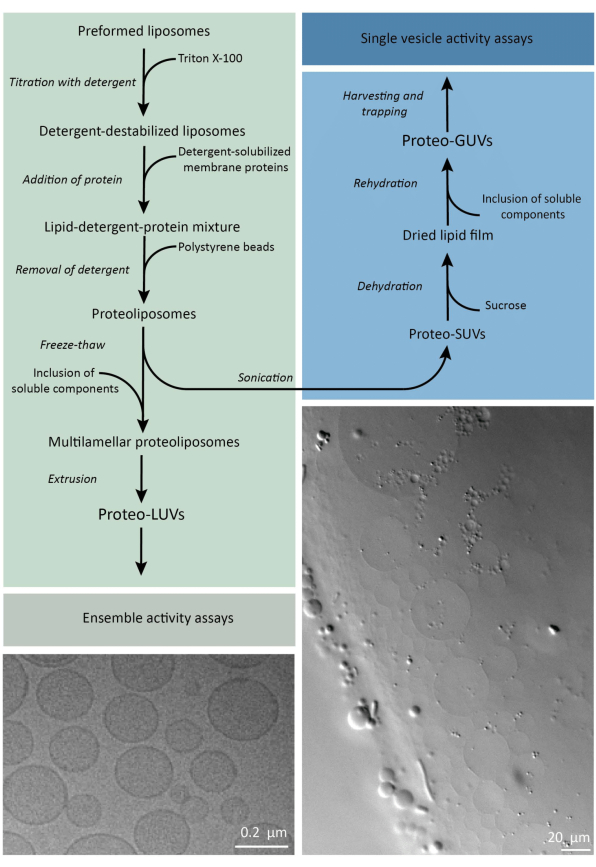
चित्रा 2: झिल्ली प्रोटीन के पुनर्गठन और उप-माइक्रोमीटर (एलयूवी) और माइक्रोमीटर आकार (जीयूवी) के लिपिड पुटिकाओं में एंजाइमों और पानी में घुलनशील घटकों को एनकैप्सुलेट करने के लिए प्रोटोकॉल का अवलोकन। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें।
पुनर्गठन और एनकैप्सुलेशन प्रोटोकॉल अच्छी तरह से काम करते हैं और प्रोटीन की कार्यक्षमता को बरकरार रखा जाता है, लेकिन प्रोटीओएलयूवी और प्रोटीओजीयूवी आकार में विषम हैं। माइक्रोफ्लुइडिक दृष्टिकोण31,32 माइक्रोमीटर आकार के पुटिकाओं के गठन की अनुमति देते हैं जो आकार में अधिक सजातीय होते हैं, लेकिन झिल्ली प्रोटीन का कार्यात्मक पुनर्गठन आमतौर पर संभव नहीं होता है क्योंकि बाइलेयर में अवशिष्ट विलायक प्रोटीन को निष्क्रिय करता है। प्रोटीओएलयूवी आकार में 100 से 400 एनएम तक होते हैं, और एंजाइमों की कम सांद्रता पर, एनकैप्सुलेशन अपूर्ण चयापचय मार्गों (स्टोकेस्टिक प्रभाव; चित्र 3 देखें) के साथ पुटिकाओं को जन्म दे सकता है। LUVs विशिष्ट चयापचय मॉड्यूल के निर्माण के लिए आदर्श हैं, जैसा कि एटीपी के उत्पादन और G3P जैसे बिल्डिंग ब्लॉक्स के लिए यहां दिखाया गया है। इस तरह के प्रोटीओएलयूवी को संभावित रूप से जीयूवी में समझाया जा सकता है और मेजबान पुटिकाओं के लिए ऑर्गेनेल जैसे डिब्बों के रूप में काम किया जा सकता है।
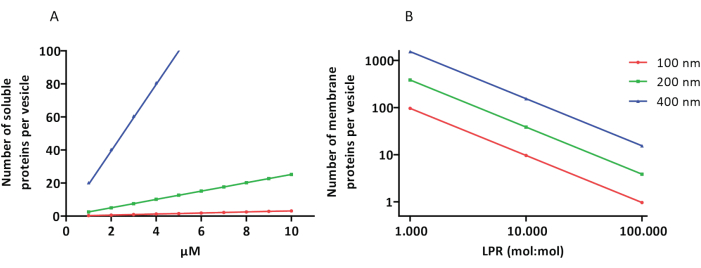
चित्रा 3: 100, 200, या 400 एनएम के व्यास के साथ प्रति पुटिका अणुओं की संख्या। (ए) जब समझाया प्रोटीन (एंजाइम, जांच) 1-10 माइक्रोन की सीमा में हैं। (बी) पुनर्गठन 1 से 1,000, 1 से 10,000, और 1 से 100,000 झिल्ली प्रोटीन प्रति लिपिड (मोल / मोल) पर किया जाता है। हम यह धारणा बनाते हैं कि अणुओं को संकेतित सांद्रता पर समझाया जाता है और इन प्रोटीन-से-लिपिड अनुपात में झिल्ली में शामिल किया जाता है। कुछ एंजाइमों के लिए, हमने देखा है कि वे झिल्ली से बंधते हैं, जो पुटिकाओं में उनकी स्पष्ट एकाग्रता को बढ़ा सकते हैं। संक्षिप्तीकरण: LPR = लिपिड-प्रोटीन-अनुपात कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें।
Protocol
Representative Results
Discussion
हम उप-माइक्रोमीटर आकार लिपिड पुटिकाओं (प्रोटीओएलयूवी) युक्त (झिल्ली) प्रोटीन के संश्लेषण के लिए एक प्रोटोकॉल प्रस्तुत करते हैं, और विशाल-यूनिलामेलर पुटिकाओं (प्रोटीओजीयूवी) में प्रोटीओएलयूवी के रूपा…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
लेखक pBAD-PercevalHR जीन की क्लोनिंग के लिए आदित्य अय्यर और प्रोटीन उत्पादन और शुद्धिकरण में सहायता के लिए Gea Schuurman-Wolters को धन्यवाद देते हैं। अनुसंधान को NWO गुरुत्वाकर्षण कार्यक्रम “बिल्डिंग ए सिंथेटिक सेल” (BaSyC) द्वारा वित्त पोषित किया गया था।
Materials
| Agarose | Sigma Aldrich | A9414-25g | |
| Amicon cut-off filter | Sigma Aldrich | Milipore centrifugal filter units Amicon Ultra | |
| BioBeads | BioRad | 152-3920 | |
| CHCl3 | Macron Fine Chemicals | MFCD00000826 | |
| D(+)-Glucose | Formedium | – | |
| D(+)-Sucrose | Formedium | – | |
| DDM | Glycon | D97002 -C | |
| Diethyl Ether | Biosolve | 52805 | |
| DMSO | Sigma-Aldrich | 276855-100ml | |
| DOPC | Avanti | 850375P-1g | |
| DOPE | Avanti | 850725P-1g | |
| DOPG | Avanti | 840475P-1g | |
| DTT | Formedium | DTT005 | |
| EtOH | J.T.Baker Avantor | MFCD00003568 | |
| Extruder | Avestin Inc | LF-1 | |
| Fluorimeter | Jasco | Spectrofluorometer FP-8300 | |
| Glycerol | BOOM | 51171608 | |
| Gravity flow column | Bio-Rad | 732-1010 | |
| Hamilton syringe 100 µL | Hamilton | 7656-01 | |
| Hamilton syringe 1000 µL | Hamilton | 81320 | |
| Handheld LCP dispenser | Art Robbins Instruments | 620-411-00 | |
| Handheld Sonicator | Hielscher Ultrasound Technology | UP50H | |
| HCl | BOOM | x76021889.1000 | |
| Imidazole | Roth | X998.4-250g | |
| K2HPO4 | Supelco | 1.05099.1000 | |
| KCl | BOOM | 76028270.1 | |
| KH2PO4 | Supelco | 1.04873.1000 | |
| Kimwipe | Kimtech Science | 7552 | |
| Large Falcon tube centrifuge | Eppendorf | Centrifuge 5810 R | |
| L-Arginine | Sigma-Aldrich | A5006-100G | |
| Light microscope | Leica | DM LS2 | |
| L-Ornithine | Roth | T204.1 | |
| LSM Laser Scanning Confocal Microscope | Zeiss | LSM 710 ConfoCor 3 | |
| MgCl2 | Sigma-Aldrich | M2670-1KG | |
| Microfluidic chip | Homemade | PDMS based | DOI: https://doi.org/10.1039/C8LC01275J |
| Na-ADP | Sigma-Aldrich | A2754-1G | |
| NaCl | Supelco | 1.06404.1000 | |
| Nanodrop Spectrometer | Isogen Life Science | ND-1000 spectrophotometer NanoDrop | |
| NaOH | Supelco | 1.06498.1000 | |
| Needles for GUVs | Henke-Ject | 14-14575 | 27 G x 3/4'' 0.4 x 20 mm |
| Needles for microfluidics | Henke-Ject | 14-15538 | 18 G x 1 1/2'' 1.2 x 40 mm |
| Ni2+ Sepharose | Cytiva | 17526802 | |
| Nigericin | Sigma-Aldrich | N7143-5MG | |
| Nutator | VWR | 83007-210 | |
| Osmolality meter | Gonotec Salmenkipp | Osmomat 3000 basic freezing point osmometer | |
| Plasmacleaner | Plasma Etch | PE-Avenger | |
| Polycarbonate filter | Cytiva Whatman | Nuclepor Track-Etch Membrane Product: 10417104 | 0.4 µm |
| Polycarbonate ultracentrifuge tube | Beckman Coulter | 355647 | |
| Pyranine | Acros Organics | H1529-1G | |
| Quartz cuvette (black) | Hellma Analytics | 108B-10-40 | |
| Sephadex G-75 resin | GE Healthcare | 17-0050-01 | |
| Sonicator | Sonics Sonics & Materials INC | Sonics vibra cell | |
| Syringe filter | Sarstedt | Filtropur S plus 0.2 | 0.2 µm |
| Syringe pump | Harvard Apparatus | A-42467 | |
| Tabletop centrifuge | Eppendorf | centrifuge 5418 | |
| Teflon spacer | Homemade | Teflon based | 45 x 26 x 1.5 or 45 x 26 x 3 or 20 x 20 x 3 mm |
| Tris | PanReac AppliChem | A1086.1000 | |
| Triton X-100 | Sigma Aldrich | T8787-100 ml | |
| Ultracentrifuge | Beckman Coulter | Optima Max-E | |
| UV lamp | Spectroline | ENB-280C/FE | |
| UV/VIS Spectrometer | Jasco | V730 spectrophotometer | |
| Valinomycin | Sigma-Aldrich | V0627-10MG | |
| Widefield fluorescence microscope | Zeiss | AxioObserver | |
| β-Casein | Sigma Aldrich | C5890-500g |
Riferimenti
- Hirschi, S., Ward, T. R., Meier, W. P., Müller, D. J., Fotiadis, D. Synthetic biology: bottom-up assembly of molecular systems. Chem Rev. 122 (21), 16294-16328 (2022).
- Ivanov, I., et al. Bottom-up synthesis of artificial cells: recent highlights and future challenges. Annu Rev Chem Biomol. Eng. 12 (1), 287-308 (2021).
- Clomburg, J. M., Crumbley, A. M., Gonzalez, R. Industrial biomanufacturing: The future of chemical production. Science. 355 (6320), (2017).
- Shi, T., Han, P., You, C., Zhang, Y. -. H. P. J. An in vitro synthetic biology platform for emerging industrial biomanufacturing: Bottom-up pathway design. Synth Syst Biotechnol. 3 (3), 186-195 (2018).
- Wang, A., et al. Liver-target and glucose-responsive polymersomes toward mimicking endogenous insulin secretion with improved hepatic glucose utilization. Adv Funct Mater. 30 (13), 1910168 (2020).
- Kanter, G., et al. Cell-free production of scFv fusion proteins: an efficient approach for personalized lymphoma vaccines. Blood. 109 (8), 3393-3399 (2007).
- Zeltins, A. Construction and characterization of virus-like particles: a review. Mol Biotechnol. 53 (1), 92-107 (2013).
- Jain, K. K. Synthetic biology and personalized medicine. Med Princ Pract. 22 (3), 209-219 (2013).
- Schwille, P., Frohn, B. P. Hidden protein functions and what they may teach us. Trends Cell Biol. 32 (2), 102-109 (2022).
- Sachsenmeier, P. Industry 5.0-The relevance and implications of bionics and synthetic biology. Ingegneria. 2 (2), 225-229 (2016).
- Schmidt, D., Jiang, Q. -. X., MacKinnon, R. Phospholipids and the origin of cationic gating charges in voltage sensors. Nature. 444 (7120), 775-779 (2006).
- Godoy-Hernandez, A., et al. Rapid and highly stable membrane reconstitution by LAiR enables the study of physiological integral membrane protein functions. ACS Cent Sci. 9 (3), 494-507 (2023).
- Sikkema, H. R., et al. Gating by ionic strength and safety check by cyclic-di-AMP in the ABC transporter OpuA. Sci Adv. 6 (47), 7697 (2020).
- Foucaud, C., Poolman, B. Lactose transport system of Streptococcus thermophilus. Functional reconstitution of the protein and characterization of the kinetic mechanism of transport. J Biol Chem. 267 (31), 22087-22094 (1992).
- Yoneda, J. S., Sebinelli, H. G., Itri, R., Ciancaglini, P. Overview on solubilization and lipid reconstitution of Na,K-ATPase: enzyme kinetic and biophysical characterization. Biophys Rev. 12 (1), 49-64 (2020).
- Simidjiev, I., et al. Self-assembly of large, ordered lamellae from non-bilayer lipids and integral membrane proteins in vitro. Proc Natl Acad Sci. 97 (4), 1473-1476 (2000).
- Harris, N. J., Booth, P. J. Folding and stability of membrane transport proteins in vitro. Biochim Biophys Acta BBA – Biomembr. 1818 (4), 1055-1066 (2012).
- Jackson, M. L., Litman, B. J. Rhodopsin-egg phosphatidylcholine reconstitution by an octyl glucoside dilution procedure. Biochim Biophys Acta BBA – Biomembr. 812 (2), 369-376 (1985).
- Geertsma, E. R., Nik Mahmood, N. A. B., Schuurman-Wolters, G. K., Poolman, B. Membrane reconstitution of ABC transporters and assays of translocator function. Nat Protoc. 3 (2), 256-266 (2008).
- Rigaud, J. -. L., Pitard, B., Levy, D. Reconstitution of membrane proteins into liposomes: application to energy-transducing membrane proteins. Biochim Biophys Acta BBA – Bioenerg. 1231 (3), 223-246 (1995).
- Szoka, F., Papahadjopoulos, D. Procedure for preparation of liposomes with large internal aqueous space and high capture by reverse-phase evaporation. Proc Natl Acad Sci. 75 (9), 4194-4198 (1978).
- . Synthetic Organelles for Energy Conservation and Delivery of Building Blocks for Lipid Biosynthesis Available from: https://www.researchsquare.com/article/rs-3385355/v1 (2023)
- Lee, K. Y., et al. Photosynthetic artificial organelles sustain and control ATP-dependent reactions in a protocellular system. Nat Biotechnol. 36 (6), 530-535 (2018).
- Méléard, P., Bagatolli, L. A., Pott, T. Giant unilamellar vesicle electroformation. Methods in Enzymology. , 161-176 (2009).
- Garten, M., Aimon, S., Bassereau, P., Toombes, G. E. S. Reconstitution of a transmembrane protein, the voltage-gated ion channel, KvAP, into giant unilamellar vesicles for microscopy and patch clamp studies. J. Vis. Exp. (95), e52281 (2015).
- Doeven, M. K., et al. lateral mobility and function of membrane proteins incorporated into giant unilamellar vesicles. Biophys J. 88 (2), 1134-1142 (2005).
- Pols, T., et al. A synthetic metabolic network for physicochemical homeostasis. Nat Commun. 10 (1), 4239 (2019).
- Bailoni, E., Poolman, B. ATP recycling fuels sustainable glycerol 3-phosphate formation in synthetic cells fed by dynamic dialysis. ACS Synth Biol. 11 (7), 2348-2360 (2022).
- Van Der Heide, T. On the osmotic signal and osmosensing mechanism of an ABC transport system for glycine betaine. EMBO J. 20 (24), 7022-7032 (2001).
- Van’T Klooster, J. S., et al. Membrane lipid requirements of the lysine transporter Lyp1 from Saccharomyces cerevisiae. J Mol Biol. 432 (14), 4023-4031 (2020).
- Lou, G., Anderluzzi, G., Woods, S., Roberts, C. W., Perrie, Y. A novel microfluidic-based approach to formulate size-tuneable large unilamellar cationic liposomes: Formulation, cellular uptake and biodistribution investigations. Eur J Pharm Biopharm. 143, 51-60 (2019).
- Weiss, M., et al. Sequential bottom-up assembly of mechanically stabilized synthetic cells by microfluidics. Nat Mater. 17 (1), 89-96 (2018).
- Pols, T., Singh, S., Deelman-Driessen, C., Gaastra, B. F., Poolman, B. Enzymology of the pathway for ATP production by arginine breakdown. FEBS J. 288 (1), 293-309 (2021).
- Yandrapalli, N., Robinson, T. Ultra-high capacity microfluidic trapping of giant vesicles for high-throughput membrane studies. Lab Chip. 19 (4), 626-633 (2019).
- Elias, M., et al. Microfluidic characterization of biomimetic membrane mechanics with an on-chip micropipette. Micro Nano Eng. 8, 100064 (2020).
- Robinson, T., Kuhn, P., Eyer, K., Dittrich, P. S. Microfluidic trapping of giant unilamellar vesicles to study transport through a membrane pore. Biomicrofluidics. 7 (4), 044105 (2013).
- Cooper, A., Girish, V., Subramaniam, A. B. Osmotic Pressure Enables High-Yield Assembly of Giant Vesicles in Solutions of Physiological Ionic Strengths. Langmuir. 39 (15), 5579-5590 (2023).
- Tantama, M., Martínez-François, J. R., Mongeon, R., Yellen, G. Imaging energy status in live cells with a fluorescent biosensor of the intracellular ATP-to-ADP ratio. Nat Commun. 4 (1), 2550 (2013).
- Setyawati, I., et al. In vitro reconstitution of dynamically interacting integral membrane subunits of energy-coupling factor transporters. eLife. 9, e64389 (2020).
- Oropeza-Guzman, E., Ríos-Ramírez, M., Ruiz-Suárez, J. C. Leveraging the coffee ring effect for a defect-free electroformation of giant unilamellar vesicles. Langmuir. 35 (50), 16528-16535 (2019).
- Estes, D. J., Mayer, M. Electroformation of giant liposomes from spin-coated films of lipids. Colloids Surf B Biointerfaces. 42 (2), 115-123 (2005).

