Анализируя Динамика белков с использованием водорода валютный масс-спектрометрии
Summary
Конформации белка и динамика являются ключевыми для понимания взаимосвязи между структурой и функции белка. Обмен водорода в сочетании с масс-спектрометрии высокого разрешения является универсальным методом для изучения конформационной динамики белков, а также характеризующие белок-лиганд и белок-белковых взаимодействий, в том числе контактных интерфейсов и аллостерических эффектов.
Abstract
Все клеточные процессы зависят от функциональности белков. Хотя функциональность данного белка является прямым следствием его уникальной аминокислотной последовательности, он реализуется только по сворачивания полипептидной цепи в единую определенную трехмерную договоренности или чаще в ансамбль взаимопревращени конформации. Исследуя связь между конформации белка и его функции Поэтому для полного понимания того, как белки в состоянии выполнить их большое разнообразие задач. Одной из возможностей для изучения конформационных изменений белка претерпевает в то время как прогрессирует через его функционального цикла представляет собой водород-1Н / 2 Н-обменного в сочетании с масс-спектрометрии высокого разрешения (HX-MS). HX-МС является универсальным и надежный метод, который добавляет новое измерение к структурной информации, полученной, например, кристаллографии. Он используется для изучения белок складывания и раскладывания, связывание небольшой мольecule лиганды, белок-белковые взаимодействия, конформационные изменения, связанные с ферментативного катализа и allostery. Кроме того, HX-MS часто используется, когда количество белка очень ограничено или кристаллизации белка не представляется возможным. Здесь мы предлагаем общий протокол для изучения динамики белков с HX-МС и описать в качестве примера, как показывают интерфейс взаимодействия двух белков в комплексе.
Introduction
Количество кристаллических структур белков и белковых комплексов резко увеличилось в последние годы. Они представляют бесценные снимки структурной организации этих белков и обеспечить основу для структуры и функции анализа. Тем не менее, динамика белков и конформационных изменений, которые необходимы для их функций, которые редко показал с помощью рентгеновской кристаллографии. Крио-Электронная микроскопия, с другой стороны, имеет возможность захвата белка и белковых комплексов в различных конформаций, но в целом не может решить конформационные изменения до среднего уровня структуры 1. Конформационная динамика белков в растворе на атомном деталей может быть решен только с помощью ЯМР, но этот метод по-прежнему ограничен белков относительно небольших размеров (обычно ≤ 30 кДа) и нуждается в высокой концентрации белков (≥ 100 мкм), что затрудняет эксперименты с олигомеризации или агрегации, склонные белков 2. Одним из методов,способна преодолеть между высоким разрешением рентгеновской кристаллографии и крио-Электронная микроскопия и который не ограничен размером белка или концентрации водорода является амид-1Н / 2 Н-обменного (HX) в сочетании с масс-спектрометрией (MS). В последние годы этот метод разработан для ценного аналитического инструмента для анализа динамики белков, сворачивания белка, стабильности белка и конформационных изменений 3-5. Молекулярные основы этого метода является лабильным характер системообразующих амидных атомов водорода в белках, которые будут обмениваться с атомами дейтерия, когда белок помещается в D 2 O раствора. Последующее увеличение массы белка с течением времени измеряется с высоким разрешением MS.
В коротких неструктурированных пептидов HX зависит только от температуры, концентрации катализатора (ОН -, Н 3 О + т.е. рН, см. рисунок 3) и боковых цепей аминокислот из соседних остатков из-за индуктивной, кошкиalytic и пространственные эффекты. Эти эффекты на внутренней химической обменный курс К ч были элегантно количественно Бай и др.. 6 и программа доступна (любезность З. Чжан), который вычисляет К ч для каждой аминокислоты в полипептид в зависимости от рН и температуры. При нейтральном рН и температуре окружающей среды К ч составляет порядка 10 1 -10 3 с -1. В свернутых белков HX может быть 2-9 порядков медленнее в основном за счет водородных связей в вторичной структуры и в меньшей степени за счет ограниченного доступа гидратированных ОН – ионов к внутренней части плотно уложенного белка. HX в нативных белков поэтому подразумевает частичную или глобальной разворачивается, химический обмен и повторной укладки в нативном состоянии в соответствии с уравнением (1) и наблюдаемые курсы обмена K набл зависеть от темпов открытия К ор, скорость закрытия К кл и внутренней химического обмена раТе К ч в соответствии с уравнением (2).
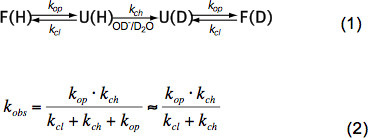
Под носителями государственных условиях к оп намного меньше, чем К ч и ими можно пренебречь в знаменателе. Есть два крайних режимов обмена называемые EX1 и EX2. Если к кл намного меньше, чем К ч (EX1) наблюдаемая скорость практически равна скорости открытия и HX позволяет сразу же наблюдение разворачивание структурного элемента. Такой обмен режим, где все амидные протоны обмен сразу при открытии структурного элемента, легко наблюдаемая в MS бимодальным распределением изотопов пиков 7. Если к кл намного больше, чем К ч (EX2) наблюдаемая скорость пропорциональна К ч причем коэффициент пропорциональности равен складных-разворачивания равновесий постоянная К U = K оп </sub> К / кл. В этих условиях многие открытия и закрытия мероприятия необходимы, прежде всего обмен амидные протонов для дейтронов, что приводит к постепенному увеличению средней массы в то время как изотопный распределение остается примерно такой же. Режим EX2 позволяет определить свободной энергии разворачивается ΔG и и поэтому стабильность структурного элемента. Под родной государственной состоянии режим EX2 является наиболее распространенным. Повышение рН и добавлением хаотропных агентов можно сдвинуть обменный механизм EX1. Таким образом, HX-MS может быть использован для изучения термодинамических а также кинетические параметры складывания белка и конформационных изменений.
Как упоминалось выше HX внутренне рН и температура зависят и обмен полураспада полностью открытой растворителя протона магистральной амидной группы находится между 5-400 мс при физиологическом значении рН (рН 7,6) и 30 ° С, но 10 мин до> 15 час при среднем значении> 2 ч при рН 2,9 и 0 °С (для протона первого магистральной амидной связи полипептида, который обменивается с периодом полураспада ок. 1-2 мин исключением). В таких медленно обменивающихся условиях можно переварить примера с использованием протеазы (например пепсин), которые активны в этих условиях, с нашими потерять всю информацию, содержащуюся в объединенных дейтронов. С момента введения язвенной пищеварения при медленных условиях обмена, не только общие HX кинетика полноразмерные белки могут быть проанализированы, но HX могут быть локализованы в конкретных регионах 8,9. Пространственное разрешение в настоящее время ограничивается размером пептической фрагментов, полученных, которая является в целом 10-30 остатков. Тем не менее, перекрывающиеся фрагменты, созданные за счет неспецифического характера расщепления пепсином может привести к увеличению пространственного разрешения. Кроме того, несколько других протеаз, оказались активными при условиях закалки, однако, значительно менее эффективен, чем пепсина 10. Кроме того увеличилисьсебе пространственного разрешения может быть достигнуто путем фрагментации пептидов в газовой фазе методами, которые сохранились дейтерирование шаблон например захвата электронов диссоциации (ECD), перенос электрона диссоциации (ETD) и инфракрасного многофотонная диссоциация (ИКМФД) 11-13. Эти методы предотвращения потери пространственного разрешения в связи с внутримолекулярной миграции протонов ("скремблирования"), которое наблюдается на столкновительное диссоциации (CID) наиболее часто используется методика фрагментации. Однако эти методы требуют оптимизации для каждого отдельного пептида и, таким образом, все еще довольно сложной задачей.
HX-MS была использована для анализа белок-лиганд и белок-белковых взаимодействий в том числе вирусного капсида сборки 14-17. Белок разворачивается и повторной укладки, а также температуры, вызванные конформационные изменения были исследованы 7,18,19. Фосфорилирования и один аминокислотный мутация связанных конформационные изменения 16,20 и nucleotязь-индуцированные изменения были проанализированы 21,22. Таким образом, этот метод кажется идеально подходит для анализа сборки и динамики молекулярных машин. Один привлекательным кандидатом, механизм которого имеет большое общий интерес, является компаньонка комплекс Hsp90.
Protocol
Representative Results
Discussion
Связывание партнера взаимодействия с белком неизбежно приводит к изменениям в доступности растворителя на сайте связывания. Кроме того, многие белки подвергаются динамические конформационные изменения при связывании, которые влияют на другие регионы, чем действующей связывающей ин…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Мы благодарим М. Boysen замечания по рукописи. Этот проект был профинансирован Deutsche Forschungsgemeinschaft (SFB638 и MA 1278/4-1 в MPM и кластера передового опыта: CellNetworks EXC 81/1). MPM является следователь кластера передового опыта: CellNetworks.
Materials
| Reagent/Equipment | Company | Catalogue number | Comments (optional) |
| maXis QTOF | Bruker | ||
| nanoAcquity UPLC | Waters Corp. | ||
| Shimadzu 10AD-VP | Shimadzu | ||
| 6-port Valve EPC6W with microelectric actuator | Valco | #EPC6W | |
| Injection valve (manual) | Rheodyne | #7725 | |
| Poros AL20 media | Applied Biosystems | #1-6029-06 | |
| Poros R2 | Applied Biosystems | #1-1118-02 | |
| Pepsin | Sigma | #P6887 | use fresh pepsin |
| Microbore (1 mm) | IDEX | #C-128 | |
| Microbore (2 mm) | IDEX | #C-130B | |
| Acquity UPLC BEH C8 Column | Waters Corp. | #186002876 | |
| Thermomixer | Eppendorf | #5355000.011 | |
| Tubing (various diameters) | IDEX | ||
| Fittings | IDEX | #PK-110 with PK-100 |
References
- Saibil, H. R. Conformational changes studied by cryo-electron microscopy. Nat. Struct. Biol. 7 (9), 711-714 (2000).
- Mittermaier, A., Kay, L. E. New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Hoofnagle, A. N., Resing, K. A., Ahn, N. G. Protein analysis by hydrogen exchange mass spectrometry. Annu. Rev. Biophys. Biomol. Struct. 32, 1-25 (2003).
- Wales, T. E., Engen, J. R. Hydrogen exchange mass spectrometry for the analysis of protein dynamics. Mass. Spectrom. Rev. 25 (1), 158-170 (2006).
- Konermann, L., Pan, J., Liu, Y. -. H. Hydrogen exchange mass spectrometry for studying protein structure and dynamics. Chem Soc. Rev. 40 (3), 1224-1210 (2011).
- Bai, Y., Milne, J. S., Mayne, L., Englander, S. W. Primary structure effects on peptide group hydrogen exchange. Proteins. 17 (1), 75-86 (1993).
- Rist, W., Jørgensen, T. J. D., Roepstorff, P., Bukau, B., Mayer, M. P. Mapping temperature-induced conformational changes in the Escherichia coli heat shock transcription factor sigma 32 by amide hydrogen exchange. The Journal of biological chemistry. 278 (51), 51415-51421 (1074).
- Englander, J. J., Rogero, J. R., Englander, S. W. Protein hydrogen exchange studied by the fragment separation method. Anal Biochem. 147 (1), 234-244 (1985).
- Zhang, Z., Smith, D. L. Determination of amide hydrogen exchange by mass spectrometry: a new tool for protein structure elucidation. Protein Sci. 2 (4), 522-531 (1993).
- Cravello, L., Lascoux, D., Forest, E. Use of different proteases working in acidic conditions to improve sequence coverage and resolution in hydrogen/deuterium exchange of large proteins. Rapid Commun. Mass Spectrom. : RCM. 17 (21), 2387-2393 (2003).
- Rand, K. D., Zehl, M., Jensen, O. N., Jørgensen, T. J. D. Protein hydrogen exchange measured at single-residue resolution by electron transfer dissociation mass spectrometry. Anal chem. 81 (14), 5577-5584 (2009).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Electron capture dissociation of electrosprayed protein ions for spatially resolved hydrogen exchange measurements. J. Am. Chem. Soc. 130 (35), 11574-11575 (2008).
- Yamada, N., Suzuki, E. -. I., Hirayama, K. Identification of the interface of a large protein-protein complex using H/D exchange and Fourier transform ion cyclotron resonance mass spectrometry. Rapid Commun. Mass Spectrom. 16 (4), 293-299 (2002).
- Lee, T., Hoofnagle, A. N., et al. Docking motif interactions in MAP kinases revealed by hydrogen exchange mass spectrometry. Mol. Cell. 14 (1), 43-55 (2004).
- Hasan, A., Smith, D. L., Smith, J. B. Alpha-crystallin regions affected by adenosine 5′-triphosphate identified by hydrogen-deuterium exchange. Biochem. 41 (52), 15876-15882 (2002).
- Lanman, J., Lam, T. T., Emmett, M. R., Marshall, A. G., Sakalian, M., Prevelige, P. E. Key interactions in HIV-1 maturation identified by hydrogen-deuterium exchange. Nat. Struct. Mol. Biol. 11 (7), 676-677 (2004).
- Wang, L., Lane, L. C., Smith, D. L. Detecting structural changes in viral capsids by hydrogen exchange and mass spectrometry. Protein Sci. 10 (6), 1234-1243 (2001).
- Pan, H., Raza, A. S., Smith, D. L. Equilibrium and kinetic folding of rabbit muscle triosephosphate isomerase by hydrogen exchange mass spectrometry. J. Mol. Biol. 336 (5), 1251-1263 (2004).
- Mazon, H., Marcillat, O., Forest, E., Smith, D. L., Vial, C. Conformational dynamics of the GdmHCl-induced molten globule state of creatine kinase monitored by hydrogen exchange and mass spectrometry. Biochem. 43 (17), 5045-5054 (2004).
- Lanman, J., Lam, T. T., et al. Identification of novel interactions in HIV-1 capsid protein assembly by high-resolution mass spectrometry. J. Mol. Biol. 325 (4), 759-772 (2003).
- Rist, W., Graf, C., Bukau, B., Mayer, M. P. Amide hydrogen exchange reveals conformational changes in hsp70 chaperones important for allosteric regulation. J. Biol. chem. 281 (24), 16493-16501 (2006).
- Graf, C., Stankiewicz, M., Kramer, G., Mayer, M. P. Spatially and kinetically resolved changes in the conformational dynamics of the Hsp90 chaperone machine. EMBO J. 28 (5), 602-613 (2009).
- Lee, C. -. T., Graf, C., Mayer, F. J., Richter, S. M., Mayer, M. P. Dynamics of the regulation of Hsp90 by the co-chaperone Sti1. EMBO J. 31 (6), 1518-1528 (2012).
- Lou, X., Kirchner, M., et al. Deuteration distribution estimation with improved sequence coverage for HX/MS experiments. Bioinformatics. 26 (12), 1535-1541 (2010).
- Kreshuk, A., Stankiewicz, M., Lou, X., Kirchner, M., Hamprecht, F. A., Mayer, M. P. Automated detection and analysis of bimodal isotope peak distributions in H/D exchange mass spectrometry using HeXicon. Intl. J. mass spectrom. 302 (1-3), 125-131 (2011).
- Walters, B. T., Ricciuti, A., Mayne, L., Englander, S. W. Minimizing back exchange in the hydrogen exchange-mass spectrometry experiment. J. Am. Soc. Mass Spectrom. 23 (12), 2132-2139 (2012).
- Weis, D. D., Wales, T. E., Engen, J. R., Hotchko, M., Ten Eyck, L. F. Identification and characterization of EX1 kinetics in H/D exchange mass spectrometry by peak width analysis. J. Am. Soc. Mass Spectrom. 17 (11), 1498-1509 (2006).
- Fang, J., Rand, K. D., Beuning, P. J., Engen, J. R. False EX1 signatures caused by sample carryover during HX MS analyses. Intl. J. Mass Spectrom. 302 (1-3), 19-25 (2011).
- Deng, Y., Smith, D. L. Identification of unfolding domains in large proteins by their unfolding rates. Biochem. 37 (18), 6256-6262 (1998).
- Wales, T. E., Engen, J. R. Partial unfolding of diverse SH3 domains on a wide timescale. J. Mol. Biol. 357 (5), 1592-1604 (2006).
- Pan, Y., Piyadasa, H., O’Neil, J. D., Konermann, L. Conformational dynamics of a membrane transport protein probed by h/d exchange and covalent labeling: the glycerol facilitator. J. Mol. Biol. 416 (3), 400-413 (2012).
- Hebling, C. M., Morgan, C. R., Stafford, D. W., Jorgenson, J. W., Rand, K. D., Engen, J. R. Conformational analysis of membrane proteins in phospholipid bilayer nanodiscs by hydrogen exchange mass spectrometry. Anal chem. 82 (13), 5415-5419 (2010).
- Abzalimov, R. R., Kaplan, D. A., Easterling, M. L., Kaltashov, I. A. Protein conformations can be probed in top-down HDX MS experiments utilizing electron transfer dissociation of protein ions without hydrogen scrambling. J. Am. Soc. Mass Spectrom. 20 (8), 1514-1517 (2009).

