Karakterisering van complexe systemen met behulp van de Design of Experiments Aanpak: Transient Eiwitexpressie in tabak als een case study
Summary
We beschrijven een experimenteel ontwerp aanpak die kan worden bepaald en modelleren invloed van transgene regulerende elementen plantengroei en ontwikkeling parameters en incubatieomstandigheden de tijdelijke expressie van monoklonale antilichamen en reporter eiwitten in planten.
Abstract
Planten bieden meerdere voordelen voor de productie van biofarmaceutische producten met lage kosten, schaalbaarheid en veiligheid. Tijdelijke expressie biedt het bijkomende voordeel van de korte ontwikkeling en productie tijden, maar expressie niveaus kunnen aanzienlijk verschillen tussen batches dus aanleiding geven tot regelgeving betreft in het kader van de goede praktijken bij het vervaardigen. We gebruikten een ontwerp van experimenten (DOE) benadering van het effect van de belangrijkste factoren als regulerende elementen in het expressieconstruct, plantengroei en ontwikkeling, alsook de incubatieomstandigheden tijdens expressie, de variabiliteit van expressie tussen partijen kan worden toegepast. We testten planten die een model anti-HIV-monoklonaal antilichaam (2G12) en een fluorescente merker proteïne (DsRed). We bespreken de reden voor het selecteren van bepaalde eigenschappen van het model en identificeren van de mogelijke beperkingen. De algemene aanpak kan gemakkelijk worden overgedragen aan andere problemen, omdat de beginselen van het model eenre breed toepasbaar: op kennis gebaseerde parameter selectie, complexiteit reductie door het splitsen van de initiële probleem in kleinere modules,-software stapsgewijze installatie van optimale experiment combinaties en stapsgewijze ontwerp augmentatie. Daarom is de methode is niet alleen nuttig voor het karakteriseren van eiwitexpressie in planten, maar ook voor het onderzoeken van andere complexe systemen ontbreekt een mechanistische beschrijving. De voorspellende vergelijkingen die de interconnectiviteit tussen parameters kunnen worden gebruikt om mechanistische modellen voor andere complexe systemen stellen.
Introduction
De productie van biofarmaceutische eiwitten in planten is gunstig omdat planten goedkoop te kweken, kan het platform worden opgeschaald door gewoon groeit planten en menselijke pathogenen niet kunnen repliceren 1,2. Tijdelijke expressie strategieën gebaseerd bijvoorbeeld op infiltratie van bladeren met Agrobacterium tumefaciens biedt extra voordelen omdat de tijd tussen het moment van levering van DNA en de levering van een gezuiverd product wordt verminderd van jaar tot minder dan 2 maanden 3. Tijdelijke expressie wordt ook gebruikt voor functionele analyse, bijvoorbeeld om genen te testen op hun vermogen om verlies-van-functie mutanten aanvulling of eiwitinteracties 4-6 onderzoeken. Echter, transiënte expressieniveaus vaak meer batch-to-batch variatie dan expressieniveaus in transgene planten 7-9 tonen. Dit vermindert de waarschijnlijkheid dat biofarmaceutische productieprocessen op basis van transiënte expressie wizal worden goedgekeurd in het kader van goede fabricagemethoden (GMP), omdat de reproduceerbaarheid is een kritische attribuut kwaliteit en is onderhevig aan risico-evaluatie 10. Dergelijke variatie kan ook maskeren alle interacties die onderzoekers willen onderzoeken. Daarom hebben we besloten om de belangrijkste factoren die transiënte expressie niveaus in planten beïnvloeden en een hoge-kwaliteit kwantitatief voorspellend model te bouwen identificeren.
De een-factor-per-tijd (OFAT) methode wordt vaak gebruikt om het effect (werking) van bepaalde parameters (factoren) van de resultaten (respons) van een experiment 11 karakteriseren. Maar dit is niet optimaal omdat de individuele tests (loopt) tijdens een onderzoek (experiment) zal worden aangepast als parels aan een koord door het potentieel gebied overspannen door de factoren die worden getest (ontwerp ruimte). De dekking van het ontwerp ruimte en daarmee de mate van informatie uit het experiment islaag, zoals getoond in figuur 1A 12. Bovendien kunnen onderlinge afhankelijkheden tussen verschillende factoren (factor interacties) blijven verborgen resulteert in slechte modellen en / of voorspelling van valse optima, zoals weergegeven in figuur 1B 13.
De hierboven beschreven nadelen kunnen worden vermeden door een ontwerp van experimenten (DOE) benadering waarbij de runs van een experiment meer gelijkmatig verspreid over de ontwerpruimte, betekent dat meer dan een factor wordt gevarieerd tussen twee punten 14. Er zijn gespecialiseerde ontwerpen voor mengsels, screening factoren (factoriële designs) en de kwantificering van factor effecten op reacties (respons oppervlak methoden, RSM en) 15. Bovendien kan RSMs worden gerealiseerd als centraal-composite ontwerpen, maar kan ook effectief worden bereikt met behulp van gespecialiseerde software die verschillende criteria van toepassing kan zijn voor de selectie van de pistes. Bijvoorbeeld, de zogenaamde D-optimality criterium zal runs selecteren, zodat om de fout te minimaliseren in de coëfficiënten van het resulterende model, terwijl de IV-optimalisatiecriterium selecteert runs die de laagste voorspelling variantie door het ontwerp ruimte 15,16 bereiken. De RSM we hier beschrijven kan de precieze kwantificering van voorbijgaande eiwit expressie in planten, maar het kan gemakkelijk worden overgedragen naar een systeem waarbij meerdere (~ 5-8) numerieke factoren (bijvoorbeeld temperatuur, tijd, concentratie) en een paar (~ 2 – 4) categorische factoren (bijv. promotor, kleur) waarin een mechanistische beschrijving is beschikbaar of te complex model.
De DoE benadering ontstaan in de landbouwwetenschappen maar heeft verspreid naar andere gebieden, omdat het overdraagbaar aan een situatie waarin het nuttig om het aantal runs moeten betrouwbare gegevens te verkrijgen verminderen en beschrijvende modellen voor complexe processen. Dit heeft geleid tot het opnemen van DoE in de "Leidraad voorIndustrie, Q8 (R2) Pharmaceutical Development ", gepubliceerd door de Internationale Conferentie voor Harmonisatie van de technische voorschriften voor de registratie van farmaceutische producten voor menselijk gebruik (ICH) 17. DoE wordt nu op grote schaal gebruikt in wetenschappelijk onderzoek en de industrie 18. Moet echter zorg tijdens worden genomen de planning en uitvoering van het experiment, omdat het selecteren van een onjuiste polynomiale graad voor de meervoudige lineaire-regressiemodel (basismodel) kan een behoefte aan extra punten voor alle factor effecten correct model te introduceren. Bovendien, beschadigde of ontbrekende gegevens te genereren onjuiste modellen en gebrekkig voorspellingen, en kan zelfs voorkomen dat modelbouw poging zoals beschreven in het protocol en discussie secties 18. In de sectie protocol, zullen we in eerste instantie de belangrijkste planning stappen voor een RSM-based experiment uiteengezet en vervolgens verklaren het ontwerp op basis van het DoE software DesignExpert v8.1. Maar gelijkaardige ontwerpen gebouwd kan worden met andere software including JMP, Modde, en STATISTICA. De experimentele procedures worden gevolgd door instructies voor data-analyse en evaluatie.
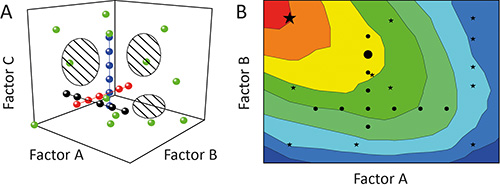
Figuur 1. Vergelijking van OFAT en DoE. A. Sequentiële variatie van een factor per keer (OFAT) in een experiment (zwart, rood en blauwe cirkels) realiseert een lage dekkingsgraad van het ontwerp ruimte (gearceerde gebieden). In tegenstelling, de variatie van meerdere factoren tegelijk met het experimenteel ontwerp (DOE) strategie (groene cirkels) verhoogt de dekking en daarmee de nauwkeurigheid van de resulterende modellen. B. De bevooroordeelde ontwerp ruimte dekking betekent dat OFAT experimenten (zwarte cirkels) ook kan falen om een optimale operationele gebieden (rood) identificeren en suboptimale oplossingen (grote zwarte cirkel) te voorspellen, terwijl DoE strategisches (zwarte sterren) hebben meer kans om de voorkeur omstandigheden (grote zwarte ster) te identificeren.
Protocol
Representative Results
Discussion
Elk experiment vereist een zorgvuldige planning omdat de middelen zijn vaak schaars en duur. Dit geldt met name voor DoE strategieën omdat fouten tijdens de planningsfase (bijvoorbeeld het selecteren van een basismodel dat geen betrekking heeft op alle belangrijke factor interacties) aanzienlijk de voorspellende kracht van de resulterende modellen kunnen verminderen en dus devalueren het hele experiment. Toch kunnen deze fouten gemakkelijk vermeden worden door het volgen van elementaire procedures.
<p clas…Disclosures
The authors have nothing to disclose.
Acknowledgements
De auteurs zijn dank aan Dr Thomas Rademacher voor het verstrekken van de PPAM plantenexpressievector en Ibrahim Al Amedi voor het kweken van de tabaksplanten gebruikt in deze studie. We willen graag Dr Richard M. Twyman bedanken voor zijn hulp bij het bewerken van het manuscript. Dit werk werd deels gefinancierd door de European Research Council Advanced Grant "Future-Pharma", voorstel nummer 269110 en het Fraunhofer Zukunftsstiftung (Fraunhofer Future Foundation).
Materials
| Design-Expert(R) 8 | Stat-Ease, Inc. | n.a. | DoE software |
| Tryptone | Carl Roth GmbH | 8952.2 | Media component |
| Yeast extract | Carl Roth GmbH | 2363.2 | Media component |
| Sodium chloride | Carl Roth GmbH | P029.2 | Media component |
| Ampicillin | Carl Roth GmbH | K029.2 | Antibiotic |
| Agar-Agar | Carl Roth GmbH | 5210.2 | Media component |
| Escherichia coli K12 DH5a | Life technologies | 18263-012 | Microorganism |
| pPAM | GenBank | AY027531 | Cloning/expression vector; |
| NucleoSpin Plasmid | MACHEREY-NAGEL GmbH | 740588.250 | Plasmid DNA isolation kit |
| NucleoSpin Gel and PCR Clean-up | MACHEREY-NAGEL GmbH | 740609.250 | Plasmid DNA purification kit |
| NanoDrop 2000 | Thermo Scientific | n.a. | Spectrophotometer |
| NcoI | New England Biolabs Inc. | R3193L | Restrictionendonuclease |
| EcoRI | New England Biolabs Inc. | R3101L | Restrictionendonuclease |
| AscI | New England Biolabs Inc. | R0558L | Restrictionendonuclease |
| NEB 4 | New England Biolabs Inc. | B7004S | Restrictionendonuclease buffer |
| TRIS | Carl Roth GmbH | 4855.3 | Media component |
| Disodium tetraborate | Carl Roth GmbH | 4403.3 | Media component |
| EDTA | Carl Roth GmbH | 8040.2 | Media component |
| Agarose | Carl Roth GmbH | 6352.4 | Media component |
| Bromophenol blue | Carl Roth GmbH | A512.1 | Color indicator |
| Xylene cyanol | Carl Roth GmbH | A513.1 | Color indicator |
| Glycerol | Carl Roth GmbH | 7530.2 | Media component |
| Mini-Sub Cell GT Cell | BioRad | 170-4406 | Gel electrophoresis chamber |
| Agrobacterium tumefaciens strain GV3101:pMP90RK | DSMZ | 12365 | Microorganism |
| Electroporator 2510 | Eppendorf | 4307000.658 | Electroporator |
| Beef extract | Carl Roth GmbH | X975.2 | Media component |
| Peptone | Carl Roth GmbH | 2365.2 | Media component |
| Sucrose | Carl Roth GmbH | 4621.2 | Media component |
| Magnesium sulfate | Carl Roth GmbH | 0261.3 | Media component |
| Carbenicillin | Carl Roth GmbH | 6344.2 | Antibiotic |
| Kanamycin | Carl Roth GmbH | T832.3 | Antibiotic |
| Rifampicin | Carl Roth GmbH | 4163.2 | Antibiotic |
| FWD primer | Eurofins MWG Operon | n.a. | CCT CAG GAA GAG CAA TAC |
| REV primer | Eurofins MWG Operon | n.a. | CCA AAG CGA GTA CAC AAC |
| 2720 Thermal cycler | Applied Biosystems | 4359659 | Thermocycler |
| RNAfold webserver | University of Vienna | n.a. | Software |
| Ferty 2 Mega | Kammlott | 5.220072 | Fertilizer |
| Grodan Rockwool Cubes 10x10cm | Grodan | n.a. | Rockwool block |
| Greenhouse | n.a. | n.a. | For plant cultivation |
| Phytotron | Ilka Zell | n.a. | For plant cultivation |
| Omnifix-F Solo | B. Braun | 6064204 | Syringe |
| Murashige and Skoog salts | Duchefa | M 0222.0010 | Media component |
| Glucose | Carl Roth GmbH | 6780.2 | Media component |
| Acetosyringone | Sigma-Aldrich | D134406-5G | Phytohormon analogon |
| BioPhotometer plus | Eppendorf | 6132 000.008 | Photometer |
| Osram cool white 36 W | Osram | 4930440 | Light source |
| Disodium phosphate | Carl Roth GmbH | 4984.3 | Media component |
| Centrifuge 5415D | Eppendorf | 5424 000.410 | Centrifuge |
| Forma -86C ULT freezer | ThermoFisher | 88400 | Freezer |
| Synergy HT | BioTek | SIAFRT | Fluorescence plate reader |
| Biacore T200 | GE Healthcare | n.a. | SPR device |
| Protein A | Life technologies | 10-1006 | Antibody binding protein |
| HEPES | Carl Roth GmbH | 9105.3 | Media component |
| Tween-20 | Carl Roth GmbH | 9127.3 | Media component |
| 2G12 antibody | Polymun | AB002 | Reference antibody |
References
- Fischer, R., Emans, N. Molecular farming of pharmaceutical proteins. Transgenic research. 9, 277-299 (2000).
- Commandeur, U., Twyman, R. M., Fischer, R. The biosafety of molecular farming in plants. AgBiotechNet. 5, 9 (2003).
- Shoji, Y., et al. A plant-based system for rapid production of influenza vaccine antigens. Influenza Other Resp. 6, 204-210 (2012).
- Goodin, M. M., Zaitlin, D., Naidu, R. A., Lommel, S. A. Nicotiana benthamiana: Its history and future as a model for plant-pathogen interactions. Mol Plant Microbe In. 21, 1015-1026 (2008).
- Berg, R. H., Beachy, R. N. Fluorescent protein applications in plants. Method Cell Biol. 85, 153 (2008).
- Chung, S. M., Vaidya, M., Tzfira, T. Agrobacterium is not alone: gene transfer to plants by viruses and other bacteria. Trends in plant science. 11, 1-4 (2006).
- Sheludko, Y. V., Sindarovska, Y. R., Gerasymenko, I. M., Bannikova, M. A., Kuchuk, N. V. Comparison of several Nicotiana species as hosts for high-scale Agrobacterium-mediated transient expression. Biotechnology and Bioengineering. 96, 608-614 (2007).
- Wydro, M., Kozubek, E., Lehmann, P. Optimization of transient Agrobacterium-mediated gene expression system in leaves of Nicotiana benthamiana. Acta Biochimica Polonica. 53, 289-298 (2006).
- Buyel, J. F., Fischer, R. Processing heterogeneous biomass: Overcoming the hurdles in model building. Bioengineered. 4, (2013).
- Fischer, R., Schillberg, S., Hellwig, S., Twyman, R. M., Drossard, J. GMP issues for recombinant plant-derived pharmaceutical proteins. Biotechnol Adv. 30, 434-439 (2012).
- Daniel, C. One-at-a-time plans. Journal of the American Statistical Association. 68, 353-360 (1973).
- Czitrom, V. One-Factor-at-a-Time versus Designed Experiments The American Statistician. 53, 6 (1999).
- Anderson, M. J., Kraber, S. L. Keys to successful designed experiments. ASQ – The global voice of quality. 6, 6 (1999).
- Montgomery, D. C. . Design and Analysis of Experiments. , (2007).
- Myers, R. H., Montgomery, D. C., Anderson-Cook, C. M. . Response Surface Methodology: Process and Product Optimization Using Designed Experiments. , (2009).
- Piepel, G. F. Programs for generating extreme vertices and centroids of linearly constrained experimental regions. J Qual Technol. 20, 15 (1988).
- . . FDA. , (2009).
- Shivhare, M., McCreath, G. Practical Considerations for DoE Implementation in Quality By Design. BioProcess International. 8, 9 (2010).
- Buyel, J. F., Fischer, R. Predictive models for transient protein expression in tobacco (Nicotiana tabacum L.) can optimize process time, yield, and downstream costs. Biotechnology and bioengineering. 109, 2575-2588 (2012).
- Buyel, J. F., Kaever, T., Buyel, J. J., Fischer, R. Predictive models for the accumulation of a fluorescent marker protein in tobacco leaves according to the promoter/5’UTR combination. Biotechnology and bioengineering. 110, 471-482 (2013).
- Anderson, M. J., Whitcomb, P. J. . DOE Simplified: Practical Tools for Effective Experimentation. , (2000).
- Anderson, M. J., Whitcomb, P. J. . Response Surface Methods Simplified. , (2005).
- De Gryze, S., Langhans, I., Vandebroek, M. Using the correct intervals for prediction: A tutorial on tolerance intervals for ordinary least-squares regression. Chemometr Intell Lab. 87, 147-154 (2007).
- . . Plasmid DNA purification User manual. , (2012).
- . . PCR clean-up Gel extraction User manual. , (2012).
- . . Quick Ligation Protocol. 4, (2009).
- Inoue, H., Nojima, H., Okayama, H. High-Efficiency Transformation of Escherichia-Coli with Plasmids. Gene. 96, 23-28 (1990).
- Main, G. D., Reynolds, S., Gartland, J. S. Electroporation protocols for Agrobacterium. Methods in Molecular Biology. 44, 405-412 (1995).
- Gruber, A. R., Lorenz, R., Bernhart, S. H., Neubock, R., Hofacker, I. L. The Vienna RNA websuite. Nucleic acids research. 36, 70-74 (2008).
- Howell, S., Kenmore, M., Kirkland, M., Badley, R. A. High-density immobilization of an antibody fragment to a carboxymethylated dextran-linked biosensor surface. J Mol Recognit. 11, 200-203 (1998).
- Newcombe, A. R., et al. Evaluation of a biosensor assay to quantify polyclonal IgG in ovine serum used for the production of biotherapeutic antibody fragments. Process Biochem. 41, 842-847 (2006).
- Peixoto, J. L. Hierarchical Variable Selection in Polynomial Regression-Models. Am Stat. 41, 311-313 (1987).
- Peixoto, J. L. A Property of Well-Formulated Polynomial Regression-Models. Am Stat. 44, 26-30 (1990).
- Sanders, P. R., Winter, J. A., Barnason, A. R., Rogers, S. G., Fraley, R. T. Comparison of cauliflower mosaic virus 35S and nopaline synthase promoters in transgenic plants. Nucleic acids research. 15, 1543-1558 (1987).
- Ma, J. K. C., et al. Generation and Assembly of Secretory Antibodies in Plants. Science. 268, 716-719 (1995).
- Wycoff, K. L. Secretory IgA antibodies from plants. Curr Pharm Design. 11, 2429-2437 (2005).
- Pace, C. N., Vajdos, F., Fee, L., Grimsley, G., Gray, T. How to measure and predict the molar absorption coefficient of a protein. Protein Sci. 4, 2411-2423 (1995).

