Charakterisierung komplexer Systeme Mit dem Design of Experiments Ansatz: Transient Protein Expression in Tabak als Case Study
Summary
Wir beschreiben eine Anordnung von Experimenten Ansatz, der verwendet werden kann, um festzustellen und das Modell des Einflusses der Transregulationselemente, Pflanzenwachstum und Entwicklung Parameter und Inkubationsbedingungen für die vorübergehende Expression von monoklonalen Antikörpern und Reporter-Proteine in Pflanzen auf.
Abstract
Pflanzen bieten mehrere Vorteile für die Produktion von Biopharmazeutika mit niedrigen Kosten, Skalierbarkeit und Sicherheit. Die transiente Expression bietet den zusätzlichen Vorteil der kurzen Entwicklungs-und Produktionszeiten, sondern Expressionsniveaus erheblich zwischen den Chargen so was zu rechtlichen Bedenken im Rahmen der guten Herstellungspraxis variieren. Wir verwendeten eine Versuchsplanung (DOE) Ansatz, die Auswirkungen der wichtigsten Faktoren wie regulatorische Elemente in dem Expressionskonstrukt, das Pflanzenwachstum und Entwicklung Parametern und den Inkubationsbedingungen während der Expression, auf die Variabilität der Expression zwischen den Chargen zu bestimmen. Wir untersuchten Pflanzen ein Modell Anti-HIV-Antikörper (2G12) und einen fluoreszierenden Marker-Protein (DsRed) ausdrückt. Wir diskutieren die Gründe für die Auswahl bestimmter Eigenschaften des Modells zu identifizieren und ihr Potenzial Einschränkungen. Das allgemeine Konzept kann leicht auf andere Probleme übertragen werden, da die Prinzipien des Modells einwieder breit anwendbar: wissensbasierte Parameterauswahl, Komplexitätsreduktion durch Aufteilung der anfängliche Problem in kleinere Module, Software-geführte Installation der optimalen Experiment Kombinationen und schrittweise Design Augmentation. Daher ist die Methode nicht nur nützlich für die Charakterisierung der Proteinexpression in Pflanzen, sondern auch für die Untersuchung von anderen komplexen Systemen fehlt eine mechanistische Beschreibung. Die Vorhersagegleichungen zur Beschreibung der Interkonnektivität zwischen Parameter können verwendet werden, um mechanistische Modelle für andere komplexe Systeme aufgebaut werden.
Introduction
Die Produktion von biopharmazeutischen Proteinen in Pflanzen ist vorteilhaft, da Pflanzen kostengünstig zu wachsen, kann die Plattform sich nur durch den Anbau von Pflanzen mehr skaliert werden, und menschliche Krankheitserreger sind nicht in der Lage zu 1,2 replizieren. Transiente Expressionsstrategien zum Beispiel auf Basis der Infiltration von Blättern mit Agrobacterium tumefaciens bietet zusätzliche Vorteile, da die Zeit zwischen dem Punkt der DNA-Zufuhr und die Abgabe eines gereinigten Produktes von Jahren auf weniger als 2 Monate 3 reduziert. Transiente Expression ist auch für die funktionelle Analyse, z. B. verwendet werden, um Gene, die für ihre Fähigkeit zur Ergänzung Verlust-Funktion Mutanten oder Protein-Wechselwirkungen zu untersuchen 4-6 testen. Allerdings neigen transiente Expressionsniveaus größere Charge zu Charge Variation als Expressionsniveaus in transgenen Pflanzen 7-9 zeigen. Dies verringert die Wahrscheinlichkeit, dass biopharmazeutische Fertigungsprozesse basierend auf transiente Expression wiwerde im Rahmen der guten Herstellungspraxis (GMP) genehmigt werden, weil die Reproduzierbarkeit ist ein kritischer Qualitätsmerkmal und unterliegt Risikobewertung 10. Solche Variation können auch alle Interaktionen, die Forscher wollen untersuchen, zu maskieren. Deshalb machten wir uns auf die wichtigsten Faktoren, die transiente Expression in Pflanzen zu beeinflussen und eine hochwertige quantitative Vorhersagemodell zu bauen identifizieren.
Das Ein-Faktor-at-a-time (OFAT)-Ansatz wird oft verwendet, um die Auswirkungen (Wirkung) bestimmter Parameter (Faktoren) auf das Ergebnis (Antwort) eines Experiments 11 charakterisieren. Aber das ist nicht optimal, weil die einzelnen Tests (läuft) während einer Untersuchung (Experiment) werden wie Perlen auf einer Schnur durch die Potentialfläche durch die Faktoren, die getestet (design space) werden aufgespannt ausgerichtet werden. Die Abdeckung des Bauraums und somit den Grad der Informationen aus dem Experiment abgeleitetenniedrig, wie in Fig. 1A 12 gezeigt. Ferner können Abhängigkeiten zwischen verschiedenen Faktoren (Faktor-Wechselwirkungen) verborgen bleiben, was zu schlechter Modelle und / oder der Vorhersage falsch Optima, wie in 1B 13 gezeigt.
Die oben beschriebenen Nachteile können durch Verwendung eines Versuchsplanung (DOE) Methode, bei der die Durchläufe eines Experiments sind mehr gleichmäßig über den Entwurfsraum verstreut, was bedeutet, dass mehr als ein Faktor zwischen zwei Läufen 14 variiert vermieden werden. Es gibt spezielle Ausführungen für Mischungen, Screening Faktoren (Faktoren-Modelle) und die Quantifizierung der Auswirkungen auf Faktor Antworten (Response Surface Methoden, RSM s) 15. Weiterhin kann RSMs als zentrale Verbund Designs realisiert werden, sondern kann auch wirksam durch Verwendung spezieller Software, die Kriterien für die Auswahl der Läufe gelten erzielt werden. Beispielsweise die sogenannten D-optimality Kriterium läuft so zu wählen, um den Fehler in den Koeffizienten des resultierenden Modells zu minimieren, während die IV-Optimalitätskriterium wählt Läufe, die die niedrigste Vorhersage Varianz in der gesamten Entwurfsraum 15,16 zu erreichen. Der RSM beschreiben wir hier ermöglicht die genaue Quantifizierung von transienten Proteinexpression in Pflanzen, aber es kann problemlos an jedes System mit mehreren (~ 5-8) übertragen werden numerische Faktoren (zB Temperatur, Zeit, Konzentration) und ein paar (~ 2 – 4) categoric Faktoren (z. B. Promotor, Farbe), in dem eine mechanistische Beschreibung ist nicht verfügbar oder zu komplex, zu modellieren.
Die DoE-Ansatz entstand in den Agrarwissenschaften, hat aber auf andere Bereiche ausgebreitet, weil es zu jeder Situation, wo es sinnvoll ist, die Anzahl der Durchläufe notwendig, um verlässliche Daten zu erhalten, verringern und Beschreibungsmodelle für komplexe Prozesse übertragbar. Dies wiederum hat zur Aufnahme von DoE in der "Anleitung für LED-Industrie, Q8 (R2) Pharmaceutical Development "von der Internationalen Konferenz zur Harmonisierung der technischen Anforderungen an die für die Registrierung von Humanarzneimitteln (ICH) 17 veröffentlicht. DoE wird heute weitgehend in der wissenschaftlichen Forschung und der Industrie 18 verwendet. Allerdings muss darauf während genommen werden die Planung und Durchführung des Experiments, weil die Auswahl eines falschen Polynomgrad für die Multiple-linearen Regressionsmodell (Basismodell) eine Notwendigkeit für zusätzliche Fahrten, um alle Effekte richtig zu modellieren Faktor einzuführen. Außerdem beschädigte oder fehlende Daten zu erzeugen falsche Modelle und fehlerhaft Vorhersagen, und kann sogar verhindern, dass Modellbildung Versuch wie in den Protokoll-und Diskussionsabschnitte 18 beschrieben. In der Protokollabschnitt, werden wir zunächst die wichtigsten Planungsschritte für eine RSM-basierten Versuchs und dann erklären, das Design auf der Grundlage des DoE Software DesignExpert v8.1. Aber ähnliche Designs lassen sich mit anderen Software-includi gebaut werdenng JMP, Modde und STATISTICA. Die experimentellen Verfahren werden von Anweisungen für die Datenanalyse und Auswertung folgt.
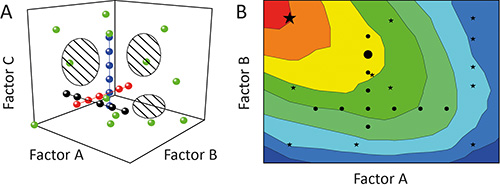
Fig. 1 ist. Vergleich von OFAT und DoE. A. Sequential Variation eines Faktors zu einer Zeit (OFAT) in einem Experiment (schwarz, rot und blau Kreise) erreicht eine geringe Abdeckung der Designraum (schraffierte Regionen). Im Gegensatz dazu die Variation von mehr als einem Faktor zu einem Zeitpunkt unter Verwendung der Versuchsplanung (DOE) Strategie (grüne Kreise) erhöht die Reichweite und damit die Genauigkeit der resultierenden Modelle. B. Das vorgespannte Design Raumabdeckung bedeutet, dass OFAT Experimente (schwarze Kreise) kann auch fehlschlagen, um optimale Betriebsbereiche (rot) zu erkennen und vorherzusagen, suboptimale Lösungen (großer schwarzer Kreis), während DoE strategisches (schwarze Sterne) sind eher zu bevorzugten Bedingungen (große schwarze Sterne) zu identifizieren.
Protocol
Representative Results
Discussion
Jedes Experiment erfordert eine sorgfältige Planung, da die Ressourcen sind oft knapp und teuer. Dies gilt insbesondere für DoE Strategien, weil Fehler in der Planungsphase (z. B. Auswahl eines Basismodell, die nicht alle wesentlichen Faktor Wechselwirkungen einschließen) im wesentlichen die Vorhersagekraft der resultierenden Modelle verringern und damit entwerten das gesamte Experiment. Allerdings können diese Fehler leicht durch folgenden grundlegenden Verfahren vermieden werden.
…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Die Autoren danken Herrn Dr. Thomas Rademacher für die Bereitstellung der pPAM Pflanzenexpressionsvektor und Ibrahim Al Amedi zur Kultivierung der Tabakpflanzen in dieser Studie verwendet. Wir möchten Dr. Richard M. Twyman für seine Unterstützung bei der Bearbeitung des Manuskripts danken. Diese Arbeit wurde zum Teil durch den European Research Council Advanced Grant "Future-Pharma" gefördert, Vorschlagsnummer 269110 und der Fraunhofer-Zukunftsstiftung (Fraunhofer-Zukunftsstiftung).
Materials
| Design-Expert(R) 8 | Stat-Ease, Inc. | n.a. | DoE software |
| Tryptone | Carl Roth GmbH | 8952.2 | Media component |
| Yeast extract | Carl Roth GmbH | 2363.2 | Media component |
| Sodium chloride | Carl Roth GmbH | P029.2 | Media component |
| Ampicillin | Carl Roth GmbH | K029.2 | Antibiotic |
| Agar-Agar | Carl Roth GmbH | 5210.2 | Media component |
| Escherichia coli K12 DH5a | Life technologies | 18263-012 | Microorganism |
| pPAM | GenBank | AY027531 | Cloning/expression vector; |
| NucleoSpin Plasmid | MACHEREY-NAGEL GmbH | 740588.250 | Plasmid DNA isolation kit |
| NucleoSpin Gel and PCR Clean-up | MACHEREY-NAGEL GmbH | 740609.250 | Plasmid DNA purification kit |
| NanoDrop 2000 | Thermo Scientific | n.a. | Spectrophotometer |
| NcoI | New England Biolabs Inc. | R3193L | Restrictionendonuclease |
| EcoRI | New England Biolabs Inc. | R3101L | Restrictionendonuclease |
| AscI | New England Biolabs Inc. | R0558L | Restrictionendonuclease |
| NEB 4 | New England Biolabs Inc. | B7004S | Restrictionendonuclease buffer |
| TRIS | Carl Roth GmbH | 4855.3 | Media component |
| Disodium tetraborate | Carl Roth GmbH | 4403.3 | Media component |
| EDTA | Carl Roth GmbH | 8040.2 | Media component |
| Agarose | Carl Roth GmbH | 6352.4 | Media component |
| Bromophenol blue | Carl Roth GmbH | A512.1 | Color indicator |
| Xylene cyanol | Carl Roth GmbH | A513.1 | Color indicator |
| Glycerol | Carl Roth GmbH | 7530.2 | Media component |
| Mini-Sub Cell GT Cell | BioRad | 170-4406 | Gel electrophoresis chamber |
| Agrobacterium tumefaciens strain GV3101:pMP90RK | DSMZ | 12365 | Microorganism |
| Electroporator 2510 | Eppendorf | 4307000.658 | Electroporator |
| Beef extract | Carl Roth GmbH | X975.2 | Media component |
| Peptone | Carl Roth GmbH | 2365.2 | Media component |
| Sucrose | Carl Roth GmbH | 4621.2 | Media component |
| Magnesium sulfate | Carl Roth GmbH | 0261.3 | Media component |
| Carbenicillin | Carl Roth GmbH | 6344.2 | Antibiotic |
| Kanamycin | Carl Roth GmbH | T832.3 | Antibiotic |
| Rifampicin | Carl Roth GmbH | 4163.2 | Antibiotic |
| FWD primer | Eurofins MWG Operon | n.a. | CCT CAG GAA GAG CAA TAC |
| REV primer | Eurofins MWG Operon | n.a. | CCA AAG CGA GTA CAC AAC |
| 2720 Thermal cycler | Applied Biosystems | 4359659 | Thermocycler |
| RNAfold webserver | University of Vienna | n.a. | Software |
| Ferty 2 Mega | Kammlott | 5.220072 | Fertilizer |
| Grodan Rockwool Cubes 10x10cm | Grodan | n.a. | Rockwool block |
| Greenhouse | n.a. | n.a. | For plant cultivation |
| Phytotron | Ilka Zell | n.a. | For plant cultivation |
| Omnifix-F Solo | B. Braun | 6064204 | Syringe |
| Murashige and Skoog salts | Duchefa | M 0222.0010 | Media component |
| Glucose | Carl Roth GmbH | 6780.2 | Media component |
| Acetosyringone | Sigma-Aldrich | D134406-5G | Phytohormon analogon |
| BioPhotometer plus | Eppendorf | 6132 000.008 | Photometer |
| Osram cool white 36 W | Osram | 4930440 | Light source |
| Disodium phosphate | Carl Roth GmbH | 4984.3 | Media component |
| Centrifuge 5415D | Eppendorf | 5424 000.410 | Centrifuge |
| Forma -86C ULT freezer | ThermoFisher | 88400 | Freezer |
| Synergy HT | BioTek | SIAFRT | Fluorescence plate reader |
| Biacore T200 | GE Healthcare | n.a. | SPR device |
| Protein A | Life technologies | 10-1006 | Antibody binding protein |
| HEPES | Carl Roth GmbH | 9105.3 | Media component |
| Tween-20 | Carl Roth GmbH | 9127.3 | Media component |
| 2G12 antibody | Polymun | AB002 | Reference antibody |
References
- Fischer, R., Emans, N. Molecular farming of pharmaceutical proteins. Transgenic research. 9, 277-299 (2000).
- Commandeur, U., Twyman, R. M., Fischer, R. The biosafety of molecular farming in plants. AgBiotechNet. 5, 9 (2003).
- Shoji, Y., et al. A plant-based system for rapid production of influenza vaccine antigens. Influenza Other Resp. 6, 204-210 (2012).
- Goodin, M. M., Zaitlin, D., Naidu, R. A., Lommel, S. A. Nicotiana benthamiana: Its history and future as a model for plant-pathogen interactions. Mol Plant Microbe In. 21, 1015-1026 (2008).
- Berg, R. H., Beachy, R. N. Fluorescent protein applications in plants. Method Cell Biol. 85, 153 (2008).
- Chung, S. M., Vaidya, M., Tzfira, T. Agrobacterium is not alone: gene transfer to plants by viruses and other bacteria. Trends in plant science. 11, 1-4 (2006).
- Sheludko, Y. V., Sindarovska, Y. R., Gerasymenko, I. M., Bannikova, M. A., Kuchuk, N. V. Comparison of several Nicotiana species as hosts for high-scale Agrobacterium-mediated transient expression. Biotechnology and Bioengineering. 96, 608-614 (2007).
- Wydro, M., Kozubek, E., Lehmann, P. Optimization of transient Agrobacterium-mediated gene expression system in leaves of Nicotiana benthamiana. Acta Biochimica Polonica. 53, 289-298 (2006).
- Buyel, J. F., Fischer, R. Processing heterogeneous biomass: Overcoming the hurdles in model building. Bioengineered. 4, (2013).
- Fischer, R., Schillberg, S., Hellwig, S., Twyman, R. M., Drossard, J. GMP issues for recombinant plant-derived pharmaceutical proteins. Biotechnol Adv. 30, 434-439 (2012).
- Daniel, C. One-at-a-time plans. Journal of the American Statistical Association. 68, 353-360 (1973).
- Czitrom, V. One-Factor-at-a-Time versus Designed Experiments The American Statistician. 53, 6 (1999).
- Anderson, M. J., Kraber, S. L. Keys to successful designed experiments. ASQ – The global voice of quality. 6, 6 (1999).
- Montgomery, D. C. . Design and Analysis of Experiments. , (2007).
- Myers, R. H., Montgomery, D. C., Anderson-Cook, C. M. . Response Surface Methodology: Process and Product Optimization Using Designed Experiments. , (2009).
- Piepel, G. F. Programs for generating extreme vertices and centroids of linearly constrained experimental regions. J Qual Technol. 20, 15 (1988).
- . . FDA. , (2009).
- Shivhare, M., McCreath, G. Practical Considerations for DoE Implementation in Quality By Design. BioProcess International. 8, 9 (2010).
- Buyel, J. F., Fischer, R. Predictive models for transient protein expression in tobacco (Nicotiana tabacum L.) can optimize process time, yield, and downstream costs. Biotechnology and bioengineering. 109, 2575-2588 (2012).
- Buyel, J. F., Kaever, T., Buyel, J. J., Fischer, R. Predictive models for the accumulation of a fluorescent marker protein in tobacco leaves according to the promoter/5’UTR combination. Biotechnology and bioengineering. 110, 471-482 (2013).
- Anderson, M. J., Whitcomb, P. J. . DOE Simplified: Practical Tools for Effective Experimentation. , (2000).
- Anderson, M. J., Whitcomb, P. J. . Response Surface Methods Simplified. , (2005).
- De Gryze, S., Langhans, I., Vandebroek, M. Using the correct intervals for prediction: A tutorial on tolerance intervals for ordinary least-squares regression. Chemometr Intell Lab. 87, 147-154 (2007).
- . . Plasmid DNA purification User manual. , (2012).
- . . PCR clean-up Gel extraction User manual. , (2012).
- . . Quick Ligation Protocol. 4, (2009).
- Inoue, H., Nojima, H., Okayama, H. High-Efficiency Transformation of Escherichia-Coli with Plasmids. Gene. 96, 23-28 (1990).
- Main, G. D., Reynolds, S., Gartland, J. S. Electroporation protocols for Agrobacterium. Methods in Molecular Biology. 44, 405-412 (1995).
- Gruber, A. R., Lorenz, R., Bernhart, S. H., Neubock, R., Hofacker, I. L. The Vienna RNA websuite. Nucleic acids research. 36, 70-74 (2008).
- Howell, S., Kenmore, M., Kirkland, M., Badley, R. A. High-density immobilization of an antibody fragment to a carboxymethylated dextran-linked biosensor surface. J Mol Recognit. 11, 200-203 (1998).
- Newcombe, A. R., et al. Evaluation of a biosensor assay to quantify polyclonal IgG in ovine serum used for the production of biotherapeutic antibody fragments. Process Biochem. 41, 842-847 (2006).
- Peixoto, J. L. Hierarchical Variable Selection in Polynomial Regression-Models. Am Stat. 41, 311-313 (1987).
- Peixoto, J. L. A Property of Well-Formulated Polynomial Regression-Models. Am Stat. 44, 26-30 (1990).
- Sanders, P. R., Winter, J. A., Barnason, A. R., Rogers, S. G., Fraley, R. T. Comparison of cauliflower mosaic virus 35S and nopaline synthase promoters in transgenic plants. Nucleic acids research. 15, 1543-1558 (1987).
- Ma, J. K. C., et al. Generation and Assembly of Secretory Antibodies in Plants. Science. 268, 716-719 (1995).
- Wycoff, K. L. Secretory IgA antibodies from plants. Curr Pharm Design. 11, 2429-2437 (2005).
- Pace, C. N., Vajdos, F., Fee, L., Grimsley, G., Gray, T. How to measure and predict the molar absorption coefficient of a protein. Protein Sci. 4, 2411-2423 (1995).

