Caratterizzazione dei sistemi complessi Utilizzando il Design of Experiments Approccio: Transient Protein Expression in tabacco come un caso di studio
Summary
Descriviamo un disegno di esperimenti approccio che può essere utilizzato per determinare e modellare l'influenza del transgene elementi regolatori, parametri di crescita e sviluppo delle piante, e condizioni di incubazione sulla espressione transiente di anticorpi monoclonali e proteine reporter nelle piante.
Abstract
Le piante offrono molteplici vantaggi per la produzione di biofarmaci tra cui bassi costi, scalabilità e sicurezza. Espressione transiente offre il vantaggio supplementare di brevi tempi di sviluppo e di produzione, ma i livelli di espressione può variare notevolmente tra i lotti dando così luogo a preoccupazioni di regolamentazione nel contesto delle buone prassi di fabbricazione. Abbiamo usato un disegno degli esperimenti (DoE) approccio per determinare l'impatto dei principali fattori, quali elementi di regolamentazione del costrutto di espressione, la crescita delle piante e parametri di sviluppo, e le condizioni di incubazione durante espressione, sulla variabilità di espressione tra i lotti. Abbiamo testato piante che esprimono un modello anti-HIV anticorpo monoclonale (2G12) e una proteina marcatore fluorescente (DsRed). Discutiamo il razionale per la selezione di alcune proprietà del modello e identificare i suoi potenziali limiti. L'approccio generale può essere facilmente trasferito ad altri problemi perché i principi del modello unre ampiamente applicabile: la selezione dei parametri basata sulla conoscenza, la riduzione della complessità suddividendo il problema iniziale in moduli più piccoli, l'installazione guidata dal software di combinazioni ottimali di esperimento e graduale aumento del design. Pertanto, il metodo non è solo utile per caratterizzare l'espressione della proteina nelle piante, ma anche per la ricerca di altri sistemi complessi privi descrizione meccanicistica. Le equazioni predittive che descrivono l'interconnettività tra i parametri possono essere utilizzati per stabilire modelli meccanicistici per altri sistemi complessi.
Introduction
La produzione di proteine biofarmaceutiche nelle piante è vantaggiosa perché le piante sono economici a crescere, la piattaforma può essere scalata solo crescendo più piante e patogeni umani sono in grado di replicare 1,2. Strategie espressione transiente basate per esempio sulla infiltrazione di foglie con Agrobacterium tumefaciens offre ulteriori vantaggi perché il tempo tra il punto di consegna del DNA e la consegna di un prodotto purificato è ridotto da anni a meno di 2 mesi 3. Espressione transiente è utilizzato anche per l'analisi funzionale, ad esempio per testare geni per la loro capacità di complementare perdita di funzione mutanti o per indagare le interazioni proteina 4-6. Tuttavia, i livelli di espressione transiente tendono a mostrare una maggiore variazione da lotto a lotto di livelli di espressione in piante transgeniche 7-9. Ciò riduce la probabilità che i processi di produzione biofarmaceutica basati su espressione transiente will essere approvato nel contesto delle buone prassi di fabbricazione (GMP), perché la riproducibilità è un attributo di qualità critico ed è soggetta a valutazione del rischio 10. Tale variazione può anche mascherare tutte le interazioni che i ricercatori intendono studiare. Pertanto, abbiamo deciso di identificare i principali fattori che influenzano i livelli di espressione transitori nelle piante e per costruire un modello predittivo quantitativo di alta qualità.
L'approccio a un fattore-at-a-tempo (OFAT) è spesso usato per caratterizzare l'impatto (effetto) di alcuni parametri (fattori) sull'esito (risposta) di un esperimento 11. Ma questo è ottimale perché le prove individuali (va) durante l'indagine (esperimento) saranno allineati come perle di una collana attraverso l'area potenziale attraversato dai fattori che vengono testati (spazio di progettazione). La copertura dello spazio di progettazione e quindi il grado di informazioni derivate da dell'esperimento èbasso, come mostrato nella Figura 1A 12. Inoltre, interdipendenze tra diversi fattori (interazioni factor) possono rimanere nascosti conseguente modelli poveri e / o la previsione di falsa Optima, come mostrato nella Figura 1B 13.
Gli inconvenienti sopra descritti possono essere evitati utilizzando un disegno di avvicinamento esperimenti (DOE) in cui viene eseguito su un esperimento sono sparsi in modo più uniforme in tutto lo spazio di progettazione, il che significa che più di un fattore è variata tra due piste 14. Ci sono disegni specializzati per le miscele, screening fattori (disegni fattoriali) e la quantificazione degli impatti dei fattori sulle risposte (metodi di superficie di risposta, RSM s) 15. Inoltre, RSMS possono essere realizzati come disegni centrali-composito ma possono anche essere realizzati efficacemente con software specializzato che può applicare diversi criteri per la selezione di piste. Ad esempio, il cosiddetto D-optimality criterio selezionerà corre in modo da minimizzare l'errore nei coefficienti del modello risultante, mentre il criterio IV-ottimalità seleziona piste che raggiungono la varianza previsione basso tutto lo spazio di progettazione 15,16. La RSM descriviamo qui permette la quantificazione precisa espressione transiente della proteina nelle piante, ma può essere facilmente trasferito su qualsiasi sistema che coinvolge vari (~ 5-8) fattori numerici (ad esempio temperatura, tempo, concentrazione) e alcuni (~ 2 – 4) fattori categorici (es. promoter, colore) in cui una descrizione meccanicistica non è disponibile o troppo complesso per modellare.
L'approccio DoE origine nelle scienze agrarie, ma si è diffuso ad altre zone, perché è trasferibile a qualsiasi situazione in cui è utile per ridurre il numero di corse necessarie per ottenere dati affidabili e generare modelli descrittivi per processi complessi. Questo a sua volta ha portato all'inserimento del DoE nella "Guida perIndustria, Q8 (R2) Pharmaceutical Development ", pubblicato dalla Conferenza internazionale sull'armonizzazione dei requisiti tecnici per la registrazione dei prodotti farmaceutici per uso umano (ICH) 17. DoE è ormai ampiamente utilizzato nella ricerca scientifica e l'industria 18. Tuttavia, la cura deve essere presa durante la pianificazione e l'esecuzione dell'esperimento, perché la scelta di un grado del polinomio improprio per il modello multi-lineare di regressione (modello base) possono introdurre la necessità di corse aggiuntive per modellare correttamente tutti gli effetti dei fattori. Inoltre, danneggiati o dati mancanti generano modelli errati e viziata previsioni, e può anche impedire qualsiasi tentativo di costruzione del modello, come descritto nel protocollo e discussione sezioni 18. Nella sezione Protocollo, ci sarà inizialmente stabilito le fasi di pianificazione più importanti per un esperimento a base di RSM e poi spiegare il progetto sulla base del DoE software DesignExpert v8.1., ma disegni simili possono essere costruiti con altri includi softwareng JMP, Modde, e STATISTICA. Le procedure sperimentali sono seguite da istruzioni per l'analisi e la valutazione dei dati.
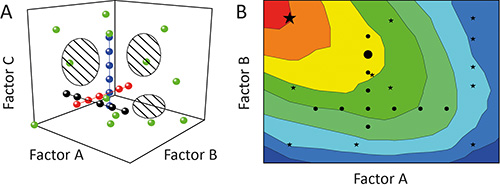
Figura 1. Confronto di OFAT e DoE. A. Variazione sequenziale di un fattore alla volta (OFAT) in un esperimento (nero, rosso e blu cerchi) raggiunge una bassa copertura dello spazio di progettazione (regioni tratteggiate). Al contrario, la variazione di più di un fattore alla volta utilizzando la progettazione di esperimenti (DoE) strategia (cerchi verdi) migliora la copertura e quindi la precisione dei modelli risultanti. B. La copertura dello spazio di progettazione di parte significa che gli esperimenti OFAT (cerchi neri) può anche non riuscire a identificare le regioni operative ottimali (rosso) e prevedere soluzioni sub-ottimali (grande cerchio nero), mentre DoE strategies (stelle nere) sono più propensi a identificare le condizioni preferibili (grande black star).
Protocol
Representative Results
Discussion
Ogni esperimento richiede un'attenta pianificazione, perché le risorse sono spesso scarse e costose. Ciò è particolarmente vero per le strategie DoE perché errori durante la fase di pianificazione (ad esempio la selezione di un modello di base che non copre tutte le interazioni significative dei fattori) possono diminuire sostanzialmente il potere predittivo dei modelli risultanti e quindi svalutare l'intero esperimento. Tuttavia, questi errori possono essere facilmente evitati seguendo procedure di…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Gli autori sono grati al Dott. Thomas Rademacher per fornire il PPAM pianta vettore di espressione e Ibrahim Al Amedi per la coltivazione delle piante di tabacco utilizzate in questo studio. Vorremmo ringraziare il Dott. Richard M. Twyman per la sua assistenza con la modifica del manoscritto. Questo lavoro è stato in parte finanziato dalla Advanced Grant "Future-Pharma" Consiglio europeo della ricerca, il numero di proposta 269.110 e il Fraunhofer Zukunftsstiftung (Fraunhofer Future Foundation).
Materials
| Design-Expert(R) 8 | Stat-Ease, Inc. | n.a. | DoE software |
| Tryptone | Carl Roth GmbH | 8952.2 | Media component |
| Yeast extract | Carl Roth GmbH | 2363.2 | Media component |
| Sodium chloride | Carl Roth GmbH | P029.2 | Media component |
| Ampicillin | Carl Roth GmbH | K029.2 | Antibiotic |
| Agar-Agar | Carl Roth GmbH | 5210.2 | Media component |
| Escherichia coli K12 DH5a | Life technologies | 18263-012 | Microorganism |
| pPAM | GenBank | AY027531 | Cloning/expression vector; |
| NucleoSpin Plasmid | MACHEREY-NAGEL GmbH | 740588.250 | Plasmid DNA isolation kit |
| NucleoSpin Gel and PCR Clean-up | MACHEREY-NAGEL GmbH | 740609.250 | Plasmid DNA purification kit |
| NanoDrop 2000 | Thermo Scientific | n.a. | Spectrophotometer |
| NcoI | New England Biolabs Inc. | R3193L | Restrictionendonuclease |
| EcoRI | New England Biolabs Inc. | R3101L | Restrictionendonuclease |
| AscI | New England Biolabs Inc. | R0558L | Restrictionendonuclease |
| NEB 4 | New England Biolabs Inc. | B7004S | Restrictionendonuclease buffer |
| TRIS | Carl Roth GmbH | 4855.3 | Media component |
| Disodium tetraborate | Carl Roth GmbH | 4403.3 | Media component |
| EDTA | Carl Roth GmbH | 8040.2 | Media component |
| Agarose | Carl Roth GmbH | 6352.4 | Media component |
| Bromophenol blue | Carl Roth GmbH | A512.1 | Color indicator |
| Xylene cyanol | Carl Roth GmbH | A513.1 | Color indicator |
| Glycerol | Carl Roth GmbH | 7530.2 | Media component |
| Mini-Sub Cell GT Cell | BioRad | 170-4406 | Gel electrophoresis chamber |
| Agrobacterium tumefaciens strain GV3101:pMP90RK | DSMZ | 12365 | Microorganism |
| Electroporator 2510 | Eppendorf | 4307000.658 | Electroporator |
| Beef extract | Carl Roth GmbH | X975.2 | Media component |
| Peptone | Carl Roth GmbH | 2365.2 | Media component |
| Sucrose | Carl Roth GmbH | 4621.2 | Media component |
| Magnesium sulfate | Carl Roth GmbH | 0261.3 | Media component |
| Carbenicillin | Carl Roth GmbH | 6344.2 | Antibiotic |
| Kanamycin | Carl Roth GmbH | T832.3 | Antibiotic |
| Rifampicin | Carl Roth GmbH | 4163.2 | Antibiotic |
| FWD primer | Eurofins MWG Operon | n.a. | CCT CAG GAA GAG CAA TAC |
| REV primer | Eurofins MWG Operon | n.a. | CCA AAG CGA GTA CAC AAC |
| 2720 Thermal cycler | Applied Biosystems | 4359659 | Thermocycler |
| RNAfold webserver | University of Vienna | n.a. | Software |
| Ferty 2 Mega | Kammlott | 5.220072 | Fertilizer |
| Grodan Rockwool Cubes 10x10cm | Grodan | n.a. | Rockwool block |
| Greenhouse | n.a. | n.a. | For plant cultivation |
| Phytotron | Ilka Zell | n.a. | For plant cultivation |
| Omnifix-F Solo | B. Braun | 6064204 | Syringe |
| Murashige and Skoog salts | Duchefa | M 0222.0010 | Media component |
| Glucose | Carl Roth GmbH | 6780.2 | Media component |
| Acetosyringone | Sigma-Aldrich | D134406-5G | Phytohormon analogon |
| BioPhotometer plus | Eppendorf | 6132 000.008 | Photometer |
| Osram cool white 36 W | Osram | 4930440 | Light source |
| Disodium phosphate | Carl Roth GmbH | 4984.3 | Media component |
| Centrifuge 5415D | Eppendorf | 5424 000.410 | Centrifuge |
| Forma -86C ULT freezer | ThermoFisher | 88400 | Freezer |
| Synergy HT | BioTek | SIAFRT | Fluorescence plate reader |
| Biacore T200 | GE Healthcare | n.a. | SPR device |
| Protein A | Life technologies | 10-1006 | Antibody binding protein |
| HEPES | Carl Roth GmbH | 9105.3 | Media component |
| Tween-20 | Carl Roth GmbH | 9127.3 | Media component |
| 2G12 antibody | Polymun | AB002 | Reference antibody |
References
- Fischer, R., Emans, N. Molecular farming of pharmaceutical proteins. Transgenic research. 9, 277-299 (2000).
- Commandeur, U., Twyman, R. M., Fischer, R. The biosafety of molecular farming in plants. AgBiotechNet. 5, 9 (2003).
- Shoji, Y., et al. A plant-based system for rapid production of influenza vaccine antigens. Influenza Other Resp. 6, 204-210 (2012).
- Goodin, M. M., Zaitlin, D., Naidu, R. A., Lommel, S. A. Nicotiana benthamiana: Its history and future as a model for plant-pathogen interactions. Mol Plant Microbe In. 21, 1015-1026 (2008).
- Berg, R. H., Beachy, R. N. Fluorescent protein applications in plants. Method Cell Biol. 85, 153 (2008).
- Chung, S. M., Vaidya, M., Tzfira, T. Agrobacterium is not alone: gene transfer to plants by viruses and other bacteria. Trends in plant science. 11, 1-4 (2006).
- Sheludko, Y. V., Sindarovska, Y. R., Gerasymenko, I. M., Bannikova, M. A., Kuchuk, N. V. Comparison of several Nicotiana species as hosts for high-scale Agrobacterium-mediated transient expression. Biotechnology and Bioengineering. 96, 608-614 (2007).
- Wydro, M., Kozubek, E., Lehmann, P. Optimization of transient Agrobacterium-mediated gene expression system in leaves of Nicotiana benthamiana. Acta Biochimica Polonica. 53, 289-298 (2006).
- Buyel, J. F., Fischer, R. Processing heterogeneous biomass: Overcoming the hurdles in model building. Bioengineered. 4, (2013).
- Fischer, R., Schillberg, S., Hellwig, S., Twyman, R. M., Drossard, J. GMP issues for recombinant plant-derived pharmaceutical proteins. Biotechnol Adv. 30, 434-439 (2012).
- Daniel, C. One-at-a-time plans. Journal of the American Statistical Association. 68, 353-360 (1973).
- Czitrom, V. One-Factor-at-a-Time versus Designed Experiments The American Statistician. 53, 6 (1999).
- Anderson, M. J., Kraber, S. L. Keys to successful designed experiments. ASQ – The global voice of quality. 6, 6 (1999).
- Montgomery, D. C. . Design and Analysis of Experiments. , (2007).
- Myers, R. H., Montgomery, D. C., Anderson-Cook, C. M. . Response Surface Methodology: Process and Product Optimization Using Designed Experiments. , (2009).
- Piepel, G. F. Programs for generating extreme vertices and centroids of linearly constrained experimental regions. J Qual Technol. 20, 15 (1988).
- . . FDA. , (2009).
- Shivhare, M., McCreath, G. Practical Considerations for DoE Implementation in Quality By Design. BioProcess International. 8, 9 (2010).
- Buyel, J. F., Fischer, R. Predictive models for transient protein expression in tobacco (Nicotiana tabacum L.) can optimize process time, yield, and downstream costs. Biotechnology and bioengineering. 109, 2575-2588 (2012).
- Buyel, J. F., Kaever, T., Buyel, J. J., Fischer, R. Predictive models for the accumulation of a fluorescent marker protein in tobacco leaves according to the promoter/5’UTR combination. Biotechnology and bioengineering. 110, 471-482 (2013).
- Anderson, M. J., Whitcomb, P. J. . DOE Simplified: Practical Tools for Effective Experimentation. , (2000).
- Anderson, M. J., Whitcomb, P. J. . Response Surface Methods Simplified. , (2005).
- De Gryze, S., Langhans, I., Vandebroek, M. Using the correct intervals for prediction: A tutorial on tolerance intervals for ordinary least-squares regression. Chemometr Intell Lab. 87, 147-154 (2007).
- . . Plasmid DNA purification User manual. , (2012).
- . . PCR clean-up Gel extraction User manual. , (2012).
- . . Quick Ligation Protocol. 4, (2009).
- Inoue, H., Nojima, H., Okayama, H. High-Efficiency Transformation of Escherichia-Coli with Plasmids. Gene. 96, 23-28 (1990).
- Main, G. D., Reynolds, S., Gartland, J. S. Electroporation protocols for Agrobacterium. Methods in Molecular Biology. 44, 405-412 (1995).
- Gruber, A. R., Lorenz, R., Bernhart, S. H., Neubock, R., Hofacker, I. L. The Vienna RNA websuite. Nucleic acids research. 36, 70-74 (2008).
- Howell, S., Kenmore, M., Kirkland, M., Badley, R. A. High-density immobilization of an antibody fragment to a carboxymethylated dextran-linked biosensor surface. J Mol Recognit. 11, 200-203 (1998).
- Newcombe, A. R., et al. Evaluation of a biosensor assay to quantify polyclonal IgG in ovine serum used for the production of biotherapeutic antibody fragments. Process Biochem. 41, 842-847 (2006).
- Peixoto, J. L. Hierarchical Variable Selection in Polynomial Regression-Models. Am Stat. 41, 311-313 (1987).
- Peixoto, J. L. A Property of Well-Formulated Polynomial Regression-Models. Am Stat. 44, 26-30 (1990).
- Sanders, P. R., Winter, J. A., Barnason, A. R., Rogers, S. G., Fraley, R. T. Comparison of cauliflower mosaic virus 35S and nopaline synthase promoters in transgenic plants. Nucleic acids research. 15, 1543-1558 (1987).
- Ma, J. K. C., et al. Generation and Assembly of Secretory Antibodies in Plants. Science. 268, 716-719 (1995).
- Wycoff, K. L. Secretory IgA antibodies from plants. Curr Pharm Design. 11, 2429-2437 (2005).
- Pace, C. N., Vajdos, F., Fee, L., Grimsley, G., Gray, T. How to measure and predict the molar absorption coefficient of a protein. Protein Sci. 4, 2411-2423 (1995).

