Caracterización de Sistemas Complejos Uso del Diseño de Experimentos Enfoque: Expresión de Proteínas transitoria en tabaco como un estudio de caso
Summary
Se describe un diseño de enfoque experimentos que se puede utilizar para determinar y modelar la influencia de los elementos reguladores de transgenes, los parámetros de crecimiento y desarrollo de la planta, y las condiciones de incubación en la expresión transitoria de los anticuerpos monoclonales y proteínas reportero en plantas.
Abstract
Las plantas proporcionan múltiples beneficios para la producción de productos biofarmacéuticos incluyendo bajos costos, escalabilidad y seguridad. La expresión transitoria ofrece la ventaja adicional de desarrollo a corto y tiempos de producción, pero los niveles de expresión puede variar significativamente entre los lotes, dando así lugar a preocupaciones de reglamentación en el contexto de las buenas prácticas de fabricación. Se utilizó un diseño de enfoque experimentos (DoE) para determinar el impacto de los factores más importantes, como elementos reguladores en la construcción de expresión, crecimiento de las plantas y de los parámetros de desarrollo y las condiciones de incubación durante la expresión, en la variabilidad de la expresión entre lotes. Hemos probado plantas que expresan un anticuerpo monoclonal anti-VIH modelo (2G12) y una proteína marcadora fluorescente (DsRed). Se discute la justificación de la selección de ciertas propiedades del modelo e identificar sus posibles limitaciones. El enfoque general puede ser fácilmente transferido a otros problemas debido a que los principios del modelo de unre ampliamente aplicable: selección de parámetros basada en el conocimiento, la reducción de la complejidad mediante la división del problema inicial en módulos más pequeños, la instalación del software guiado de las combinaciones óptimas de experimentos y el aumento de diseño por etapas. Por lo tanto, la metodología no sólo es útil para la caracterización de la expresión de proteínas en las plantas, sino también para la investigación de otros sistemas complejos que carecen de una descripción mecanicista. Las ecuaciones de predicción que describen la interconectividad entre los parámetros pueden ser utilizados para establecer modelos mecánicos para otros sistemas complejos.
Introduction
La producción de proteínas biofarmacéuticas en las plantas es ventajoso porque las plantas son de bajo costo para crecer, la plataforma puede ser ampliado con sólo crece más plantas, y los patógenos humanos son incapaces de replicarse 1,2. Las estrategias de expresión transitoria basados por ejemplo en la infiltración de las hojas con Agrobacterium tumefaciens proporciona beneficios adicionales ya que el tiempo entre el punto de entrega de ADN y la entrega de un producto purificado se reduce de año a menos de 2 meses 3. La expresión transitoria también se utiliza para el análisis funcional, por ejemplo, para probar los genes por su capacidad para complementar mutantes de pérdida de función o para investigar las interacciones de proteínas 4-6. Sin embargo, los niveles de expresión transitoria tienden a mostrar una mayor variación de lote a lote de los niveles de expresión en plantas transgénicas 7-9. Esto reduce la probabilidad de que los procesos de fabricación biofarmacéutica basan en wi expresión transitoriall ser aprobado en el marco de las buenas prácticas de fabricación (GMP), ya que la reproducibilidad es un atributo crítico calidad y está sujeta a la evaluación de riesgos 10. Esta variación también puede enmascarar cualquier interacción que los investigadores tienen la intención de investigar. Por lo tanto, nos propusimos identificar los principales factores que afectan los niveles de expresión transitoria en plantas y construir un modelo de predicción cuantitativa de alta calidad.
El enfoque de un factor-en-un-tiempo (OFAT) se utiliza a menudo para caracterizar el impacto (efecto) de ciertos parámetros (factores) sobre el resultado (respuesta) de un experimento 11. Pero esto no es óptima debido a que las pruebas individuales (corre) durante una investigación (experimento) estarán alineados como perlas en una cuerda a través del área potencial abarcado por los factores que se evalúan (espacio de diseño). La cobertura del espacio de diseño y por lo tanto el grado de información derivada de la experimento esbajo, como se muestra en la Figura 1A 12. Además, las interdependencias entre los diferentes factores (interacciones de factores) pueden permanecer ocultos que resulta en modelos pobres y / o la predicción de falsa Optima, como se muestra en la Figura 1B 13.
Los inconvenientes descritos anteriormente se pueden evitar mediante el uso de un diseño de experimentos de aproximación (DoE) en la que se ejecuta de un experimento se dispersan de manera más uniforme en todo el espacio de diseño, lo que significa que más de un factor se varía entre dos carreras 14. Hay diseños especializados para las mezclas, los factores (diseños factoriales) y la cuantificación de los impactos de los factores sobre las respuestas (métodos de superficie de respuesta, RSM s) 15 de detección. Además, RSM se pueden realizar como diseños-compuesto central, pero también se puede lograr de manera efectiva mediante el uso de software especializado que puede aplicar diferentes criterios para la selección de carreras. Por ejemplo, el llamado D-optimalitcriterio y seleccionará carreras a fin de minimizar el error en los coeficientes del modelo resultante, mientras que el criterio IV-optimalidad selecciona carreras que logran la varianza de predicción más bajo en todo el espacio de diseño 15,16. El RSM como se describe aquí permite la cuantificación precisa de la expresión de proteína transitoria en plantas, pero que puede ser fácilmente transferido a cualquier sistema que implica varios (~ 5-8) factores numéricos (por ejemplo, temperatura, tiempo, concentración) y unos pocos (~ 2 – 4) factores categóricas (por ejemplo, promotor, color) en el que una descripción mecanicista no está disponible o demasiado complejo para modelar.
El enfoque DoE se originó en las ciencias agrícolas, pero se ha extendido a otras áreas, ya que es transferible a cualquier situación en la que es útil para reducir el número de ciclos necesarios para obtener datos fiables y generar modelos descriptivos de procesos complejos. Esto a su vez ha dado lugar a la inclusión del DoE en la "Guía para laIndustria, Q8 (R2) Desarrollo Farmacéutico ", publicado por la Conferencia Internacional sobre Armonización de los Requisitos Técnicos para el Registro de Productos Farmacéuticos para Uso Humano (ICH) 17. DoE ahora se utiliza ampliamente en la investigación científica y la industria 18. Sin embargo, se debe tener cuidado durante la planificación y ejecución del experimento, porque la selección de un grado del polinomio impropio para el modelo de regresión lineal múltiple (modelo base) pueden presentar una necesidad de tiradas adicionales para modelar los efectos de los factores correctamente. Además, dañados o datos faltantes generan modelos incorrectos y defectuoso predicciones, y puede incluso prevenir cualquier intento de construcción de modelos, como se describe en el protocolo y de discusión de los artículos 18. En la sección de protocolo, que inicialmente se establecerán los pasos de planificación más importantes para un experimento basado en la RSM y luego explicar el diseño basado en el DoE software v8.1 DesignExpert. Pero diseños similares se pueden construir con otros includi softwareng JMP, Modde y STATISTICA. Los procedimientos experimentales son seguidos por instrucciones para el análisis y evaluación de datos.
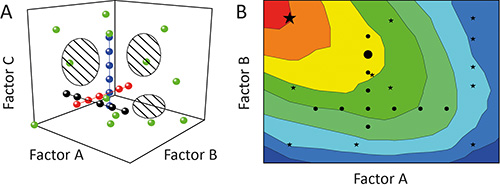
Figura 1. Comparativa de. OFAT y DoE A. Variación secuencial de un factor a la vez (OFAT) en un experimento (círculos negros, rojos y azules) logra una baja cobertura del diseño de espacio (regiones sombreadas). Por el contrario, la variación de más de un factor a la vez usando el diseño de experimentos de estrategia (DoE) (círculos verdes) mejora la cobertura y por lo tanto la precisión de los modelos resultantes. B. La cobertura de espacio de diseño sesgada significa que los experimentos OFAT (círculos negros) también pueden fallar para identificar regiones óptimas de operación (rojo) y predecir soluciones sub-óptimas (gran círculo negro), mientras que el DoE strategies (estrellas negras) son más propensos a identificar condiciones preferentes (gran estrella negro).
Protocol
Representative Results
Discussion
Cada experimento requiere una planificación cuidadosa, porque los recursos suelen ser escasos y caros. Esto es particularmente cierto para las estrategias del Departamento de energía debido a errores durante la fase de planificación (por ejemplo, la selección de un modelo base que no cubra todas las interacciones significativas factores) pueden disminuir sustancialmente la capacidad predictiva de los modelos resultantes y así devaluar todo el experimento. Sin embargo, estos errores se pueden evitar fácilm…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Los autores agradecen al Dr. Thomas Rademacher para proporcionar el vector de expresión de planta ppam e Ibrahim Al Amedi para el cultivo de las plantas de tabaco utilizadas en este estudio. Nos gustaría dar las gracias al doctor Richard M. Twyman por su ayuda con la edición del manuscrito. Este trabajo ha sido parcialmente financiado por el Advanced Grant "Future-Pharma" Consejo Europeo de Investigación, la propuesta número 269.110 y la Fraunhofer Zukunftsstiftung (Fundación Fraunhofer Futuro).
Materials
| Design-Expert(R) 8 | Stat-Ease, Inc. | n.a. | DoE software |
| Tryptone | Carl Roth GmbH | 8952.2 | Media component |
| Yeast extract | Carl Roth GmbH | 2363.2 | Media component |
| Sodium chloride | Carl Roth GmbH | P029.2 | Media component |
| Ampicillin | Carl Roth GmbH | K029.2 | Antibiotic |
| Agar-Agar | Carl Roth GmbH | 5210.2 | Media component |
| Escherichia coli K12 DH5a | Life technologies | 18263-012 | Microorganism |
| pPAM | GenBank | AY027531 | Cloning/expression vector; |
| NucleoSpin Plasmid | MACHEREY-NAGEL GmbH | 740588.250 | Plasmid DNA isolation kit |
| NucleoSpin Gel and PCR Clean-up | MACHEREY-NAGEL GmbH | 740609.250 | Plasmid DNA purification kit |
| NanoDrop 2000 | Thermo Scientific | n.a. | Spectrophotometer |
| NcoI | New England Biolabs Inc. | R3193L | Restrictionendonuclease |
| EcoRI | New England Biolabs Inc. | R3101L | Restrictionendonuclease |
| AscI | New England Biolabs Inc. | R0558L | Restrictionendonuclease |
| NEB 4 | New England Biolabs Inc. | B7004S | Restrictionendonuclease buffer |
| TRIS | Carl Roth GmbH | 4855.3 | Media component |
| Disodium tetraborate | Carl Roth GmbH | 4403.3 | Media component |
| EDTA | Carl Roth GmbH | 8040.2 | Media component |
| Agarose | Carl Roth GmbH | 6352.4 | Media component |
| Bromophenol blue | Carl Roth GmbH | A512.1 | Color indicator |
| Xylene cyanol | Carl Roth GmbH | A513.1 | Color indicator |
| Glycerol | Carl Roth GmbH | 7530.2 | Media component |
| Mini-Sub Cell GT Cell | BioRad | 170-4406 | Gel electrophoresis chamber |
| Agrobacterium tumefaciens strain GV3101:pMP90RK | DSMZ | 12365 | Microorganism |
| Electroporator 2510 | Eppendorf | 4307000.658 | Electroporator |
| Beef extract | Carl Roth GmbH | X975.2 | Media component |
| Peptone | Carl Roth GmbH | 2365.2 | Media component |
| Sucrose | Carl Roth GmbH | 4621.2 | Media component |
| Magnesium sulfate | Carl Roth GmbH | 0261.3 | Media component |
| Carbenicillin | Carl Roth GmbH | 6344.2 | Antibiotic |
| Kanamycin | Carl Roth GmbH | T832.3 | Antibiotic |
| Rifampicin | Carl Roth GmbH | 4163.2 | Antibiotic |
| FWD primer | Eurofins MWG Operon | n.a. | CCT CAG GAA GAG CAA TAC |
| REV primer | Eurofins MWG Operon | n.a. | CCA AAG CGA GTA CAC AAC |
| 2720 Thermal cycler | Applied Biosystems | 4359659 | Thermocycler |
| RNAfold webserver | University of Vienna | n.a. | Software |
| Ferty 2 Mega | Kammlott | 5.220072 | Fertilizer |
| Grodan Rockwool Cubes 10x10cm | Grodan | n.a. | Rockwool block |
| Greenhouse | n.a. | n.a. | For plant cultivation |
| Phytotron | Ilka Zell | n.a. | For plant cultivation |
| Omnifix-F Solo | B. Braun | 6064204 | Syringe |
| Murashige and Skoog salts | Duchefa | M 0222.0010 | Media component |
| Glucose | Carl Roth GmbH | 6780.2 | Media component |
| Acetosyringone | Sigma-Aldrich | D134406-5G | Phytohormon analogon |
| BioPhotometer plus | Eppendorf | 6132 000.008 | Photometer |
| Osram cool white 36 W | Osram | 4930440 | Light source |
| Disodium phosphate | Carl Roth GmbH | 4984.3 | Media component |
| Centrifuge 5415D | Eppendorf | 5424 000.410 | Centrifuge |
| Forma -86C ULT freezer | ThermoFisher | 88400 | Freezer |
| Synergy HT | BioTek | SIAFRT | Fluorescence plate reader |
| Biacore T200 | GE Healthcare | n.a. | SPR device |
| Protein A | Life technologies | 10-1006 | Antibody binding protein |
| HEPES | Carl Roth GmbH | 9105.3 | Media component |
| Tween-20 | Carl Roth GmbH | 9127.3 | Media component |
| 2G12 antibody | Polymun | AB002 | Reference antibody |
References
- Fischer, R., Emans, N. Molecular farming of pharmaceutical proteins. Transgenic research. 9, 277-299 (2000).
- Commandeur, U., Twyman, R. M., Fischer, R. The biosafety of molecular farming in plants. AgBiotechNet. 5, 9 (2003).
- Shoji, Y., et al. A plant-based system for rapid production of influenza vaccine antigens. Influenza Other Resp. 6, 204-210 (2012).
- Goodin, M. M., Zaitlin, D., Naidu, R. A., Lommel, S. A. Nicotiana benthamiana: Its history and future as a model for plant-pathogen interactions. Mol Plant Microbe In. 21, 1015-1026 (2008).
- Berg, R. H., Beachy, R. N. Fluorescent protein applications in plants. Method Cell Biol. 85, 153 (2008).
- Chung, S. M., Vaidya, M., Tzfira, T. Agrobacterium is not alone: gene transfer to plants by viruses and other bacteria. Trends in plant science. 11, 1-4 (2006).
- Sheludko, Y. V., Sindarovska, Y. R., Gerasymenko, I. M., Bannikova, M. A., Kuchuk, N. V. Comparison of several Nicotiana species as hosts for high-scale Agrobacterium-mediated transient expression. Biotechnology and Bioengineering. 96, 608-614 (2007).
- Wydro, M., Kozubek, E., Lehmann, P. Optimization of transient Agrobacterium-mediated gene expression system in leaves of Nicotiana benthamiana. Acta Biochimica Polonica. 53, 289-298 (2006).
- Buyel, J. F., Fischer, R. Processing heterogeneous biomass: Overcoming the hurdles in model building. Bioengineered. 4, (2013).
- Fischer, R., Schillberg, S., Hellwig, S., Twyman, R. M., Drossard, J. GMP issues for recombinant plant-derived pharmaceutical proteins. Biotechnol Adv. 30, 434-439 (2012).
- Daniel, C. One-at-a-time plans. Journal of the American Statistical Association. 68, 353-360 (1973).
- Czitrom, V. One-Factor-at-a-Time versus Designed Experiments The American Statistician. 53, 6 (1999).
- Anderson, M. J., Kraber, S. L. Keys to successful designed experiments. ASQ – The global voice of quality. 6, 6 (1999).
- Montgomery, D. C. . Design and Analysis of Experiments. , (2007).
- Myers, R. H., Montgomery, D. C., Anderson-Cook, C. M. . Response Surface Methodology: Process and Product Optimization Using Designed Experiments. , (2009).
- Piepel, G. F. Programs for generating extreme vertices and centroids of linearly constrained experimental regions. J Qual Technol. 20, 15 (1988).
- . . FDA. , (2009).
- Shivhare, M., McCreath, G. Practical Considerations for DoE Implementation in Quality By Design. BioProcess International. 8, 9 (2010).
- Buyel, J. F., Fischer, R. Predictive models for transient protein expression in tobacco (Nicotiana tabacum L.) can optimize process time, yield, and downstream costs. Biotechnology and bioengineering. 109, 2575-2588 (2012).
- Buyel, J. F., Kaever, T., Buyel, J. J., Fischer, R. Predictive models for the accumulation of a fluorescent marker protein in tobacco leaves according to the promoter/5’UTR combination. Biotechnology and bioengineering. 110, 471-482 (2013).
- Anderson, M. J., Whitcomb, P. J. . DOE Simplified: Practical Tools for Effective Experimentation. , (2000).
- Anderson, M. J., Whitcomb, P. J. . Response Surface Methods Simplified. , (2005).
- De Gryze, S., Langhans, I., Vandebroek, M. Using the correct intervals for prediction: A tutorial on tolerance intervals for ordinary least-squares regression. Chemometr Intell Lab. 87, 147-154 (2007).
- . . Plasmid DNA purification User manual. , (2012).
- . . PCR clean-up Gel extraction User manual. , (2012).
- . . Quick Ligation Protocol. 4, (2009).
- Inoue, H., Nojima, H., Okayama, H. High-Efficiency Transformation of Escherichia-Coli with Plasmids. Gene. 96, 23-28 (1990).
- Main, G. D., Reynolds, S., Gartland, J. S. Electroporation protocols for Agrobacterium. Methods in Molecular Biology. 44, 405-412 (1995).
- Gruber, A. R., Lorenz, R., Bernhart, S. H., Neubock, R., Hofacker, I. L. The Vienna RNA websuite. Nucleic acids research. 36, 70-74 (2008).
- Howell, S., Kenmore, M., Kirkland, M., Badley, R. A. High-density immobilization of an antibody fragment to a carboxymethylated dextran-linked biosensor surface. J Mol Recognit. 11, 200-203 (1998).
- Newcombe, A. R., et al. Evaluation of a biosensor assay to quantify polyclonal IgG in ovine serum used for the production of biotherapeutic antibody fragments. Process Biochem. 41, 842-847 (2006).
- Peixoto, J. L. Hierarchical Variable Selection in Polynomial Regression-Models. Am Stat. 41, 311-313 (1987).
- Peixoto, J. L. A Property of Well-Formulated Polynomial Regression-Models. Am Stat. 44, 26-30 (1990).
- Sanders, P. R., Winter, J. A., Barnason, A. R., Rogers, S. G., Fraley, R. T. Comparison of cauliflower mosaic virus 35S and nopaline synthase promoters in transgenic plants. Nucleic acids research. 15, 1543-1558 (1987).
- Ma, J. K. C., et al. Generation and Assembly of Secretory Antibodies in Plants. Science. 268, 716-719 (1995).
- Wycoff, K. L. Secretory IgA antibodies from plants. Curr Pharm Design. 11, 2429-2437 (2005).
- Pace, C. N., Vajdos, F., Fee, L., Grimsley, G., Gray, T. How to measure and predict the molar absorption coefficient of a protein. Protein Sci. 4, 2411-2423 (1995).

