DNA-affiniteit-gezuiverd Chip (DAP-chip) methode om Gene Doelen bepalen voor bacteriële Twee componenten Regulatory Systems
Summary
Deze video artikel beschrijft een in vitro microarray gebaseerde methode om het gen doelstellingen en bindingsplaatsen bepalen voor twee componenten systeem respons toezichthouders.
Abstract
In vivo methoden, zoals chip-chip zijn goed gevestigde technieken die worden gebruikt om de wereldwijde gendoelstellingen bepalen voor transcriptiefactoren. Ze zijn echter van weinig nut in het verkennen van bacteriële tweecomponenten regulerende systemen met ongekarakteriseerde activatie. Dergelijke systemen reguleren transcriptie wanneer geactiveerd in de aanwezigheid van unieke signalen. Aangezien deze signalen zijn vaak onbekend is, kan de in vitro microarray gebaseerde methode in deze video artikel beschreven worden gebruikt om gendoelstellingen en bindingsplaatsen voor respons toezichthouders bepalen. Deze DNA-affiniteit-gezuiverde-chip werkwijze kan worden gebruikt voor elke gezuiverde regulator een organisme met een genoom gesequenced. Het protocol omvat waarbij het gezuiverde gelabeld eiwit binden aan genomisch DNA geschoren en affiniteit zuiveren van het eiwit gebonden DNA, gevolgd door fluorescentie labeling van DNA en hybridisatie met een aangepaste tegels array. Voorafgaand stappen die kunnen worden gebruikt om de analyse te specifi optimaliserenc regulatoren worden beschreven. De door de matrix gegevensanalyse pieken worden gebruikt bindingsplaats motieven, die vervolgens experimenteel bevestigd voorspellen. Het motief voorspellingen kunnen verder worden gebruikt om gendoelstellingen van orthologe respons regulatoren bepalen nauw verwante soorten. We tonen de toepasbaarheid van deze methode door het bepalen van het gen doelen en bindingsplaats motieven en daarmee de functie voor een sigma54-afhankelijke respons regulator DVU3023 in de milieu-bacterie Desulfovibrio vulgaris Hildenborough voorspellen.
Introduction
Het vermogen van bacteriën om te overleven en gedijen kritisch afhankelijk van hoe goed zij kunnen waarnemen en reageren op verstoringen in hun omgeving, en dit op zijn beurt afhankelijk is van de signaaltransductie systemen. Het aantal signaleringssystemen een bacterie codeert is genoemd zijn "microbiële IQ" en kan een indicatie van zowel de variabiliteit van haar omgeving en de mogelijkheid om meerdere signalen te detecteren en fine-tunen zijn reactie 1 zijn. Twee componenten signaaltransductie-systemen (TCS) zijn de meest voorkomende signaleringssystemen die door bacteriën, en bestaan uit een histidine kinase (HK) dat de externe signaleert en stuurt via fosforylering een effector response regulator (RR) 2. RR een grote productie domeinen en dus verschillende effector functies, maar de meest voorkomende reactie is transcriptionele regulatie via een DNA-bindend domein 1. De signalen waargenomen en de bijbehorende functies van de vast merendeel van TCS onbekend blijven.
Hoewel in vivo werkwijzen zoals chip-chip routinematig worden gebruikt voor de bepaling van genomische bindingsplaatsen van transcriptiefactoren 3, kunnen ze alleen worden gebruikt voor bacteriële tweecomponenten systeem RR als de activerende voorwaarden of signalen bekend zijn. Vaak zijn de omgevingsfactoren die een TCS activeren zijn moeilijker te bepalen dan hun gendoelstellingen. De in vitro microarray gebaseerde test hier beschreven kan worden gebruikt om effectief en snel bepalen gendoelstellingen en functies van TCS voorspellen. Deze test maakt gebruik van het feit dat RR kunnen worden gefosforyleerd en dus geactiveerd in vitro gebruik van kleine moleculen donors zoals acetyl fosfaat 4.
In deze werkwijze, genaamd DAP-chip voor DNA-affiniteit-gezuiverde-chip (figuur 1), de RR gen van belang wordt gekloneerd met een His-tag in E. coli, en vervolgens gezuiverd gelabeld proteïne mag bind om genomische DNA geschoren. Het eiwit gebonden DNA wordt vervolgens verrijkt door affiniteit zuivering, zijn de verrijkte en input-DNA geamplificeerd, fluorescerend gemerkte, samengevoegd en gehybridiseerd aan een array die tegels op maat is gemaakt om het organisme plaats (figuur 1). Microarray experimenten kunnen artefacten en dus extra stappen worden gebruikt om de test te optimaliseren. Een dergelijke stap is om te proberen een doel bepalen RR onderzochte middels elektroforetische mobiliteit shift assays (EMSA) (zie workflow in figuur 2). Dan, na binding aan genomisch DNA en de DAP stappen het eiwitgebonden en input-DNA onderzocht door qPCR of de positieve doelstelling wordt verrijkt in de eiwitgebonden fractie ten opzichte van de inputfractie, hetgeen bevestigt optimale bindingsomstandigheden voor de RR (figuur 2). Na scala hybridisatie worden de gegevens geanalyseerd om pieken van hogere intensiteit signaal dat aangeeft genomische loci vinden waar de eiwit had gebonden. Functies kunnen worden voorspeld RR basis van de gendoelstellingen verkregen. De beoogde genomische loci worden gebruikt bindingsplaats motieven, die vervolgens experimenteel gevalideerd middels EMSA's (figuur 2) te voorspellen. De functionele voorspellingen en gendoelstellingen de RR kan worden uitgebreid tot nauw verwante soorten die orthologe RR coderen door het scannen van de genomen soortgelijke bindende motieven (figuur 2). De DAP-chip methode kan een schat aan informatie voor een TCS waar voorheen was er geen. De werkwijze kan ook worden gebruikt voor transcriptionele regulator indien het eiwit kan worden gezuiverd en DNA bindende voorwaarden kan worden bepaald, en voor elk organisme plaats met een genoomsequentie beschikbaar.
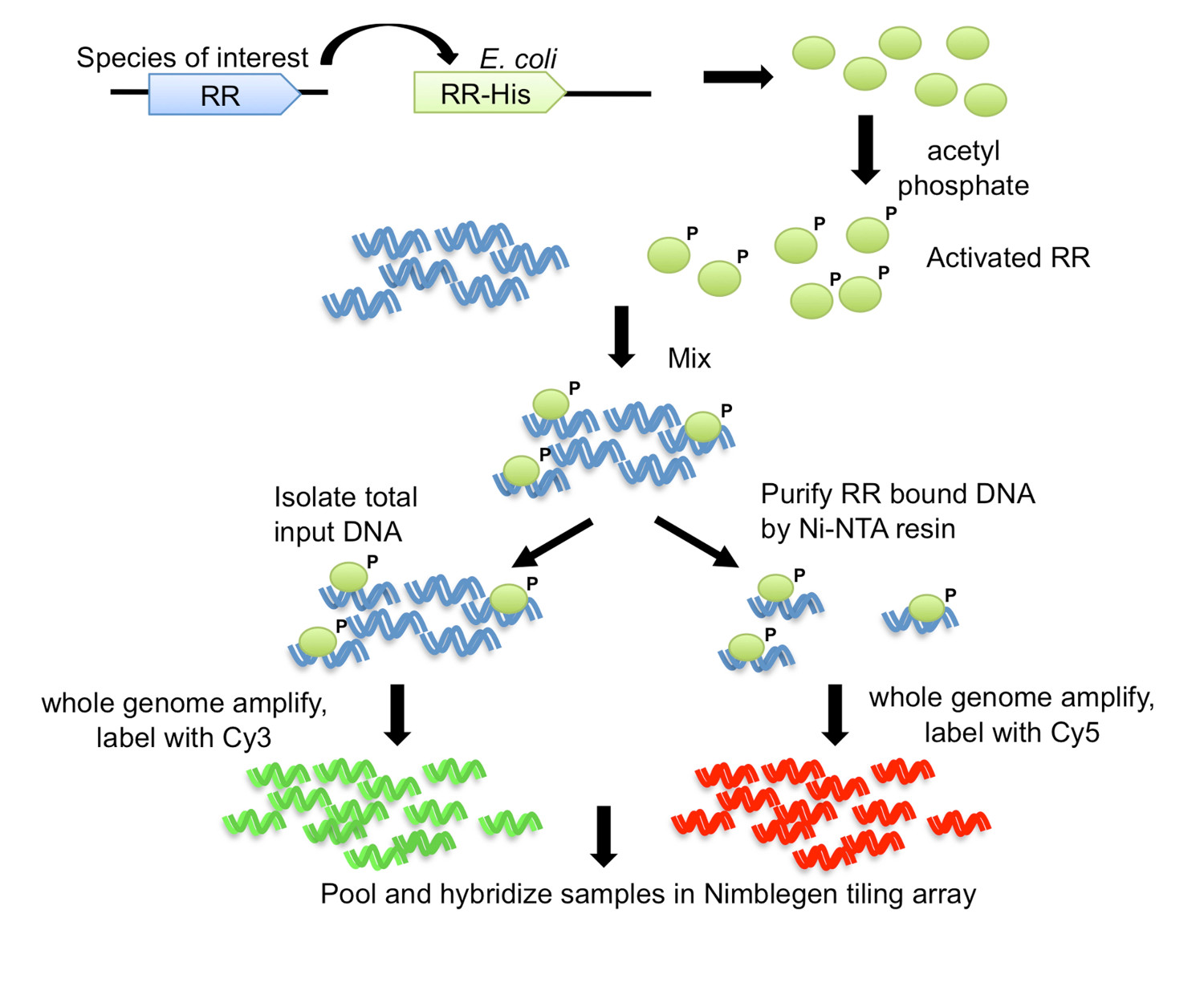
Figuur 1. Het DNA-affiniteit-gezuiverde chip (DAP-chip) strategie 7. De RR-gen van het organisme van belang wordt gekloneerd met een carboxy-eindstandige His-tag in een E. coli-expressie stam. Gezuiverd His-gemerkt eiwit wordt geactiveerd door fosforylering met acetyl fosfaat en gemengd met geschoren genomisch DNA. Een portie van de bindingsreactie wordt opgeslagen als input DNA, terwijl de rest wordt onderworpen aan affiniteitszuivering met Ni-NTA hars. De ingang en de RR-gebonden DNA zijn gehele genoom versterkt, en gelabeld met Cy3 en Cy5, respectievelijk. Het gemerkte DNA wordt samengevoegd en gehybridiseerd met een reeks tegels, dat vervolgens wordt geanalyseerd om de gendoelstellingen bepalen. Figuur aangepast en herdrukt met de creative commons licentie van 7.
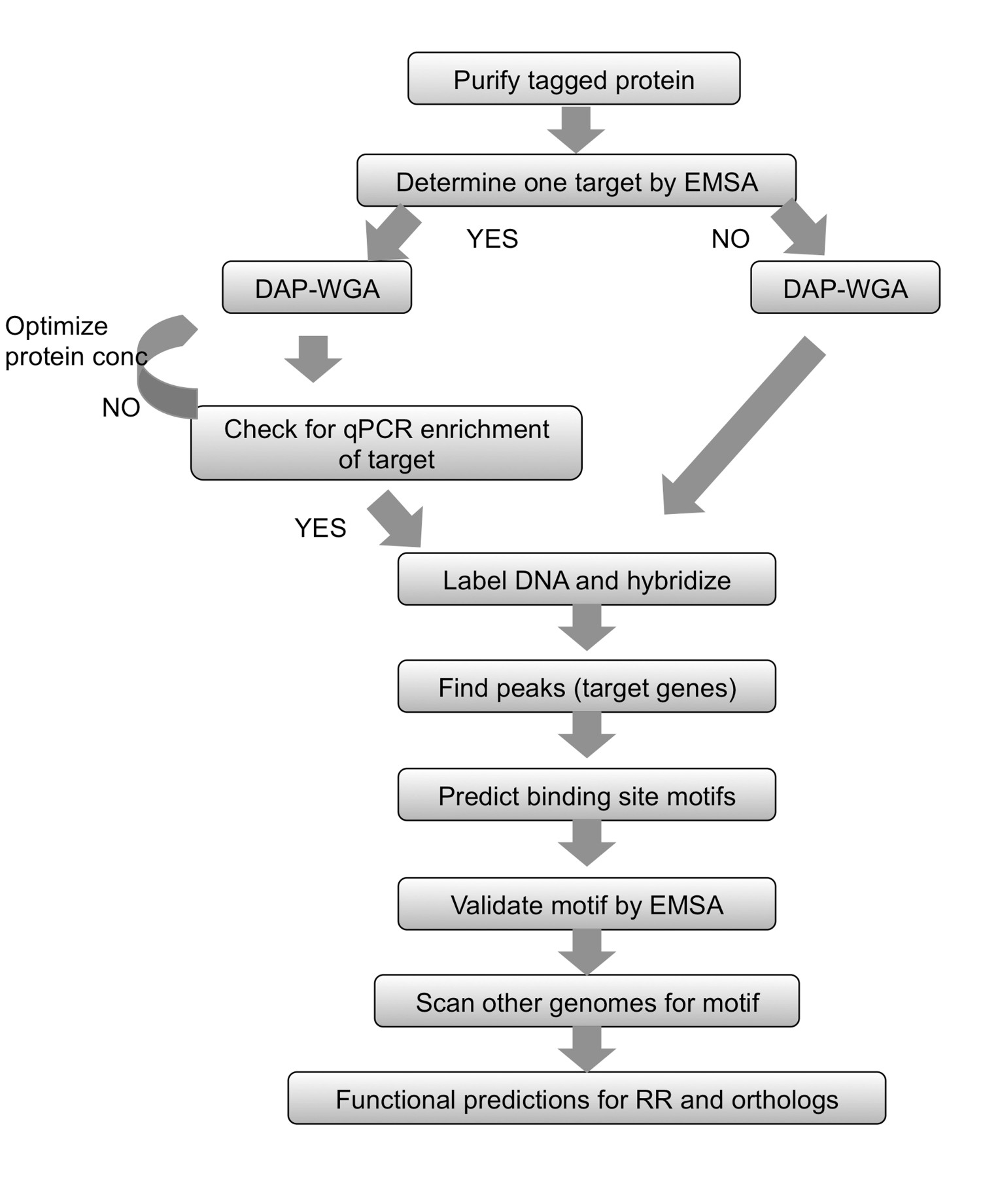
Figuur 2. Samenvatting van de workflow. Voor elk gezuiverd getagd protein, beginnen met het bepalen van een doel met behulp van EMSA. Laat eiwit aan genomisch DNA en DNA-affiniteit-zuiveren (DAP) en hele genoom binden versterken (WGA) de verrijkte en input DNA. Als een gen doelwit is bekend, gebruik qPCR zodat de target dat is verrijkt in de eiwitgebonden fractie. Als er geen doel kon worden bepaald, gaat u rechtstreeks naar DNA-etikettering en-array hybridisatie. Als verrijking door qPCR niet kon worden waargenomen, herhaal dan de eiwit-gDNA bindend en DAP-WGA stappen met verschillende hoeveelheden eiwit. Gebruik array analyse om pieken te vinden en in kaart hen om genen te richten. Gebruik de upstream-regio's van target genen bindingsplaats motieven voorspellen. Valideren van de motieven experimenteel gebruik van EMSA's. Gebruik het motief om de genomen van verwante soorten coderen orthologen van de RR in studie te scannen, en zo goed voorspellen genen gericht in die soorten. Op basis van de gendoelstellingen verkregen, kan de fysiologische functie RR en de orthologen worden voorspeld. Figuur aangepast en herdrukt met de creatieve commons licentie van 7.
Protocol
Representative Results
Discussion
De DAP-chip hier beschreven methode is succesvol gebruikt voor de gendoelstellingen bepalen meerdere bronrecords in Desulfovibrio vulgaris Hildenborough 7 waarvan een hier weergegeven als een representatief resultaat. Voor RR DVU3023, het kiezen van een kandidaat-gen doelwit was eenvoudig. DVU3025 is gelegen direct achter de RR-gen, en de RR en target genen worden bewaard in verschillende Desulfovibrio soorten, en bovendien DVU3025 heeft een voorspelde sigma54-afhankelijke promotor. De EMSA …
Disclosures
The authors have nothing to disclose.
Acknowledgements
Wij danken Amy Chen voor haar hulp bij de voorbereiding van de video-shoot en voor het aantonen van de techniek. Dit werk uitgevoerd door ENIGMA: Ecosystems and Networks Integrated met Genes and Molecular Assemblies (http://enigma.lbl.gov), een wetenschappelijk Focus Area Program bij het Lawrence Berkeley National Laboratory, werd gesteund door de Office of Science, Bureau van biologische en Environmental Research, van het Amerikaanse ministerie van Energie onder contract nummer DE-AC02-05CH11231.
Materials
| Name of Material/Equipment | Company | Catalog number | Comments |
| HisTrapFF column (Ni-Sepharose column) | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5255-01 | |
| Akta explorer (FPLC instrument) | GE Lfe Sciences, Pittsburgh, PA, USA | ||
| HiPrep 26/10 Desalting column | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5087-01 | |
| Qiaquick Gel extraction kit | Qiagen Inc, Valencia, CA, USA | 28704 | |
| Biotin-labeled oligonucleotides | Integrated DNA Technologies | N/A | |
| 6% polyacrylamide-0.5X TBE precast mini DNA retardation gel | Life Technologies, Grand Island, NY, USA | EC63652BOX | Alternately, you can pour your own gel. |
| Nylon membrane | EMD Millipore, Billerica, MA, USA | INYC00010 | |
| Trans-Blot SD Semi-dry electrophoretic transfer cell | Biorad, Hercules, CA, USA | 170-3940 | |
| Extra thick blot paper, 8 x 13.5 cm | Biorad, Hercules, CA, USA | 170-3967 | |
| UV crosslinker Model XL-1000 | Fisher Scientific | 11-992-89 | |
| Nucleic Acid chemiluminescent detection kit (Pierce) | Thermo fisher Scientific, Rockford, IL, USA | 89880 | |
| Ni-NTA agarose resin | Qiagen Inc, Valencia, CA, USA | 30210 | |
| GenomePlex Whole genome amplification kit (Fragmentation buffer, library preparation buffer, library stabilization solution, library preparation enzyme, 10X amplification master mix, WGA polymerase ) | Sigma-Aldrich, St. Louis, MO, USA | WGA2-50RXN | |
| Nanodrop ND-1000 | Thermo Scientific, Wilmington, DE, USA | For quantitation of DNA | |
| Perfecta Sybr Green SuperMix, with ROX | Quanta biosciences | 95055-500 | Any Sybr Green PCR mix may be used |
| PlateMax Ultra clear heat sealing film for qPCR | Axygen | ||
| 96 well clear low profile PCR microplate | Life Technologies, Grand Island, NY, USA | PCR-96-LP-AB-C | |
| Applied Biosystems StepOne Plus Real time PCR system | Life Technologies, Grand Island, NY, USA | 4376600 | Any real time PCR system may be used |
| Qiaquick PCR purification kit | Qiagen Inc, Valencia, CA, USA | 28104 | Any PCR clean up kit may be used |
| Cy3/Cy5-labeled nonamers | Trilink biotechnologies, San Diego, CA, USA | N46-0001, N46-0002 | |
| Klenow polymerase 50,000U/ml, 3'-5' exo- | New England Biolabs, Ipswich, MA | M0212M | |
| Hybridization system | Roche-Nimblegen, Madison, WI, USA | N/A | This company no longer makes arrays or related items, so alternate sources such as Agilent or Affymetrix will need to be used, |
| Custom printed microarrays and mixers | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Hybridization kit (2X Hybridization buffer, Hybridization component A, Alignment oligo) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Wash buffer kit (10X Wash buffer I, II, III, 1 M DTT) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| GenePix 4200A microarray scanner | Molecular Devices, Sunnyvale CA, USA | This model has been replaced by superior ones | |
| GenePix Pro microarray software | Molecular Devices, Sunnyvale CA, USA | ||
| Nimblescan v.2.4, ChIP-chip analysis software | Roche-Nimblegen, Madison, WI, USA | N/A | |
References
- Galperin, M. Y. Diversity of structure and function of response regulator output domains. Curr Opin Microbiol. 13, 150-159 (2010).
- Casino, P., Rubio, V., Marina, A. The mechanism of signal transduction by two-component systems. Curr Opin Struct Biol. 20, 763-771 (2010).
- Aparicio, O., Geisberg, J. V., Struhl, K. Chromatin immunoprecipitation for determining the association of proteins with specific genomic sequences in vivo. Curr Protoc Cell Biol. 17 (17), (2004).
- McCleary, W. R., Stock, J. B. Acetyl phosphate and the activation of two-component response regulators. J Biol Chem. 269, 31567-31572 (1994).
- Novichkov, P. S., et al. RegPrecise web services interface: programmatic access to the transcriptional regulatory interactions in bacteria reconstructed by comparative genomics. Nucleic Acids Res. 40, 604-608 (2012).
- Studholme, D. J., Buck, M., Nixon, T. Identification of potential sigma(N)-dependent promoters in bacterial genomes. Microbiology. 146 (12), 3021-3023 (2000).
- Rajeev, L., et al. Systematic mapping of two component response regulators to gene targets in a model sulfate reducing bacterium. Genome Biol. 12, 99 (2011).
- Crooks, G. E., Hon, G., Chandonia, J. M., Brenner, S. E. WebLogo: a sequence logo generator. Genome Res. 14, 1188-1190 (2004).
- Barbieri, C. M., Mack, T. R., Robinson, V. L., Miller, M. T., Stock, A. M. Regulation of response regulator autophosphorylation through interdomain contacts. J Biol Chem. 285, 32325-32335 (2010).
- Bourret, R. B. Receiver domain structure and function in response regulator proteins. Curr Opin Microbiol. 13, 142-149 (2010).
- Gao, R., Stock, A. M. Molecular strategies for phosphorylation-mediated regulation of response regulator activity. Curr Opin Microbiol. 13, 160-167 (2010).
- Hung, D. C., et al. Oligomerization of the response regulator ComE from Streptococcus mutans is affected by phosphorylation. J Bacteriol. 194, 1127-1135 (2012).
- Ladds, J. C., et al. The response regulator Spo0A from Bacillus subtilis is efficiently phosphorylated in Escherichia coli. FEMS Microbiol Lett. 223, 153-157 (2003).
- Wen, Y., Feng, J., Scott, D. R., Marcus, E. A., Sachs, G. Involvement of the HP0165-HP0166 two-component system in expression of some acidic-pH-upregulated genes of Helicobacter pylori. J Bacteriol. 188, 1750-1761 (2006).
- Liu, X., Noll, D. M., Lieb, J. D., Clarke, N. D. DIP-chip: rapid and accurate determination of DNA-binding specificity. Genome Res. 15, 421-427 (2005).
- Gossett, A. J., Lieb, J. D. DNA Immunoprecipitation (DIP) for the Determination of DNA-Binding Specificity. CSH Protoc. 2008, (2008).
- Mascher, T., et al. The Streptococcus pneumoniae cia regulon: CiaR target sites and transcription profile analysis. J Bacteriol. 185, 60-70 (2003).
- Park, P. J. ChIP-seq: advantages and challenges of a maturing technology. Nat Rev Genet. 10, 669-680 (2009).
- Lefrancois, P., Zheng, W., Snyder, M. ChIP-Seq using high-throughput DNA sequencing for genome-wide identification of transcription factor binding sites. Methods Enzymol. 470, 77-104 (2010).
- Ho, J. W., et al. ChIP-chip versus ChIP-seq: lessons for experimental design and data analysis. BMC Genomics. 12, 134 (2011).
- Liu, X., Lee, C. K., Granek, J. A., Clarke, N. D., Lieb, J. D. Whole-genome comparison of Leu3 binding in vitro and in vivo reveals the importance of nucleosome occupancy in target site selection. Genome Res. 16, 1517-1528 (2006).

