ADN purifié par affinité Chip (DAP-puce) Méthode pour déterminer les cibles génétiques pour bactérienne Deux systèmes de réglementation composants
Summary
Cet article vidéo décrit une méthode de puces à ADN sur la base in vitro afin de déterminer les gènes cibles et des sites de liaison pour deux régulateurs de réponse du système de composants.
Abstract
Les méthodes in vivo tels que ChIP-chip sont des techniques bien établies utilisées pour déterminer les cibles mondiales de gènes pour des facteurs de transcription. Cependant, ils sont d'une utilité limitée dans l'exploration de deux systèmes de réglementation composants bactériens avec des conditions d'activation non caractérisés. De tels systèmes régulent la transcription seulement quand elle est activée en présence de signaux uniques. Etant donné que ces signaux sont souvent inconnus, le procédé in vitro de puces à ADN située à décrit dans cet article vidéo peut être utilisée pour déterminer des cibles de gènes et des sites de liaison pour les régulateurs de réponse. Cette méthode puce ADN purifié par affinité peut être utilisée pour n'importe quel régulateur purifiée dans un organisme avec un génome séquencé. Le protocole consiste à permettre la protéine marquée purifiée à se lier à l'ADN génomique cisaillée et ensuite la purification par affinité de l'ADN lié à une protéine, suivie par le marquage fluorescent de l'ADN et l'hybridation à un réseau de carrelage personnalisé. Précédentes étapes qui peuvent être utilisés pour optimiser le dosage de spécificationc régulateurs sont également décrits. Les pics générés par l'analyse des données du tableau sont utilisés pour prédire motifs de sites de liaison, qui sont ensuite validés expérimentalement. Les prédictions de motifs peuvent être utilisés en outre pour déterminer les gènes cibles de régulateurs de réponse orthologues d'espèces étroitement apparentées. Nous démontrons l'applicabilité de ce procédé en déterminant les gènes cibles et de motifs de site de liaison et la prédiction ainsi la fonction d'un régulateur de réponse DVU3023 de sigma54-dépendante de l'environnement dans la bactérie Desulfovibrio vulgaris Hildenborough.
Introduction
La capacité des bactéries à survivre et à prospérer dépend de façon critique sur la façon dont ils sont capables de percevoir et de répondre aux perturbations de leur environnement, et ce, à son tour dépend de leurs systèmes de transduction du signal. Le nombre de systèmes de signalisation une bactérie de encode a été appelé son "QI microbienne" et peut être une indication de la variabilité de son environnement et de sa capacité à détecter des signaux multiples et d'affiner sa réponse 1. Deux systèmes de transduction de signaux composants (TCS) sont des systèmes de signalisation les plus répandues utilisées par les bactéries, et ils sont constitués d'une histidine kinase (HK) qui détecte le signal externe et le transmet par l'intermédiaire de la phosphorylation d'un régulateur de réponse effectrice (RR) 2. RR peuvent avoir une variété de domaines de la production et donc différents modes effectrices, mais la réponse la plus fréquente est la régulation transcriptionnelle via un domaine 1 liaison à l'ADN. Les signaux détectés et les fonctions correspondantes du canal déférentt majorité des TCS restent inconnus.
Bien que des procédés in vivo tels que ChIP-chip sont couramment utilisés pour la détermination des sites de liaison de facteurs de transcription génomiques 3, ils ne peuvent être utilisés pour les deux RR bactériennes du système composant si les conditions ou les signaux d'activation sont connus. Souvent, les signaux environnementaux qui activent un TCS sont plus difficiles à déterminer que leurs gènes cibles. La puce à ADN in vitro dosage à base décrit ici peut être utilisé pour déterminer rapidement et efficacement les gènes cibles et de prévoir des fonctions de TCS. Cet essai tire parti du fait que les RR peuvent être phosphorylés et donc activés in vitro à l'aide de petites molécules telles que des donneurs de phosphate d'acétyle 4.
Dans ce procédé, appelé DAP-puce pour l'ADN-affinité-puce purifié (Figure 1), le gène de la RR d'intérêt est clone avec un His-tag dans E. coli, et une protéine marquée par la suite purifié est autorisé à bie à la tonte de l'ADN génomique. L'ADN lié à une protéine est ensuite enrichi par affinité purification, l'ADN enrichi et entrée sont amplifiés, marqué par fluorescence, mis en commun et hybride à une gamme de carrelage qui est faite sur mesure pour l'organisme d'intérêt (figure 1). expériences de puces à ADN sont soumis à des artefacts et donc des mesures supplémentaires sont utilisés pour optimiser le dosage. Une telle démarche est de tenter de déterminer une cible pour le RR à l'étude en utilisant des tests électrophorétique de décalage de mobilité (EMSA) (voir flux de travail dans la figure 2). Puis, après liaison à l'ADN génomique et les étapes de la DAP, l'ADN lié à une protéine et d'entrée sont examinés par qPCR pour déterminer si la cible positif est enrichi dans la fraction liée aux protéines par rapport à la fraction d'entrée, confirmant ainsi les conditions optimales de liaison pour l' RR (figure 2). Après tableau hybridation, les données sont analysées pour trouver des pics de signal d'intensité plus élevée indiquant loci génomiques où la protéine hannonce lié. Les fonctions peuvent être prévus pour la RR en fonction des objectifs de gènes obtenus. Les loci génomiques cibles sont utilisés pour prédire motifs de sites de liaison, qui sont ensuite validés expérimentalement à l'aide EMSA (Figure 2). Les prédictions fonctionnelles et cibles génétiques pour la RR peuvent ensuite être étendues aux espèces qui codent RR orthologues en scannant les génomes pour des motifs de liaison similaires (figure 2) étroitement liés. La méthode DAP-puce peut fournir une mine d'informations pour un TCS où il n'y avait auparavant aucune. Le procédé peut également être utilisé pour n'importe quel régulateur transcriptionnel si la protéine peut être purifiée et des conditions de liaison d'ADN peut être déterminée, et pour n'importe quel organisme d'intérêt avec une séquence génomique disponible.
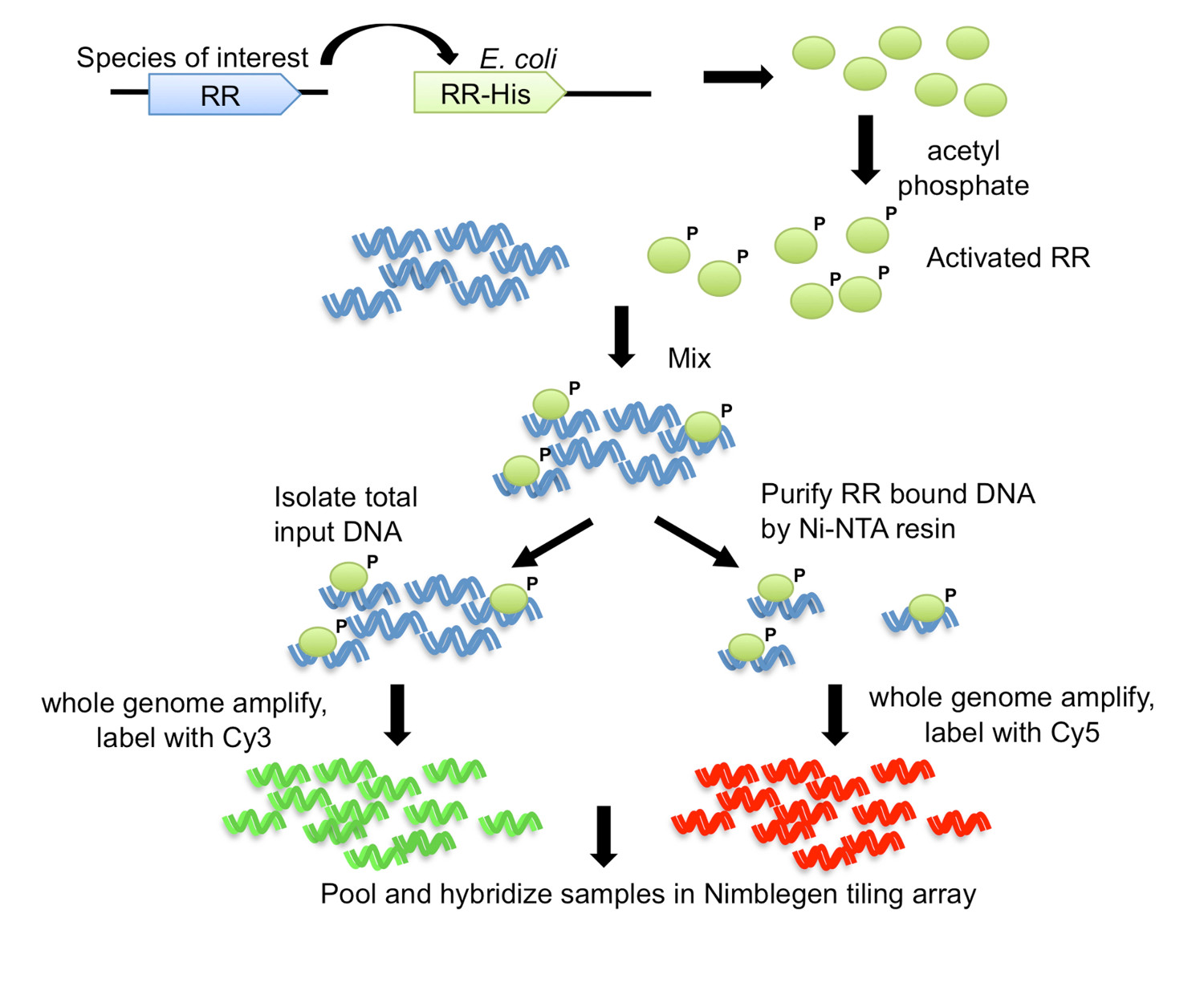
Figure 1. L'-puce ADN purifié par affinité (DAP-puce) stratégie 7. Le gène de la RR de l'organisme d'intérêt est cloné avec un His-tag carboxy-terminale dans un E. coli souche d'expression. Protéine purifiée marquée par His est activée par phosphorylation avec du phosphate d'acétyle, et on le mélange avec de l'ADN génomique cisaillé. Une aliquote de la réaction de liaison est enregistré sous forme d'ADN d'entrée, tandis que le reste est soumis à une purification par affinité en utilisant une résine Ni-NTA. L'entrée et l'ADN de RR lié sont génome amplifié, et marqué avec Cy3 et Cy5, respectivement. L'ADN marqué est mis en commun et hybridée à une matrice de carrelage, qui est ensuite analysé afin de déterminer les gènes cibles. Figure modifiée et réimprimée en utilisant la licence creative commons de 7.
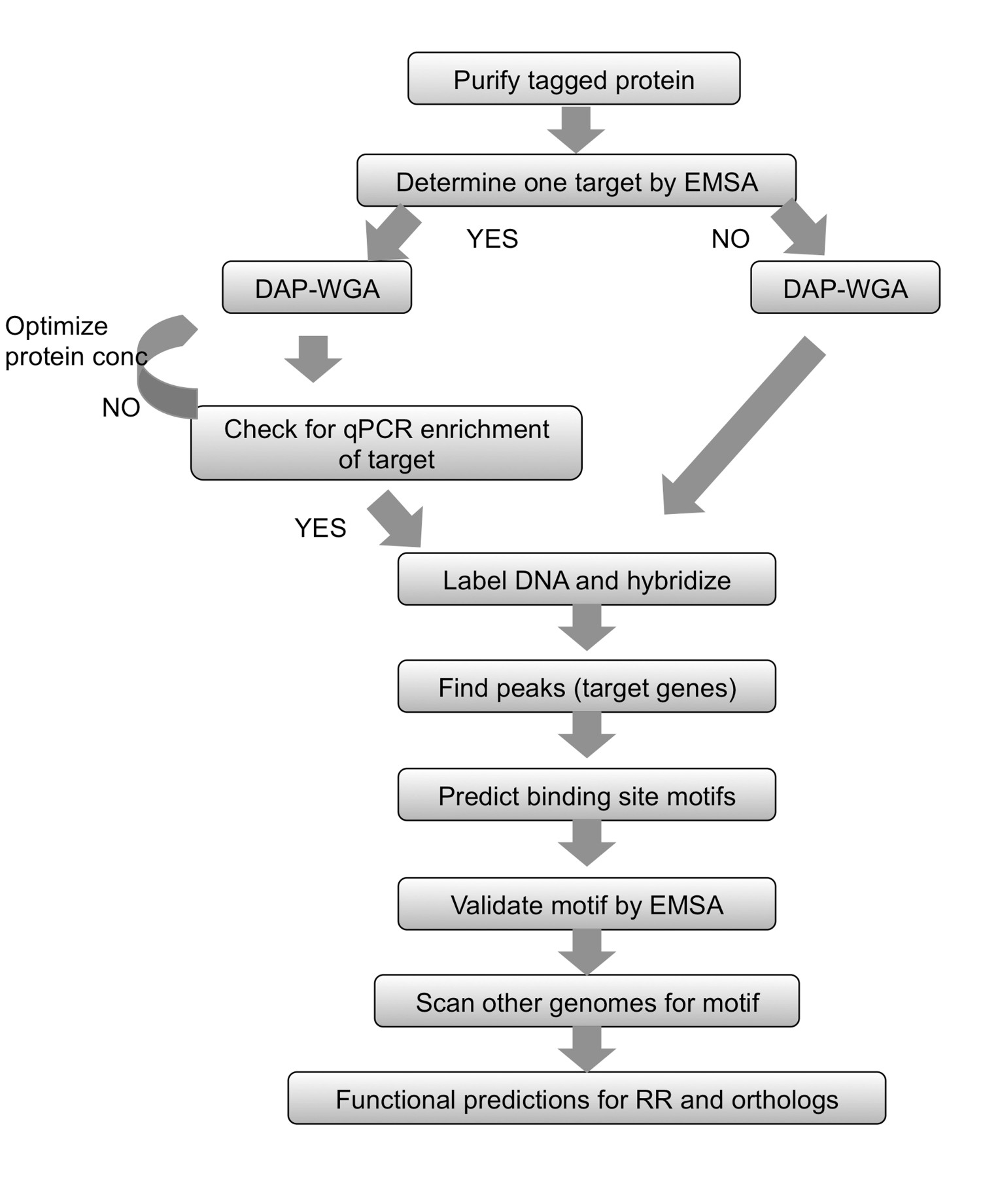
Figure 2. Sommaire des flux de travail. Pour toute protei marqué purifién, commencer par déterminer une cible en utilisant l'EMSA. Autoriser protéine à lier l'ADN génomique et l'ADN purifier par affinité (DAP) et l'ensemble du génome amplifier (WGA), le ADN enrichi et entrée. Si une cible du gène est connue, utiliser qPCR pour s'assurer que la cible connue est enrichi dans la fraction liée aux protéines. Si aucune cible pourrait être déterminée, passez directement à l'étiquetage de l'ADN et l'hybridation tableau. Si l'enrichissement par qPCR pas pu être observée, puis répétez la liaison protéine-ADNg et étapes DAP-WGA utilisant différentes quantités de protéines. Utiliser l'analyse de réseau pour trouver les pics et les faire correspondre à cibler des gènes. Utiliser les régions en amont de gènes cibles pour prédire motifs de site de liaison. Valider les motifs expérimentalement à l'aide EMSA. Utilisez le motif de scanner les génomes d'espèces apparentées codant orthologues de la RR à l'étude, et de prévoir les gènes ciblés dans ces espèces. Sur la base des cibles de gènes obtenus, la fonction physiologique de la RR et ses orthologues peut être prédite. Figure modifié et reproduit à l'aide du c créativeommons licence de 7.
Protocol
Representative Results
Discussion
La méthode DAP-puce décrit ici a été utilisé avec succès pour déterminer les cibles génétiques pour plusieurs RI dans Desulfovibrio vulgaris Hildenborough 7 dont un est représenté ici comme un résultat représentatif. Pour RR DVU3023, le choix d'une cible de gène candidat était simple. DVU3025 est située immédiatement en aval du gène de la RR, et les gènes de la RR et cible sont conservées dans plusieurs espèces Desulfovibrio, et en outre DVU3025 a un promoteur de sig…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Nous remercions Amy Chen pour son aide dans la préparation pour le tournage de la vidéo et pour démontrer la technique. Ce travail mené par ENIGMA: écosystèmes intégrés et des réseaux avec des gènes et des assemblages moléculaires (http://enigma.lbl.gov), un programme scientifique Secteur d'intérêt au Lawrence Berkeley National Laboratory, a été pris en charge par le Bureau de la science, Bureau du biologique et recherche sur l'environnement, du Département américain de l'Énergie sous contrat DE-AC02-05CH11231.
Materials
| Name of Material/Equipment | Company | Catalog number | Comments |
| HisTrapFF column (Ni-Sepharose column) | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5255-01 | |
| Akta explorer (FPLC instrument) | GE Lfe Sciences, Pittsburgh, PA, USA | ||
| HiPrep 26/10 Desalting column | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5087-01 | |
| Qiaquick Gel extraction kit | Qiagen Inc, Valencia, CA, USA | 28704 | |
| Biotin-labeled oligonucleotides | Integrated DNA Technologies | N/A | |
| 6% polyacrylamide-0.5X TBE precast mini DNA retardation gel | Life Technologies, Grand Island, NY, USA | EC63652BOX | Alternately, you can pour your own gel. |
| Nylon membrane | EMD Millipore, Billerica, MA, USA | INYC00010 | |
| Trans-Blot SD Semi-dry electrophoretic transfer cell | Biorad, Hercules, CA, USA | 170-3940 | |
| Extra thick blot paper, 8 x 13.5 cm | Biorad, Hercules, CA, USA | 170-3967 | |
| UV crosslinker Model XL-1000 | Fisher Scientific | 11-992-89 | |
| Nucleic Acid chemiluminescent detection kit (Pierce) | Thermo fisher Scientific, Rockford, IL, USA | 89880 | |
| Ni-NTA agarose resin | Qiagen Inc, Valencia, CA, USA | 30210 | |
| GenomePlex Whole genome amplification kit (Fragmentation buffer, library preparation buffer, library stabilization solution, library preparation enzyme, 10X amplification master mix, WGA polymerase ) | Sigma-Aldrich, St. Louis, MO, USA | WGA2-50RXN | |
| Nanodrop ND-1000 | Thermo Scientific, Wilmington, DE, USA | For quantitation of DNA | |
| Perfecta Sybr Green SuperMix, with ROX | Quanta biosciences | 95055-500 | Any Sybr Green PCR mix may be used |
| PlateMax Ultra clear heat sealing film for qPCR | Axygen | ||
| 96 well clear low profile PCR microplate | Life Technologies, Grand Island, NY, USA | PCR-96-LP-AB-C | |
| Applied Biosystems StepOne Plus Real time PCR system | Life Technologies, Grand Island, NY, USA | 4376600 | Any real time PCR system may be used |
| Qiaquick PCR purification kit | Qiagen Inc, Valencia, CA, USA | 28104 | Any PCR clean up kit may be used |
| Cy3/Cy5-labeled nonamers | Trilink biotechnologies, San Diego, CA, USA | N46-0001, N46-0002 | |
| Klenow polymerase 50,000U/ml, 3'-5' exo- | New England Biolabs, Ipswich, MA | M0212M | |
| Hybridization system | Roche-Nimblegen, Madison, WI, USA | N/A | This company no longer makes arrays or related items, so alternate sources such as Agilent or Affymetrix will need to be used, |
| Custom printed microarrays and mixers | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Hybridization kit (2X Hybridization buffer, Hybridization component A, Alignment oligo) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Wash buffer kit (10X Wash buffer I, II, III, 1 M DTT) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| GenePix 4200A microarray scanner | Molecular Devices, Sunnyvale CA, USA | This model has been replaced by superior ones | |
| GenePix Pro microarray software | Molecular Devices, Sunnyvale CA, USA | ||
| Nimblescan v.2.4, ChIP-chip analysis software | Roche-Nimblegen, Madison, WI, USA | N/A | |
References
- Galperin, M. Y. Diversity of structure and function of response regulator output domains. Curr Opin Microbiol. 13, 150-159 (2010).
- Casino, P., Rubio, V., Marina, A. The mechanism of signal transduction by two-component systems. Curr Opin Struct Biol. 20, 763-771 (2010).
- Aparicio, O., Geisberg, J. V., Struhl, K. Chromatin immunoprecipitation for determining the association of proteins with specific genomic sequences in vivo. Curr Protoc Cell Biol. 17 (17), (2004).
- McCleary, W. R., Stock, J. B. Acetyl phosphate and the activation of two-component response regulators. J Biol Chem. 269, 31567-31572 (1994).
- Novichkov, P. S., et al. RegPrecise web services interface: programmatic access to the transcriptional regulatory interactions in bacteria reconstructed by comparative genomics. Nucleic Acids Res. 40, 604-608 (2012).
- Studholme, D. J., Buck, M., Nixon, T. Identification of potential sigma(N)-dependent promoters in bacterial genomes. Microbiology. 146 (12), 3021-3023 (2000).
- Rajeev, L., et al. Systematic mapping of two component response regulators to gene targets in a model sulfate reducing bacterium. Genome Biol. 12, 99 (2011).
- Crooks, G. E., Hon, G., Chandonia, J. M., Brenner, S. E. WebLogo: a sequence logo generator. Genome Res. 14, 1188-1190 (2004).
- Barbieri, C. M., Mack, T. R., Robinson, V. L., Miller, M. T., Stock, A. M. Regulation of response regulator autophosphorylation through interdomain contacts. J Biol Chem. 285, 32325-32335 (2010).
- Bourret, R. B. Receiver domain structure and function in response regulator proteins. Curr Opin Microbiol. 13, 142-149 (2010).
- Gao, R., Stock, A. M. Molecular strategies for phosphorylation-mediated regulation of response regulator activity. Curr Opin Microbiol. 13, 160-167 (2010).
- Hung, D. C., et al. Oligomerization of the response regulator ComE from Streptococcus mutans is affected by phosphorylation. J Bacteriol. 194, 1127-1135 (2012).
- Ladds, J. C., et al. The response regulator Spo0A from Bacillus subtilis is efficiently phosphorylated in Escherichia coli. FEMS Microbiol Lett. 223, 153-157 (2003).
- Wen, Y., Feng, J., Scott, D. R., Marcus, E. A., Sachs, G. Involvement of the HP0165-HP0166 two-component system in expression of some acidic-pH-upregulated genes of Helicobacter pylori. J Bacteriol. 188, 1750-1761 (2006).
- Liu, X., Noll, D. M., Lieb, J. D., Clarke, N. D. DIP-chip: rapid and accurate determination of DNA-binding specificity. Genome Res. 15, 421-427 (2005).
- Gossett, A. J., Lieb, J. D. DNA Immunoprecipitation (DIP) for the Determination of DNA-Binding Specificity. CSH Protoc. 2008, (2008).
- Mascher, T., et al. The Streptococcus pneumoniae cia regulon: CiaR target sites and transcription profile analysis. J Bacteriol. 185, 60-70 (2003).
- Park, P. J. ChIP-seq: advantages and challenges of a maturing technology. Nat Rev Genet. 10, 669-680 (2009).
- Lefrancois, P., Zheng, W., Snyder, M. ChIP-Seq using high-throughput DNA sequencing for genome-wide identification of transcription factor binding sites. Methods Enzymol. 470, 77-104 (2010).
- Ho, J. W., et al. ChIP-chip versus ChIP-seq: lessons for experimental design and data analysis. BMC Genomics. 12, 134 (2011).
- Liu, X., Lee, C. K., Granek, J. A., Clarke, N. D., Lieb, J. D. Whole-genome comparison of Leu3 binding in vitro and in vivo reveals the importance of nucleosome occupancy in target site selection. Genome Res. 16, 1517-1528 (2006).

