שיטת צ'יפ (DAP-שבב) מטוהרת-DNA-זיקה לקביעת מטרות גן לחיידקית שתי מערכות רגולטוריות רכיב
Summary
מאמר וידאו זה מתאר שיטת microarray מבוסס במבחנה כדי לקבוע את מטרות הגן ואתרי קישור לשני רגולטורים תגובת מערכת רכיב.
Abstract
בשיטות vivo כגון שבב שבב טכניקות מבוססות היטב המשמשות לקביעת מטרות גן הגלובליות לגורמי שעתוק. עם זאת, הם של שימוש מוגבל בחקר שתי מערכות רגולציה מרכיב חיידקים עם תנאי הפעלת uncharacterized. מערכות כאלה להסדיר שעתוק רק כאשר מופעלים בנוכחות אותות ייחודיים. מאז האותות הללו הם לעתים קרובות לא ידועים, חוץ גופית בשיטה microarray מבוססת שבתוארה במאמר זה וידאו יכול לשמש כדי לקבוע מטרות גן ואתרי קישור לרגולטורי תגובה. שיטה מטוהרת-DNA-זיקת שבב זה עשויה לשמש לכל רגולטור מטוהר בכל אורגניזם עם הגנום רצף. הפרוטוקול כרוך מאפשר מטוהר החלבון מתויג להיקשר הדנ"א הגנומי טעון ולאחר מכן טיהור זיקת DNA הנכנס החלבון, ואחריו ניאון תיוג של ה-DNA וכלאה למערך ריצוף מותאם אישית. שקדמו לצעדים שעשויים לשמש כדי לייעל את assay לספציפיהרגולטורים ג גם מתוארים. הפסגות שנוצרו על ידי ניתוח נתוני מערך משמשות כדי לחזות מוטיבים אתר קישור, אשר לאחר מכן הם תוקף באופן ניסיוני. תחזיות המוטיב יכולות לשמש נוספת כדי לקבוע מטרות גן של רגולטורים תגובת orthologous במינים קרובים. אנו מדגימים את תחולתה של שיטה זו על ידי קביעת מטרות הגן ומחייב מוטיבים אתר ובכך לחזות את הפונקציה עבור DVU3023 רגולטור תגובת sigma54 תלוי בחיידק הסביבתי Desulfovibrio vulgaris Hildenborough.
Introduction
היכולת של חיידקים לשרוד ולשגשג תלויה באופן קריטי על כמה טוב הם מסוגלים לקלוט ולהגיב להפרעות בסביבתם, וזה בתורו הוא תלוי במערכות העברת אותותיהם. מספר מערכות אזעקת encodes חיידק כבר נקרא "IQ של חיידקים" שלה ויכול להיות אינדיקציה של שתי השתנות של סביבתו ואת יכולתו לחוש אותות מרובים ולכוונן את תגובתה 1. שתי מערכות רכיב אות התמרה (TCS) הן מערכות האיתות הנפוצות ביותר בשימוש על ידי חיידקים, והם מורכבים מקינאז היסטידין (HK) שחש את האות החיצונית ומעביר באמצעות זירחון לרגולטור תגובת מפעיל (RR) 2. RRs יכול להיות מגוון של תחומים פלט ומצבים שונים ובכך מפעיל, אבל התגובה הנפוצה ביותר היא תקנת תעתיק באמצעות DNA מחייב תחום 1. האותות חשו ופונקציות המקבילה vasלא רוב TCSs אינו ידוע.
למרות in vivo שיטות כגון שבב שבב משמשות באופן שיגרתי לקביעת אתרי קישור הגנומי של שעתוק גורמי 3, הם יכולים לשמש רק לRRs שתי מערכת מרכיב חיידקים אם התנאים להפעלה או אותות ידועים. לעתים קרובות הרמזים הסביבתיים המפעילים TCS הם יותר קשים לקבוע ממטרות הגן שלהם. במבחנה microarray assay המבוסס המתואר כאן ניתן להשתמש ביעילות ובמהירות כדי לקבוע את מטרות הגן ולחזות פונקציות של TCSs. assay זה מנצל את העובדה שניתן phosphorylated RRs וכך הופעל במבחנה באמצעות תורמי מולקולה קטנים כמו פוספט אצטיל 4.
בשיטה זו, בשם DAP-שבבים עבור ה-DNA זיקה מטוהר שבב (איור 1), גן RR של עניין הוא משובט עם התג שלו בE. coli, וטהר לאחר מכן חלבון מתויג מותר דוnd לטעון הדנ"א הגנומי. ה-DNA חייב החלבון מועשר לאחר מכן על ידי זיקה לטיהור, ה-DNA המועשר וקלט הם מוגברות, שכותרתו fluorescently, אספו יחד והכלאה למגוון ריצוף כי הוא עשה מותאם אישית לאורגניזם של עניין (איור 1). ניסויי microarray כפופים לחפצים, ולכן צעדים נוספים מועסקים כדי לייעל את assay. צעד אחד כזה הוא לנסות ולקבוע יעד אחד לRR תחת מחקר באמצעות מבחני משמרת ניידות electrophoretic (EMSA) (ראה זרימת עבודה באיור 2). לאחר מכן, בעקבות מחייב הדנ"א הגנומי וצעדי DAP, ה-DNA חייב החלבון והקלט נבדקים על ידי qPCR כדי לראות אם היעד החיובי הוא מועשר בשבריר מאוגד החלבון ביחס לשבריר הקלט, ובכך אישרה תנאים אופטימליים מחייב עבור RR (איור 2). לאחר הכלאת מערך, הנתונים מנותחים למצוא פסגות של אות בעוצמה גבוהה המעידות על לוקוסים הגנומי שבו שעות החלבוןמודעה קשורה. פונקציות יכולות להיות חזויה עבור RR מבוסס על מטרות הגן שהושגו. הלוקוסים הגנומי היעד משמשים כדי לחזות מוטיבים אתר מחייב, אשר לאחר מכן בניסוי תוקף באמצעות EMSAs (איור 2). התחזיות פונקציונליות ומטרות גן לRR יכולים אז להיות מורחבות למינים קשורות באופן הדוק המקודדים RRs orthologous ידי סריקת הגנום אלה למוטיבים המחייבים דומים (איור 2). שיטת DAP-השבב יכולה לספק שפע של מידע לTCS בהם בעבר לא היה כלום. גם השיטה יכולה לשמש לכל רגולטור תעתיק אם החלבון יכול להיות מטוהרת וניתן לקבוע תנאים המחייבים DNA, ולכל אורגניזם של עניין עם רצף הגנום זמין.
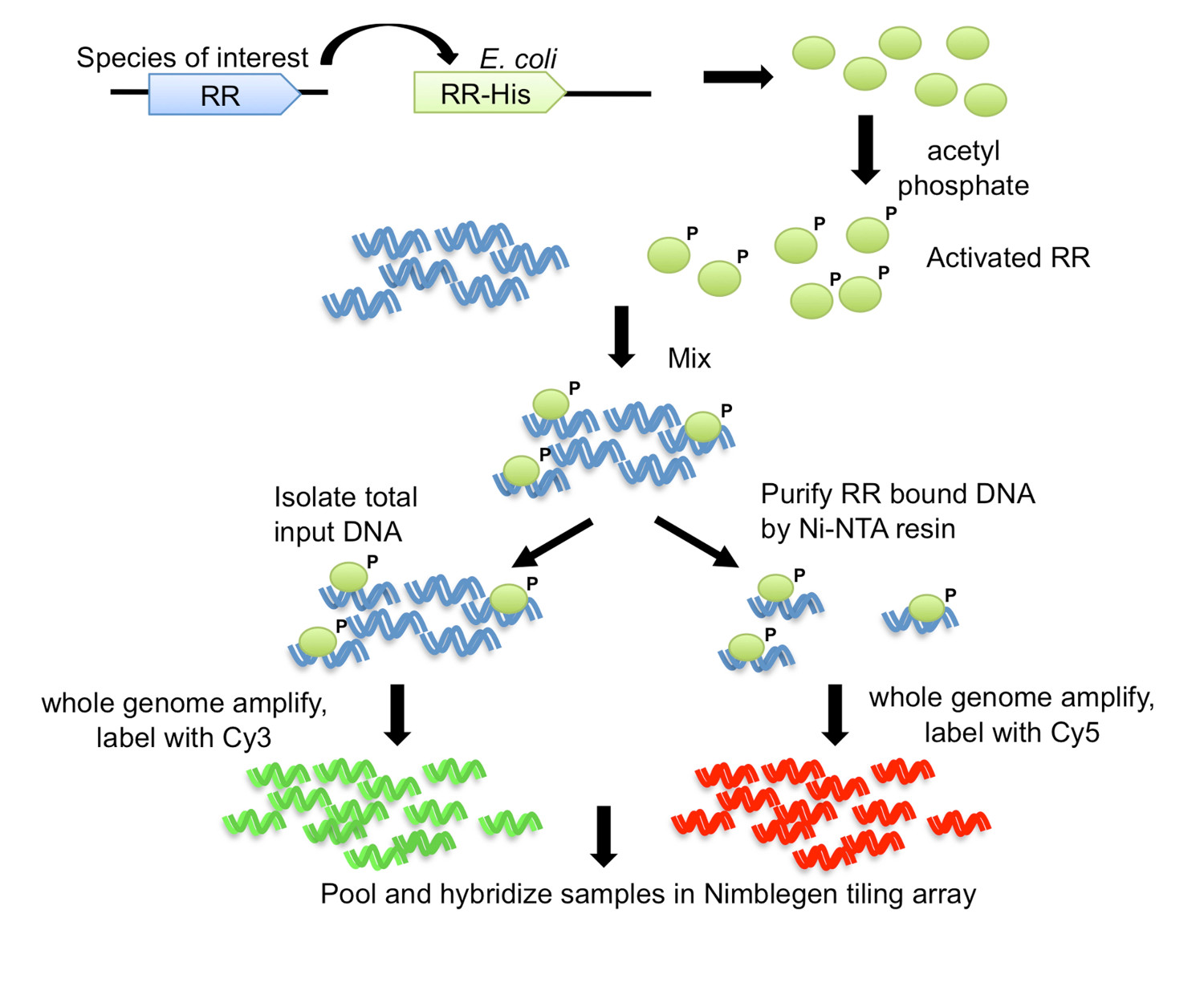
איור 1. מטוהר-DNA-זיקת השבב (DAP-אסטרטגית שבב) 7. גן RR מהאורגניזם של עניין הוא משובט עם התג שלו carboxy-מסוף לE. זן ביטוי coli. חלבון שלו מתויג מטוהר מופעל על ידי זירחון עם פוספט אצטיל, ומעורבב עם הדנ"א הגנומי טעון. Aliquot של התגובה מחייבת נשמר כDNA קלט, ואילו השאר הוא נתון לטיהור זיקה באמצעות שרף Ni-נ.ת. ע. הקלט ואת ה-DNA הנכנס RR הוא הגנום כולו מוגבר, ושכותרתו עם Cy3 וCy5, בהתאמה. ה-DNA שכותרתו היא אספו יחד והכלאה למגוון ריצוף, אשר לאחר מכן נותח על מנת לקבוע מטרות הגן. איור שונה והודפס מחדש באמצעות הרישיון של Creative Commons מ7.
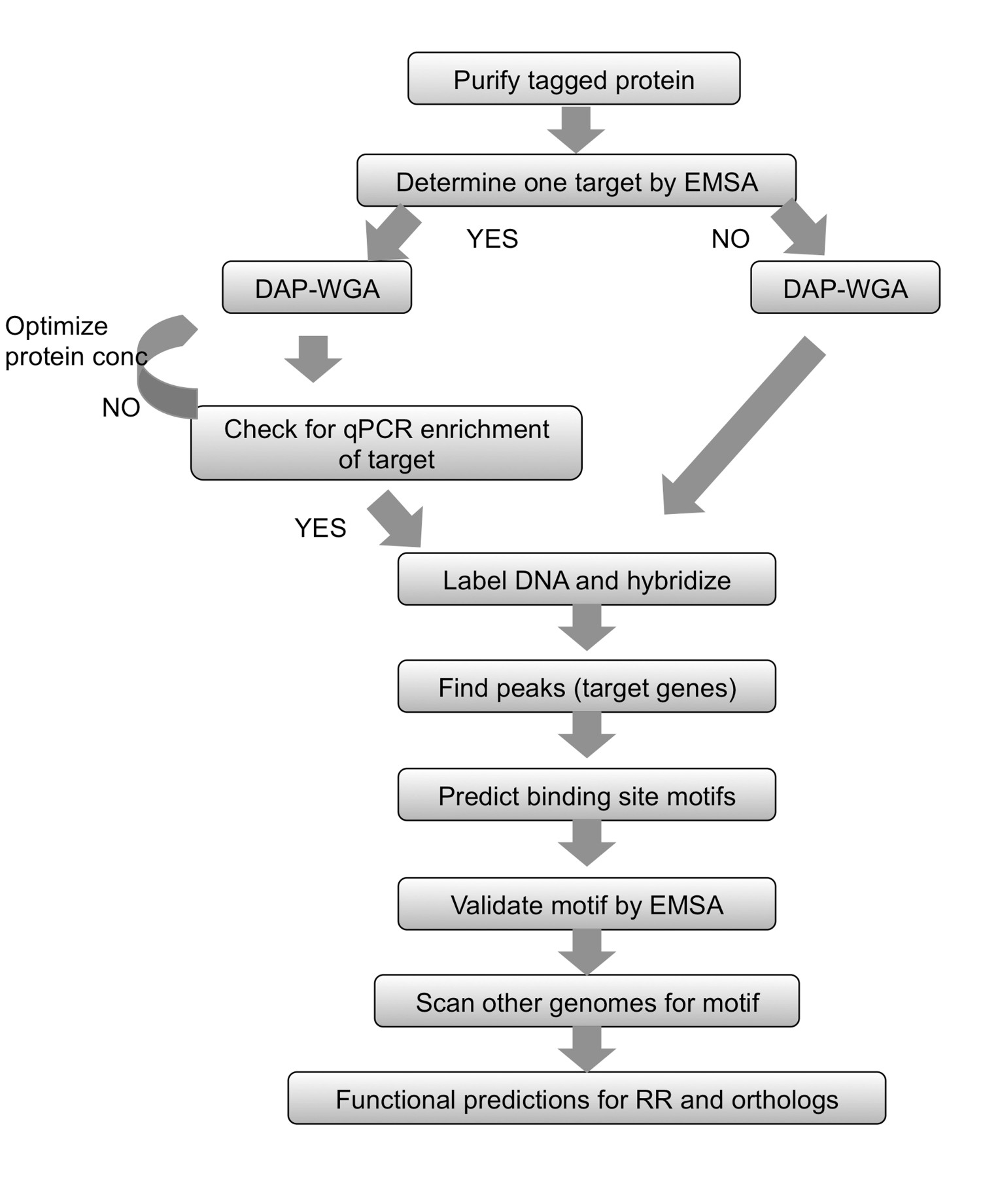
איור 2. סיכום של זרימת עבודה. לכל מטוהר protei מתויגn, להתחיל על ידי קביעת יעד באמצעות EMSA. אפשר לחלבון לקשור הדנ"א הגנומי ולאחר מכן ה-DNA זיקה לטהר (DAP) והגנום כולו להגביר את ה-DNA והקלט המועשר (WGA). אם יעד גן ידוע, השתמש qPCR כדי להבטיח כי היעד הידוע הוא מועשר בשבריר מאוגד החלבון. אם לא ניתן לקבוע יעד, להמשיך ישירות לתיוג ה-DNA וכלת מערך. אם העשרה על ידי qPCR לא יכולה להיות שנצפתה, ולאחר מכן לחזור על החלבון מחייב-gDNA וצעדי DAP-WGA המשתמשים בכמויות חלבון שונות. השתמש בניתוח מערך למצוא פסגות ולמפות אותם כדי למקד את הגנים. השתמש באזורים במעלה הזרם של גנים המטרה לחזות מוטיבים אתר קישור. לאמת את המוטיבים בניסוי באמצעות EMSAs. השתמש במוטיב לסרוק את הגנום של מינים קרובים קידוד orthologs של RR תחת מחקר, ולחזות גנים ממוקדים במינים אלה גם כן. בהתבסס על מטרות הגן השיגו, התפקוד הפיזיולוגי של RR וorthologs ניתן לחזות. איור שונה והודפס מחדש באמצעות ג יצירתייםommons רישיון מ7.
Protocol
Representative Results
Discussion
שיטת DAP-השבב שתוארה כאן הייתה הצלחה בשימוש כדי לקבוע את מטרות הגן במשך כמה RRs בDesulfovibrio vulgaris 7 Hildenborough של איזה מהם מוצג כאן כתוצאה נציג. לDVU3023 RR, בחירת יעד גן מועמד הייתה פשוטה. DVU3025 ממוקם מייד במורד הזרם של גן RR, וגני RR והיעד הם נשמרים בכמה מיני Desulfovibrio, ובנוס…
Disclosures
The authors have nothing to disclose.
Acknowledgements
אנו מודים איימי חן על עזרתה בהכנה לצילומי הווידאו ולהפגין את הטכניקה. עבודה זו שנערכה על ידי ENIGMA: מערכות אקולוגיות ורשתות משולבות עם גנים והרכבות מולקולריות (http://enigma.lbl.gov), תכנית אזור המוקד מדעי במעבדה הלאומית לורנס ברקלי, נתמכת על ידי משרד מדע, המשרד הביולוגי ו מחקר סביבתי, של משרד אנרגיה של ארה"ב תחת חוזה מס 'DE-AC02-05CH11231.
Materials
| Name of Material/Equipment | Company | Catalog number | Comments |
| HisTrapFF column (Ni-Sepharose column) | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5255-01 | |
| Akta explorer (FPLC instrument) | GE Lfe Sciences, Pittsburgh, PA, USA | ||
| HiPrep 26/10 Desalting column | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5087-01 | |
| Qiaquick Gel extraction kit | Qiagen Inc, Valencia, CA, USA | 28704 | |
| Biotin-labeled oligonucleotides | Integrated DNA Technologies | N/A | |
| 6% polyacrylamide-0.5X TBE precast mini DNA retardation gel | Life Technologies, Grand Island, NY, USA | EC63652BOX | Alternately, you can pour your own gel. |
| Nylon membrane | EMD Millipore, Billerica, MA, USA | INYC00010 | |
| Trans-Blot SD Semi-dry electrophoretic transfer cell | Biorad, Hercules, CA, USA | 170-3940 | |
| Extra thick blot paper, 8 x 13.5 cm | Biorad, Hercules, CA, USA | 170-3967 | |
| UV crosslinker Model XL-1000 | Fisher Scientific | 11-992-89 | |
| Nucleic Acid chemiluminescent detection kit (Pierce) | Thermo fisher Scientific, Rockford, IL, USA | 89880 | |
| Ni-NTA agarose resin | Qiagen Inc, Valencia, CA, USA | 30210 | |
| GenomePlex Whole genome amplification kit (Fragmentation buffer, library preparation buffer, library stabilization solution, library preparation enzyme, 10X amplification master mix, WGA polymerase ) | Sigma-Aldrich, St. Louis, MO, USA | WGA2-50RXN | |
| Nanodrop ND-1000 | Thermo Scientific, Wilmington, DE, USA | For quantitation of DNA | |
| Perfecta Sybr Green SuperMix, with ROX | Quanta biosciences | 95055-500 | Any Sybr Green PCR mix may be used |
| PlateMax Ultra clear heat sealing film for qPCR | Axygen | ||
| 96 well clear low profile PCR microplate | Life Technologies, Grand Island, NY, USA | PCR-96-LP-AB-C | |
| Applied Biosystems StepOne Plus Real time PCR system | Life Technologies, Grand Island, NY, USA | 4376600 | Any real time PCR system may be used |
| Qiaquick PCR purification kit | Qiagen Inc, Valencia, CA, USA | 28104 | Any PCR clean up kit may be used |
| Cy3/Cy5-labeled nonamers | Trilink biotechnologies, San Diego, CA, USA | N46-0001, N46-0002 | |
| Klenow polymerase 50,000U/ml, 3'-5' exo- | New England Biolabs, Ipswich, MA | M0212M | |
| Hybridization system | Roche-Nimblegen, Madison, WI, USA | N/A | This company no longer makes arrays or related items, so alternate sources such as Agilent or Affymetrix will need to be used, |
| Custom printed microarrays and mixers | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Hybridization kit (2X Hybridization buffer, Hybridization component A, Alignment oligo) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Wash buffer kit (10X Wash buffer I, II, III, 1 M DTT) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| GenePix 4200A microarray scanner | Molecular Devices, Sunnyvale CA, USA | This model has been replaced by superior ones | |
| GenePix Pro microarray software | Molecular Devices, Sunnyvale CA, USA | ||
| Nimblescan v.2.4, ChIP-chip analysis software | Roche-Nimblegen, Madison, WI, USA | N/A | |
References
- Galperin, M. Y. Diversity of structure and function of response regulator output domains. Curr Opin Microbiol. 13, 150-159 (2010).
- Casino, P., Rubio, V., Marina, A. The mechanism of signal transduction by two-component systems. Curr Opin Struct Biol. 20, 763-771 (2010).
- Aparicio, O., Geisberg, J. V., Struhl, K. Chromatin immunoprecipitation for determining the association of proteins with specific genomic sequences in vivo. Curr Protoc Cell Biol. 17 (17), (2004).
- McCleary, W. R., Stock, J. B. Acetyl phosphate and the activation of two-component response regulators. J Biol Chem. 269, 31567-31572 (1994).
- Novichkov, P. S., et al. RegPrecise web services interface: programmatic access to the transcriptional regulatory interactions in bacteria reconstructed by comparative genomics. Nucleic Acids Res. 40, 604-608 (2012).
- Studholme, D. J., Buck, M., Nixon, T. Identification of potential sigma(N)-dependent promoters in bacterial genomes. Microbiology. 146 (12), 3021-3023 (2000).
- Rajeev, L., et al. Systematic mapping of two component response regulators to gene targets in a model sulfate reducing bacterium. Genome Biol. 12, 99 (2011).
- Crooks, G. E., Hon, G., Chandonia, J. M., Brenner, S. E. WebLogo: a sequence logo generator. Genome Res. 14, 1188-1190 (2004).
- Barbieri, C. M., Mack, T. R., Robinson, V. L., Miller, M. T., Stock, A. M. Regulation of response regulator autophosphorylation through interdomain contacts. J Biol Chem. 285, 32325-32335 (2010).
- Bourret, R. B. Receiver domain structure and function in response regulator proteins. Curr Opin Microbiol. 13, 142-149 (2010).
- Gao, R., Stock, A. M. Molecular strategies for phosphorylation-mediated regulation of response regulator activity. Curr Opin Microbiol. 13, 160-167 (2010).
- Hung, D. C., et al. Oligomerization of the response regulator ComE from Streptococcus mutans is affected by phosphorylation. J Bacteriol. 194, 1127-1135 (2012).
- Ladds, J. C., et al. The response regulator Spo0A from Bacillus subtilis is efficiently phosphorylated in Escherichia coli. FEMS Microbiol Lett. 223, 153-157 (2003).
- Wen, Y., Feng, J., Scott, D. R., Marcus, E. A., Sachs, G. Involvement of the HP0165-HP0166 two-component system in expression of some acidic-pH-upregulated genes of Helicobacter pylori. J Bacteriol. 188, 1750-1761 (2006).
- Liu, X., Noll, D. M., Lieb, J. D., Clarke, N. D. DIP-chip: rapid and accurate determination of DNA-binding specificity. Genome Res. 15, 421-427 (2005).
- Gossett, A. J., Lieb, J. D. DNA Immunoprecipitation (DIP) for the Determination of DNA-Binding Specificity. CSH Protoc. 2008, (2008).
- Mascher, T., et al. The Streptococcus pneumoniae cia regulon: CiaR target sites and transcription profile analysis. J Bacteriol. 185, 60-70 (2003).
- Park, P. J. ChIP-seq: advantages and challenges of a maturing technology. Nat Rev Genet. 10, 669-680 (2009).
- Lefrancois, P., Zheng, W., Snyder, M. ChIP-Seq using high-throughput DNA sequencing for genome-wide identification of transcription factor binding sites. Methods Enzymol. 470, 77-104 (2010).
- Ho, J. W., et al. ChIP-chip versus ChIP-seq: lessons for experimental design and data analysis. BMC Genomics. 12, 134 (2011).
- Liu, X., Lee, C. K., Granek, J. A., Clarke, N. D., Lieb, J. D. Whole-genome comparison of Leu3 binding in vitro and in vivo reveals the importance of nucleosome occupancy in target site selection. Genome Res. 16, 1517-1528 (2006).

