מהדמיה מהירה פלואורסצנטי לחוק דיפוזיה מולקולרי בקרום תא חי במיקרוסקופ מסחרי
Summary
Spatial distribution and temporal dynamics of plasma membrane proteins and lipids is a hot topic in biology. Here this issue is addressed by a spatio-temporal image fluctuation analysis that provides conceptually the same physical quantities of single particle tracking, but it uses small molecular labels and standard microscopy setups.
Abstract
זה הפך להיות ברור יותר ויותר כי הפריסה המרחבית והתנועה של רכיבי קרום כמו שומנים וחלבונים הם גורמי מפתח בוויסות של פונקציות רבות תאיות. עם זאת, בשל הדינמיקה מהירה והמבנים הזעירים המעורבים, נדרשת רזולוציה מרחב ובזמן גבוהה מאוד כדי לתפוס את ההתנהגות האמיתית של מולקולות. כאן אנו מציגים את פרוטוקול הניסוי ללימוד הדינמיקה של חלבוני פלזמה קרום fluorescently שהכותרת ושומנים בתאים חיים עם רזולוציה spatiotemporal גבוהה. יש לציין, גישה זו אינה צריכה לעקוב אחר כל מולקולה, אך היא מחשבת התנהגות אוכלוסייה תוך שימוש בכל המולקולות באזור נתון של הקרום. נקודת המוצא היא הדמיה מהירה של אזור נתון על הממברנה. לאחר מכן, פונקצית autocorrelation מרחב ובזמן מוחלטת מחושבת מקשרת תמונות שנרכשו בהגדלת עיכובי זמן, למשל כל 2, 3, n חזרות. ניתן להוכיח כי הרוחבשל השיא של עליות פונקצית autocorrelation מרחבית בהגדלת עיכוב זמן כפונקציה של תנועת חלקיקים עקב דיפוזיה. לכן, ראוי של הסדרה של פונקציות autocorrelation מאפשר לחלץ את החלבון בפועל מתכוון עקירה מרובעת מההדמיה (iMSD), כאן מוצג בצורה של diffusivity לכאורה נגד עקירה ממוצעת. זה מניב מבט כמותית של הדינמיקה הממוצעת של מולקולות בודדות עם דיוק ננומטר. על ידי שימוש בגרסת GFP-tagged של transferrin קולטן (TFR) וATTO488 המסומנת 1-palmitoyl-2-הידרוקסי sn -glycero-3-phosphoethanolamine (PPE) ניתן לצפות ברגולצית spatiotemporal של דיפוזיה חלבון והשומנים על אזורי קרום מיקרומטר בגודל בטווח זמן מיקרו ל-מילי שניות.
Introduction
החל מהמודל המקורי "פסיפס נוזל" על ידי זינגר וניקולסון, התמונה של קרום פלזמה סלולארי כבר מעודכנת באופן רציף במהלך העשורים האחרונים על מנת לכלול את התפקיד המתפתח של תחומים שלד תא ושומנים בדם 1,2.
התצפיות הראשונות התקבלו על ידי שחזור ניאון לאחר photobleaching הסרת לוט (FRAP) שחלק ניכר של חלבונים בממברנה הוא נייח 3-5. מחקריו החלוציים הללו, למרות שמאוד אינפורמטיבי, סבלו מעניה יחסית ברזולוציה במרחב (מיקרון) וזמן (שניות) של מערכי FRAP. כמו כן, להיות מדידת מיצוע הרכב, FRAP חסר במתן מידע על מולקולה בודדת התנהגות.
בהקשר זה, את האפשרות לתייג מולקולה בודדת עם תגים בהירים מאוד (המאפשרים מחקר של המולקולה אחת תהליך דיפוזיה בכל פעם) במיוחד היה מוצלח מאוד. במיוחד, על ידי לחיצה עלרזולוציה של הגישה יחידה חלקיקים מעקב (SPT) ללוח הזמנים מיקרו, Kusumi, et גישה צברה אל. לתכונות ידועות של דינמיקת שומנים וחלבונים שתרמו רב להכרה בתפקידו של שלד קרום מבוסס אקטין בפיזיולוגיה קרום 6 זמן , 7. ממצאים אלה שנוצרו מה שנקרא 'כלונסאות וגדר' מודל, שבו דיפוזיה שומנים והחלבונים מוסדרת על ידי שלד מבוסס אקטין. עם זאת, על מנת לקבל גישה לכמות העצומה של מידע המסופק על ידי נושאים רבים ניסיוניים SPT יש לטפל. במיוחד, הליך התיוג מורכב בדרך כלל על ידי צעדים רבים כמו ייצור, טיהור והכנסה של המינים שכותרתו לתוך המערכת. יתר על כן, חברות תקליטים גדולות, כמו נקודות קוונטיות או חלקיקי מתכת, נדרשות לעתים קרובות כדי להגיע ללוח זמנים משנה אלפית השנייה וcrosslinking של מולקולות המטרה על ידי התווית לא ניתן היה להימנע במקרים רבים. לבסוף, מסלולים רביםיש שיירשם כדי שיתאים לקריטריונים סטטיסטיים ובו זמנית בצפיפות נמוכה של התווית נדרש כדי לאפשר מעקב.
בהשוואה לSPT, ספקטרוסקופיה מתאם הקרינה (FCS), להתגבר על רבים מחסרונות אלה, מייצגת גישה מאוד מבטיחה ללמוד דינמיקה מולקולרית. למעשה, FCS עובד היטב גם עם תוויות עמומות וצפופות, מה שמאפשר ללמוד את הדינמיקה של מולקולות מתויג חלבון פלואורסצנטי בתאי transfected הזמני. כמו כן, זה מאפשר להגיע לסטטיסטיקה גבוהה בכמות מוגבלת של זמן. לבסוף, למרות הצפיפות "הגבוהה" של תוויות FCS מספק מידע מולקולות בודדות. תודה לכל המאפיינים האלה, FCS מייצגת גישה מאוד פשוטה ויושם בהרחבה ללמוד דינמיקת שומנים וחלבונים הן בממברנות מודל ובשידור חיים תאים 8-10. גישות רבות ושונות הוצעו כדי להגביר את היכולת של FCS כדי לחשוף את הפרטים של דיפוזיה מולקולרית. לדוגמא, זה היה shשלו כי על ידי ביצוע FCS באזורי תצפית שונה בגודל אחד יכול להגדיר תכונות "חוק דיפוזיה FCS" מאיר עיניים מוסתרות של תנועה המולקולרית 11,12. חוץ מזה שהיה שונה בגודל, באזור המוקד היה גם משוכפל 13, עבר בחלל לאורך קווי 14-20 או מצומדות עם מצלמות מהירות 21,22. באמצעות המתאם 'מרחב ובזמן "אלה גישות, פרמטרים ביולוגיים רלוונטיים של מספר מרכיבי קרום תוארו כמותית משני קרומי מודל ואלה ביולוגיים בפועל, ובכך תובנה מניבות לארגון המרחבי קרום.
עם זאת, בכל FRAP ויישומי FCS שתואר עד כה בגודל של אזור המוקד מייצג מגבלה ברזולוציה מרחבית שלא ניתן להתגבר עליו. שיטות הדמיה ברזולוציה סופר כמה פותחו לאחרונה כדי לעקוף מגבלה זו. חלקם מבוססים על דיוק לוקליזציה, כגון מיקרוסקופיה אופטית סטוכסטיים שחזור (STORM) <sup> 23,24, מיקרוסקופיה לוקליזציה photoactivation (PALM) 25, PALM הקרינה (FPALM) 26, וPALM מעקב חלקיק יחיד (sptPALM) 27: הכמות הגדולה יחסית של פוטונים הנדרשים בכל תמונה, לעומת זאת, מגביל את הרזולוציה של הזמן שיטות אלה לפחות כמה אלפיות השניה, ובכך פוגעות תחולתם in vivo.
בניגוד לכך, חלופה מבטיחה להדמיה ברזולוציה סופר נפתח על ידי מרחבית ויסות פליטת הקרינה עם שיטות מגורה דלדול פליטה (STED או מעברים הפיכים saturable אופטי הקרינה (RESOLFT)) 28,29. גישות אלה משלבים עיצוב תצפית הנפח נמוך בהרבה מהמגבלה העקיפה עם האפשרות להשתמש מיקרוסקופי סריקה מהירה ומערכות איתור. בשילוב עם ניתוח תנודות הקרינה, מיקרוסקופיה STED אפשרה לחקור ישירות את דינמיקת spatiotemporal ננומטריים של שומנים וproteins בקרום תא חי 30,31.
ניתן להשיג את אותו כמויות הפיזיות של מיקרוסקופיה מבוססת STED ידי ספקטרוסקופיה הותאם מרחב ובזמן מתאם תמונה (STICS 32,33) שיטה שמתאימה ללימוד הדינמיקה של חלבוני fluorescently- מתויג קרום ו / או שומנים בתאים חיים ועל ידי מיקרוסקופ מסחרי. פרוטוקול הניסוי המובא כאן מורכב על ידי כמה צעדים. הראשון דורש הדמיה מהירה של האזור של עניין על הממברנה. לאחר מכן, הערימה של תמונות וכתוצאה מכך משמשת לחישוב פונקציות מתאם מרחב וזמן הממוצע. על ידי התאמת הסדרה של פונקציות מתאם, ניתן להשיג "חוק דיפוזיה 'המולקולרי ישירות מהדמיה בצורה של diffusivity לכאורה (אפליקציה D) – לעומת עלילת עקירת -average. עלילה זו באופן ביקורתי תלויה בסביבה נבחנה על ידי המולקולות ומאפשרת הכרה ישירות מצבי דיפוזיה בפועלשל השומנים בדם / החלבון של עניין.
בפרטים נוספים, כפי שמוצג 34 בעבר, פונקצית קורלציה אוטומטית מרחב ובזמן של סדרת תמונה שנרכשה באופן ביקורתי תלויה בדינמיקה של המולקולות נעות בסדרת התמונה שנאסף (יש לציין כי באותו ההיגיון ניתן להחיל ברכישת אונליין שבו רק ממד אחד בחלל נחשב). בפרט, אנו מגדירים את פונקצית המתאם הגבוהה ככל:
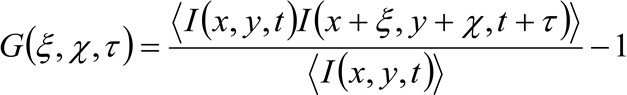 (1)
(1)
בי 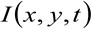 מייצג את עוצמת הקרינה שנמדדה בx העמדה, y ובזמן t,
מייצג את עוצמת הקרינה שנמדדה בx העמדה, y ובזמן t, ![]() ו
ו ![]() מייצג את המרחק בx וכיווני y בהתאמה,
מייצג את המרחק בx וכיווני y בהתאמה, ![]() מייצג את פער הזמן, ו
מייצג את פער הזמן, ו ![]() מייצג את הממוצע. פונקציה זו יכולה לבוא לידי ביטוי כמו:
מייצג את הממוצע. פונקציה זו יכולה לבוא לידי ביטוי כמו:
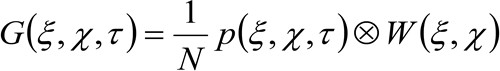 (2)
(2)
שבו 'N' מייצגת את המספר הממוצע של מולקולות באזור התצפית, ![]() מייצגת את פעולת פיתול בחלל, ו
מייצגת את פעולת פיתול בחלל, ו 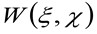 מייצג את autocorrelation של המותניים אינסטרומנטלי. זה אחרון יכול להתפרש כמדד של כמה הפוטונים של פולט אחד פרושים במרחב בשל ההתקנה האופטית / הקלטה (פונקצית התפשטות הנקודה שנקרא, כוחות הביטחון הפלסטיניים, גןעצרת מקורב היטב על ידי פונקצית גאוס). לבסוף,
מייצג את autocorrelation של המותניים אינסטרומנטלי. זה אחרון יכול להתפרש כמדד של כמה הפוטונים של פולט אחד פרושים במרחב בשל ההתקנה האופטית / הקלטה (פונקצית התפשטות הנקודה שנקרא, כוחות הביטחון הפלסטיניים, גןעצרת מקורב היטב על ידי פונקצית גאוס). לבסוף, 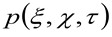 מייצג את ההסתברות למצוא חלקיק במרחק
מייצג את ההסתברות למצוא חלקיק במרחק ![]() ו
ו ![]() לאחר השהיה
לאחר השהיה ![]() . אם ניקח בחשבון את דינמיקת diffusive, שבו חלקיקים לנוע באופן אקראי לכל הכיוונים ונתיבים נטו אינם נוכחים, פונקציה זו גם מקורב היטב על ידי פונקצית גאוס בי השונות יכולות להיות מזוהות כממוצעת כיכר העקירה (MSD) של החלקיקים המרגשים . לפיכך, המותניים של פונקצית המתאם (המכונה גם כ
. אם ניקח בחשבון את דינמיקת diffusive, שבו חלקיקים לנוע באופן אקראי לכל הכיוונים ונתיבים נטו אינם נוכחים, פונקציה זו גם מקורב היטב על ידי פונקצית גאוס בי השונות יכולות להיות מזוהות כממוצעת כיכר העקירה (MSD) של החלקיקים המרגשים . לפיכך, המותניים של פונקצית המתאם (המכונה גם כ ![]() ), יכול להיות מוגדר כסכום של MSDS החלקיקים ומותני אינסטרומנטלי וניתן למדוד על ידי גאוס בכושרטינג של פונקצית המתאם עבור כל עיכוב זמן. אני MSD נמדד ניתן להשתמש כדי לחשב diffusivity לכאורה של המולקולות נעות
), יכול להיות מוגדר כסכום של MSDS החלקיקים ומותני אינסטרומנטלי וניתן למדוד על ידי גאוס בכושרטינג של פונקצית המתאם עבור כל עיכוב זמן. אני MSD נמדד ניתן להשתמש כדי לחשב diffusivity לכאורה של המולקולות נעות ![]() ועקירה ממוצעת
ועקירה ממוצעת ![]() כ:
כ:
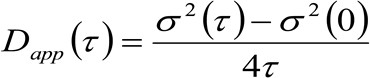 (3)
(3)
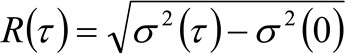 (4)
(4)
שיקולים כמה בהגדרת הניסוי נעשה שימוש יכול להנחות את הקורא בכל הסעיפים הבאים. על מנת לרגש באופן סלקטיבי fluorophores על הקרום הבסיסי של תאי חיים נשתמש תאורה כוללת ההשתקפות פנימית (TIR), תוך שימוש בקרינת TIR מסחרי מיקרוסקופ (TIRF) (ניתן למצוא את פרטים בסעיף החומר). יתר על כן, על מנת לאסוף את ההקרינה דואר נשתמש מטרת הגדלה גבוהה (100x NA 1.47, צמצם מספרי גבוה נדרש לתאורת TIRF) ומצלמת EMCCD (גודל פיזי של פיקסל על מיקרומטר שבב 16). כדי להגיע לגודל פיקסל של 100 ננומטר אנחנו מיישמים עדשת הגדלה נוספת של 1.6x. כפי שנראה להלן, תידרש זמן ברזולוציה מתחת ל -1 msec כדי לתאר כראוי את הדינמיקה של שומני קרום מהירים להלן 100 ננומטר. על מנת להגיע לפתרון זמני זה אנחנו צריכים לבחור אזור של העניין (ROI) קטן יותר מכל השבב של המצלמה (512 x 512). בדרך זו, המצלמה תהיה לקרוא מספר מופחת של קווי הגדלת הרזולוציה הזמן. עם זאת, במשטר קריאה זו מסגרת הזמן יהיה מוגבל על ידי הזמן הנדרש כדי להעביר את החיובים מהחשיפה לשבב הקריאה על המצלמה והיא בדרך כלל בסדר הגודל של אלפיות שנייה לEMCCD 512 x 512 פיקסל. להכות מגבלה זו, טכנולוגיה מתפתחת מאפשרת הסטת ROI-הקווים רק במקום כל המסגרת, with הפחתה יעילה מעשית של הגודל נחשף שבב (הנקרא חיישן קצוץ מצב בEMCCD שלנו). לתצורה זו תהיה אפקטיבית, השבב מחוץ להחזר על ההשקעה חייב להיות מכוסה על ידי כמה חריצים רכובים בנתיב האופטי. הודות להתקנה זו רזולוציה זמן עד ל10 -4 שניות יכולים להיות מושגת. שים לב, עם זאת, כי גישה זו יכולה להיות בשילוב עם setups ניסיוני רב ושונה, כפי שהוסברה בסעיף "הדיון".
הפגנה של השיטה תסופק בתאים חיים, על ידי השימוש בשתי ATTO488 מסומן 1-palmitoyl-2-הידרוקסי sn -glycero-3-phosphoethanolamine (ATTO488-PPE) וגרסה שכותרתו GFP של קולטן transferrin (GFP- TFR). במקרה של ATTO488-PPE גישה זו יכולה לשחזר בהצלחה יישום D כמעט קבוע כפונקציה של תזוזה ממוצעת המצביעה על דיפוזיה לרוב ללא תשלום, כפי שדווחה בעבר 30,35. בניגוד לכך, TFR-GFP תערוכות D יורד <suב יישום> כפונקציה של תזוזה ממוצעת, המצביעה על דיפוזיה באופן חלקי-מוגבל 6. יתר על כן, במקרה האחרון זה אפשרי לכמת את הדיפוזיה המקומית ואזור הכליאה הממוצע מעל מיקרון רב במישור הקרום.
Protocol
Representative Results
Discussion
מעקב אחר חלקיקים בודדים (SPT) מייצג את אחת מהאסטרטגיות הנפוצות ביותר ללמוד דינמיקה מולקולרית ויש לו את היתרון הגדול של מדידת מסלולי חלקיקים. זה בתורו מאפשר חיטוט ההתנהגות של כמה אפילו חלקיקים שכותרתו במערכת מורכבת. עם זאת, כדי להגיע ליתרון זה SPT בדרך כלל זקוק לצפיפות נמ…
Disclosures
The authors have nothing to disclose.
Acknowledgements
This work is supported in part by NIH-P41 P41-RRO3155 and NIH P50-GM076516 (grant to EG), and Fondazione Monte dei Paschi di Siena (grant to FB).
Materials
| Name of Material/ Equipment | Company | Catalog Number | Comments/Description |
| iXon Ultra 897 | Andor | DU-897U-CS0 | |
| Solis | Andor | ||
| CHO-K1 | ATCC | CCL-61 | |
| ATTO 488 labeled PPE | ATTO-TEC GmbH | AD 488-151 | |
| DOPE | Avanti Polar Lipids, Inc. | 850725 | |
| DOTAP | Avanti Polar Lipids, Inc. | 890890 | |
| 100X Penicillin-Streptomycin-Glutamine | Gibco | 10378-016 | |
| DMEM/F-12 | Gibco | 21331 | |
| FBS | Gibco | 10082147 | |
| HEPES | Gibco | 15630-106 | |
| PBS | Gibco | 10010-023 | |
| SimFCS 3.0 | Globals Software | the software can be downloaded here: http://www.lfd.uci.edu/globals/ | |
| DMI6000 with TIRF modulus | Leica | ||
| LAS AF | Leica | ||
| Lipofectamine 2000 | Lipofectamine | 11668019 | |
| Matlab | MathWork | ||
| Imagej | NIH | ||
| C-terminal GFP tagged Tranferrin Receptor | OriGene | RG200980 | |
| Agar | Sigma Aldrich | A5306 | |
| Chloroform | Sigma Aldrich | 528730 | |
| Latex beads, fluorescent yellow-green, 30 nm | Sigma Aldrich | L5155 | |
| SONICA Ultrasonic Cleaners | SOLTEC | ETH S3 | |
| Petri Dishes | Willco | GWSt-3522 | |
| Bio-Format importer for Matlab | http://www.openmicroscopy.org/site/support/bio-formats5/users/matlab/ | ||
| ICS-MatLab Tools | https://www.cellmigration.org/resource/imaging/software/ICSMATLAB_28-02-06.zip | ||
| Simulation by Matlab Tutorial | https://www.cellmigration.org/resource/imaging/icsmatlab/ICSTutorial.html | ||
| Simulation by SimFCS Tutorial | https://www.cellmigration.org/resource/imaging/ppt-pdf/RICS%20Simulations.ppt |
References
- Engelman, D. M. Membranes are more mosaic than fluid. Nature. 438 (7068), 578-580 (2005).
- Vereb, G., et al. yet structured: The cell membrane three decades after the Singer-Nicolson model. Proc. Natl. Acad. Sci. U. S. A. 100 (14), 8053-8058 (1073).
- Ishihara, A., Hou, Y., Jacobson, K. The Thy-1 antigen exhibits rapid lateral diffusion in the plasma membrane of rodent lymphoid cells and fibroblasts. 84 (5), 1290-1293 (1987).
- Axelrod, D., et al. Lateral motion of fluorescently labeled acetylcholine receptors in membranes of developing muscle fibers. Proc. Natl. Acad. Sci. U. S. A. 73 (12), 4594-4598 (1976).
- Jacobson, K., Derzko, Z., Wu, E. S., Hou, Y., Poste, G. Measurement of the lateral mobility of cell surface components in single, living cells by fluorescence recovery after photobleaching. J. Supramol. Struct. 5 (4), 10-1002 (1976).
- Kusumi, A., et al. Paradigm shift of the plasma membrane concept from the two-dimensional continuum fluid to the partitioned fluid: high-speed single-molecule tracking of membrane molecules. Annu. Rev. Biophys. Biomol. Struct. 34, 351-378 (2005).
- Kusumi, A., Ike, H., Nakada, C., Murase, K., Fujiwara, T. Single-molecule tracking of membrane molecules: plasma membrane compartmentalization and dynamic assembly of raft-philic signaling molecules. Semin. Immunol. 17 (1), 3-21 (2005).
- Schwille, P., Korlach, J., Webb, W. W. Fluorescence correlation spectroscopy with single-molecule sensitivity on cell and model membranes. Cytometry. 36, 176-182 (1999).
- Gielen, E., et al. Diffusion of sphingomyelin and myelin oligodendrocyte glycoprotein in the membrane of OLN-93 oligodendroglial cells studied by fluorescence correlation spectroscopy. C. R. Biol. 328 (12), 1057-1064 (2005).
- Weiss, M., Hashimoto, H., Nilsson, T. Anomalous protein diffusion in living cells as seen by fluorescence correlation spectroscopy. Biophys. J. 84, 4043-4052 (2003).
- Wawrezinieck, L., Rigneault, H., Marguet, D., Lenne, P. F. Fluorescence correlation spectroscopy diffusion laws to probe the submicron cell membrane organization. Biophys. J. 89 (6), 4029-4042 (2005).
- Lenne, P. F., et al. Dynamic molecular confinement in the plasma membrane by microdomains and the cytoskeleton meshwork. EMBO J. 25 (14), 3245-3256 (2006).
- Ries, J., Schwille, P. Studying slow membrane dynamics with continuous wave scanning fluorescence correlation spectroscopy. Biophys. J. 91 (5), 1915-1924 (2006).
- Ruan, Q., Cheng, M. A., Levi, M., Gratton, E., Mantulin, W. W. Spatial-temporal studies of membrane dynamics: scanning fluorescence correlation spectroscopy (SFCS). Biophys. J. 87 (2), 1260-1267 (2004).
- Berland, K. M., So, P. T., Chen, Y., Mantulin, W. W., Gratton, E. Scanning two-photon fluctuation correlation spectroscopy: particle counting measurements for detection of molecular aggregation. Biophys. J. 71, 410-420 (1996).
- Heinemann, F., Betaneli, V., Thomas, F. A., Schwille, P. Quantifying lipid diffusion by fluorescence correlation spectroscopy: a critical treatise. Langmuir. 28 (37), 13395-13404 (2012).
- Cardarelli, F., Lanzano, L., Gratton, E. Capturing directed molecular motion in the nuclear pore complex of live cells. Proc. Natl. Acad. Sci. U. S. A. 109 (25), 9863-9868 (2012).
- Sanchez, S. A., Tricerri, M. A., Gratton, E. Laurdan generalized polarization fluctuations measures membrane packing micro-heterogeneity in vivo. Proc. Natl. Acad. Sci. U. S. A. 109 (19), 7314-7319 (2012).
- Cardarelli, F., Lanzano, L., Gratton, E. Fluorescence correlation spectroscopy of intact nuclear pore complexes. Biophys. J. 101 (4), 27-29 (2012).
- Di Rienzo, C., et al. Unveiling LOX-1 receptor interplay with nanotopography: mechanotransduction and atherosclerosis onset. Sci. Rep. 3, 10-1038 (2013).
- Unruh, J. R., Gratton, E. Analysis of molecular concentration and brightness from fluorescence fluctuation data with an electron multiplied CCD camera. Biophys. J. 95 (11), 5385-5398 (2008).
- Kannan, B., et al. Electron multiplying charge-coupled device camera based fluorescence correlation spectroscopy. Anal. Chem. 78 (10), 3444-3451 (2006).
- Jones, S. A., Shim, S. H., He, J., Fast Zhuang, X. three-dimensional super-resolution imaging of live cells. Nat. Methods. 8 (6), 499-508 (2011).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy. 3 (10), 793-795 (2006).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Hess, S. T., Girirajan, T. P., Mason, M. D. Ultra-high resolution imaging by fluorescence photoactivation localization microscopy. Biophys. J. 91 (11), 4258-4272 (2006).
- Manley, S., et al. High-density mapping of single-molecule trajectories with photoactivated localization microscopy. Nat. Methods. 5 (2), 155-157 (2008).
- Hell, S. W. Far-field optical nanoscopy. Science. 316 (5828), 1153-1158 (2007).
- Klar, T. A., Hell, S. W. Subdiffraction resolution in far-field fluorescence microscopy. Opt. Lett. 24 (14), 954-956 (1999).
- Eggeling, C., et al. Direct observation of the nanoscale dynamics of membrane lipids in a living cell. Nature. 457 (7233), 1159-1162 (2009).
- Hedde, P. N., et al. Stimulated emission depletion-based raster image correlation spectroscopy reveals biomolecular dynamics in live cells. Nat. Commun. 4, .
- Hebert, B., Costantino, S., Wiseman, P. W. Spatiotemporal image correlation spectroscopy (STICS) theory, verification, and application to protein velocity mapping in living CHO cells. Biophys. J. 88 (5), 3601-3614 (2005).
- Brown, C. M., et al. Probing the integrin-actin linkage using high-resolution protein velocity mapping. J. Cell Sci. 119, 5204-5214 (2006).
- Di Rienzo, C., Gratton, E., Beltram, F., Cardarelli, F. Fast spatiotemporal correlation spectroscopy to determine protein lateral diffusion laws in live cell membranes. Proc. Natl. Acad. Sci. U. S. A. 110 (30), 12307-12312 (2013).
- Mueller, V., et al. STED nanoscopy reveals molecular details of cholesterol- and cytoskeleton-modulated lipid interactions in living cells. Biophys. J. 101 (7), 1651-1660 (2011).
- Kleusch, C., Hersch, N., Hoffmann, B., Merkel, R., Csiszar, A. Fluorescent lipids: functional parts of fusogenic liposomes and tools for cell membrane labeling and visualization. Molecules. 17 (1), 1055-1073 (2012).
- Ries, J., Chiantia, S., Schwille, P. Accurate determination of membrane dynamics with line-scan FCS. Biophys. J. 96 (5), 1999-2008 (2009).
- Kolin, D. L., Wiseman, P. W. Advances in image correlation spectroscopy: measuring number densities, aggregation states, and dynamics of fluorescently labeled macromolecules in cells. Cell Biochem. Biophys. 49 (3), 141-164 (2007).
- Digman, M. A., et al. Measuring fast dynamics in solutions and cells with a laser scanning microscope. Biophys. J. 89 (2), 1317-1327 (2005).
- Ritchie, K., et al. Detection of non-Brownian diffusion in the cell membrane in single molecule tracking. Biophys. J. 88 (3), 2266-2277 (2005).
- Voie, A. H., Burns, D. H., Spelman, F. A. Orthogonal-plane fluorescence optical sectioning: three-dimensional imaging of macroscopic biological specimens. J. Microsc. 170, 229-236 (1993).
- Huisken, J., Swoger, J., Del Bene, ., Wittbrodt, F., J, E. H., Stelzer, Optical sectioning deep inside live embryos by selective plane illumination microscopy. Science. 305 (5686), 1007-1009 (2004).
- Wohland, T., Shi, X., Sankaran, J., Stelzer, E. H. Single plane illumination fluorescence correlation spectroscopy (SPIM-FCS) probes inhomogeneous three-dimensional environments. Opt. Express. 18 (10), 10627-10641 (2010).

