Screening voor Functionele niet-coderende genetische varianten Met behulp van Electroforetische Mobility Shift Assay (EMSA) en DNA-affiniteit Neerslag Assay (DAPA)
Summary
We present a strategic plan and protocol for identifying non-coding genetic variants affecting transcription factor (TF) DNA binding. A detailed experimental protocol is provided for electrophoretic mobility shift assay (EMSA) and DNA affinity precipitation assay (DAPA) analysis of genotype-dependent TF DNA binding.
Abstract
Population and family-based genetic studies typically result in the identification of genetic variants that are statistically associated with a clinical disease or phenotype. For many diseases and traits, most variants are non-coding, and are thus likely to act by impacting subtle, comparatively hard to predict mechanisms controlling gene expression. Here, we describe a general strategic approach to prioritize non-coding variants, and screen them for their function. This approach involves computational prioritization using functional genomic databases followed by experimental analysis of differential binding of transcription factors (TFs) to risk and non-risk alleles. For both electrophoretic mobility shift assay (EMSA) and DNA affinity precipitation assay (DAPA) analysis of genetic variants, a synthetic DNA oligonucleotide (oligo) is used to identify factors in the nuclear lysate of disease or phenotype-relevant cells. For EMSA, the oligonucleotides with or without bound nuclear factors (often TFs) are analyzed by non-denaturing electrophoresis on a tris-borate-EDTA (TBE) polyacrylamide gel. For DAPA, the oligonucleotides are bound to a magnetic column and the nuclear factors that specifically bind the DNA sequence are eluted and analyzed through mass spectrometry or with a reducing sodium dodecyl sulfate polyacrylamide gel electrophoresis (SDS-PAGE) followed by Western blot analysis. This general approach can be widely used to study the function of non-coding genetic variants associated with any disease, trait, or phenotype.
Introduction
Sequencing en genotypering op basis van studies, met inbegrip van Genome-Wide Association Studies (GWAS), kandidaat locus studies, en diep-sequencing studies, hebben veel genetische varianten die statistisch worden geassocieerd met een ziekte, trek, of fenotype geïdentificeerd. Anders dan vroege voorspellingen meeste van deze varianten (85-93%) zijn in niet-coderende gebieden en niet de aminozuursequentie van eiwitten 1,2 niet veranderen. De functie van deze niet-coderende varianten interpreteren en die de biologische mechanismen ze op de geassocieerde ziekte heeft eigenschap of fenotype bewezen uitdagende 3-6. genexpressie – We hebben een algemene strategie om de moleculaire mechanismen die varianten verwijzen naar een belangrijk tussenproduct fenotype te identificeren ontwikkeld. Deze leiding is speciaal ontworpen om modulatie van TF binding door genetische varianten te identificeren. Deze strategie combineert computationele benaderingen en moleculair biologische technieken om te voorspellenbiologische effecten van kandidaat-varianten in silico en controleer deze voorspellingen empirisch (figuur 1).
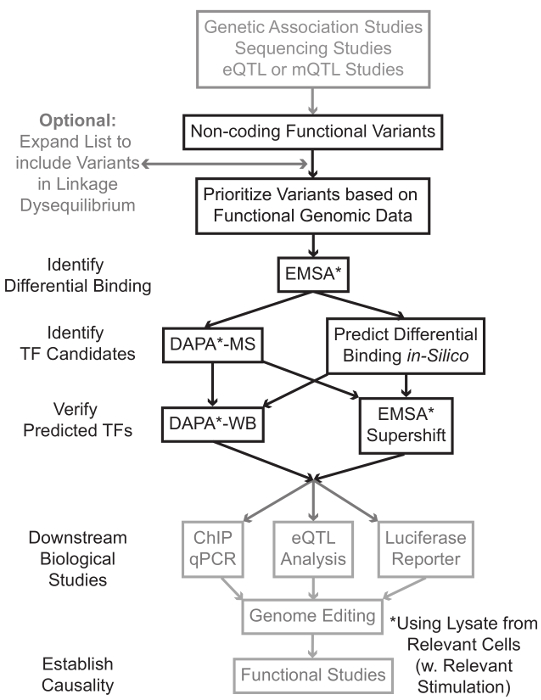
Figuur 1:.. Strategische aanpak voor de analyse van niet-coderende genetische varianten stappen die niet zijn opgenomen in de gedetailleerde protocol in verband met dit manuscript zijn grijs Klik hier om een grotere versie van deze figuur te bekijken.
In veel gevallen is het belangrijk om te beginnen uitbreiding van de lijst van varianten die allemaal in hoge linkage-onevenwicht (LD) met elkaar verbonden statistisch variant omvatten. LD is een maat voor niet-willekeurige associatie van allelen op twee verschillende chromosomale posities, die kan worden gemeten door de r 2 7 statistiek. r 2 is een maat voor de linKage onevenwichtigheid tussen twee varianten, met een r 2 = 1 aangeeft perfecte verbinding tussen twee varianten. Allelen hoge LD blijken co-segregeren op het chromosoom in voorouderlijke populaties. Huidige genotypering arrays niet alle bekende varianten in het menselijk genoom. In plaats daarvan gebruik maken van de LD in het menselijk genoom en omvatten een subset van de bekende varianten die als proxies voor andere varianten binnen een bepaalde regio van LD 8. Aldus kan een variant zonder biologische gevolg worden geassocieerd met een bepaalde ziekte omdat het in LD met de causale variant-de variant met een zinvolle biologisch effect. Procedureel, is het raadzaam om te zetten van de nieuwste versie van de 1000 genomen projecteren 9 variant call-bestanden (VCF) in binaire bestanden compatibel zijn met Plink 10,11, een open-source tool voor het gehele genoom vereniging analyse. Vervolgens worden alle andere genetische varianten met LD r 2> 0,8 bij elke ingang genetische variant kunnen worden geïdentificeerd als kandidaten. Het is belangrijk om de overeenkomstige referentiegroep voor dit stap- bijvoorbeeld als een variant geïdentificeerd bij personen van Europese afkomst, de data van onderwerpen van soortgelijke afkomst moeten worden gebruikt voor LD expansie.
LD expansie resulteert vaak in tientallen kandidaat varianten, en het is waarschijnlijk dat slechts een klein deel daarvan bijdragen aan ziektemechanisme. Vaak is onhaalbaar experimenteel onderzoeken elk van deze varianten afzonderlijk. Daarom is het nuttig om de duizenden publiekelijk beschikbare functionele genomische datasets te benutten als een filter om voorrang te geven aan de varianten. Zo heeft de ENCODE consortium 12 duizenden ChIP-seq experimenten waarin de binding van TF en co-factoren en histon markeringen uitgevoerd in diverse contexten, samen met chromatine bereikbaarheidsgegevens van technologieën zoals DNase-seq 13, ATAC -seq 14 en FAIRE-seq 15. databases en webservers zoals de UCSC Genome Browser 16, Roadmap Epigenomics 17, Blueprint Epigenome 18, Cistrome 19 en Remap 20 bieden gratis toegang tot de gegevens die door deze en andere experimentele technieken in een breed scala van celtypen en voorwaarden. Wanneer er te veel varianten experimentele onderzoeken, kunnen deze gegevens worden gebruikt om prioriteit die zich binnen waarschijnlijk regulerende gebieden desbetreffende cel- en weefseltypen. Verder wanneer een variant binnen een ChIP-seq piek voor een specifiek eiwit, deze gegevens kunnen potentiële leads geven over de specifieke TF (s) of co-factoren waarvan de binding zou kunnen beïnvloeden.
Vervolgens worden de resulterende prioriteit varianten experimenteel gescreend om voorspelde genotype-afhankelijke eiwit binding te valideren met behulp van EMSA 21,22. EMSA meet de verandering in de migratie van de oligo op een niet-reducerende TBE gel. Fluorescent gemerkte oligo wordt geïncubeerd met denucleaire lysaat en binding van nucleaire factoren de beweging van de oligo vertragen op de gel. Op deze wijze oligo dat meer kernfactoren is gebonden zal een sterker fluorescentiesignaal op scantoepassingen. Met name EMSA geen voorspellingen over de specifieke eiwitten waarvan de binding zal worden beïnvloed vereisen.
Zodra varianten zijn geïdentificeerd die zich binnen het voorspelde regelende gebieden en kunnen verschillend binding kernfactoren zijn computationele werkwijzen toegepast om de specifieke TF (n) waarvoor bindende ze invloed kunnen voorspellen. Wij geven de voorkeur aan CIS-BP 23,24, RegulomeDB 25, UniProbe 26 en JASPAR 27 gebruiken. Zodra de kandidaat-TF's worden geïdentificeerd, kunnen deze voorspellingen specifiek worden getest met behulp van antilichamen tegen deze TF's (EMSA-supershifts en DAPA-Westerns). Een EMSA-supershift bij de bereiding een TF-antilichamen voor de nucleaire lysaat en oligo. Een positief resultaat in een EMSA-supershift is represented een verdere verschuiving in de EMSA band, of een verlies van de band (besproken in referentie 28). In de complementaire DAPA, een 5'-gebiotinyleerde oligo duplex die de variant en 20 basenparen flankerende nucleotiden zijn geïncubeerd met nucleaire lysaat uit relevante type (s) cel nucleaire factoren die specifiek binden de oligo vangen. De duplex-oligo nucleaire factor complex geïmmobiliseerd op streptavidine microkralen in een magnetische kolom. De gebonden nucleaire factoren worden rechtstreeks verzameld via elutie 29,48. Bindende voorspellingen kunnen dan worden beoordeeld door een Western blot met gebruik van antilichamen specifiek voor het eiwit. In gevallen waarin er geen duidelijke voorspellingen, of te veel voorspellingen, de eluties van variant pull-downs van de DAPA experimenten kan een proteomics kern worden gestuurd om de kandidaat-TF's te identificeren met behulp van massaspectrometrie, die vervolgens kunnen worden gevalideerd met behulp van deze eerder beschreven werkwijzen.
In de rest van de article, is de gedetailleerde protocol voor het EMSA en DAPA analyse van genetische varianten voorzien.
Protocol
Representative Results
Discussion
Hoewel vooruitgang in sequencing en genotypering technologieën zijn sterk verbeterd ons vermogen om genetische varianten geassocieerd met de ziekte te identificeren, is ons vermogen om de functionele mechanismen beïnvloed door deze varianten begrijpen achter. Een belangrijke bron van het probleem is dat veel ziekte-geassocieerde varianten liggen in n on-coderende gebieden van het genoom, die waarschijnlijk van invloed moeilijker te voorspellen mechanismen die genexpressie. Hier presenteren we een protocol gebase…
Disclosures
The authors have nothing to disclose.
Acknowledgements
We thank Erin Zoller, Jessica Bene, and Lindsey Hays for input and direction in protocol development. MTW was supported in part by NIH R21 HG008186 and a Trustee Award grant from the Cincinnati Children’s Hospital Research Foundation. ZHP was supported in part by T32 GM063483-13.
Materials
| Custom DNA Oligonucleotides | Integrated DNA Technologies | http://www.idtdna.com/site/order/oligoentry | |
| Potassium Chloride | Fisher Scientific | BP366-500 | KCl, for CE buffer |
| HEPES (1M) | Fisher Scientific | 15630-080 | For CE and NE buffer |
| EDTA (0.5M), pH 8.0 | Life Technologies | R1021 | For CE, NE, and annealing buffer |
| Sodium Chloride | Fisher Scientific | BP358-1 | NaCl, for NE buffer |
| Tris-HCl (1M), pH 8.0 | Invitrogen | BP1756-100 | For annealing buffer |
| Phosphate Buffered Saline (1X) | Fisher Scientific | MT21040CM | PBS, for cell wash |
| DL-Dithiothreitol solution (1M) | Sigma | 646563 | Reducing agent |
| PMSF | Thermo Scientific | 36978 | Protease Inhibitor |
| Phosphatase Inhibitor Cocktail | Thermo Scientific | 78420 | Prevents dephosphorylation of TFs |
| Nonidet P-40 Substitute | IBI Scientific | IB01140 | NP-40, for nuclear extraction |
| BCA Protein Assay Kit | Thermo Scientific | 23225 | For measuring protein concentration |
| Odyssey EMSA Buffer Kit | Licor | 829-07910 | Contains all necessary EMSA buffers |
| TBE Gels, 6%, 12 Wells | Invitrogen | EC6265BOX | For EMSA |
| TBE Buffer (10X) | Thermo Scientific | B52 | For EMSA |
| FactorFinder Starting Kit | Miltenyi Biotec | 130-092-318 | Contains all necessary DAPA buffers |
| Licor Odyssey CLx | Licor | Recommended scanner for DAPA/EMSA | |
| Antibiotic-Antimycotic | Gibco | 15240-062 | Contains 10,000 units/mL of penicillin, 10,000 µg/mL of streptomycin, and 25 µg/mL of Fungizone® Antimycotic |
| Fetal Bovine Serum | Gibco | 26140-079 | FBS, for culture media |
| RPMI 1640 Medium | Gibco | 22400-071 | Contains L-glutamine and 25mM HEPES |
References
- Hindorff, L. A., et al. Potential etiologic and functional implications of genome-wide association loci for human diseases and traits. Proc Natl Acad Sci U S A. 106 (23), 9362-9367 (2009).
- Maurano, M. T., et al. Systematic localization of common disease-associated variation in regulatory DNA. Science. 337 (6099), 1190-1195 (2012).
- Ward, L. D., Kellis, M. Interpreting noncoding genetic variation in complex traits and human disease. Nat Biotechnol. 30 (11), 1095-1106 (2012).
- Paul, D. S., Soranzo, N., Beck, S. Functional interpretation of non-coding sequence variation: concepts and challenges. Bioessays. 36 (2), 191-199 (2014).
- Zhang, F., Lupski, J. R. Non-coding genetic variants in human disease. Hum Mol Genet. , (2015).
- Lee, T. I., Young, R. A. Transcriptional regulation and its misregulation in disease. Cell. 152 (6), 1237-1251 (2013).
- Slatkin, M. Linkage disequilibrium–understanding the evolutionary past and mapping the medical future. Nat Rev Genet. 9 (6), 477-485 (2008).
- Bush, W. S., Moore, J. H. Chapter 11: Genome-wide association studies. PLoS Comput Biol. 8 (12), e1002822 (2012).
- 1000 Genomes Project Consortium. An integrated map of genetic variation from 1,092 human genomes. Nature. 491 (7422), 56-65 (2012).
- Chang, C. C., et al. Second-generation PLINK: rising to the challenge of larger and richer datasets. Gigascience. 4, 7 (2015).
- Purcell, S., et al. PLINK: a tool set for whole-genome association and population-based linkage analyses. Am J Hum Genet. 81 (3), 559-575 (2007).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Crawford, G. E., et al. Genome-wide mapping of DNase hypersensitive sites using massively parallel signature sequencing (MPSS). Genome Res. 16 (1), 123-131 (2006).
- Buenrostro, J. D., Giresi, P. G., Zaba, L. C., Chang, H. Y., Greenleaf, W. J. Transposition of native chromatin for fast and sensitive epigenomic profiling of open chromatin, DNA-binding proteins and nucleosome position. Nat Methods. 10 (12), 1213-1218 (2013).
- Giresi, P. G., Kim, J., McDaniell, R. M., Iyer, V. R., Lieb, J. D. FAIRE Formaldehyde-Assisted Isolation of Regulatory Elements) isolates active regulatory elements from human chromatin. Genome Res. 17 (6), 877-885 (2007).
- Kent, W. J., et al. The human genome browser at UCSC. Genome Res. 12 (6), 996-1006 (2002).
- Roadmap Epigenomics Consortium. Integrative analysis of 111 reference human epigenomes. Nature. 518 (7539), 317-330 (2015).
- Martens, J. H., Stunnenberg, H. G. BLUEPRINT: mapping human blood cell epigenomes. Haematologica. 98 (10), 1487-1489 (2013).
- Liu, T., et al. Cistrome: an integrative platform for transcriptional regulation studies. Genome Biol. 12 (8), R83 (2011).
- Griffon, A., et al. Integrative analysis of public ChIP-seq experiments reveals a complex multi-cell regulatory landscape. Nucleic Acids Res. 43 (4), e27 (2015).
- Staudt, L. M., et al. A lymphoid-specific protein binding to the octamer motif of immunoglobulin genes. Nature. 323 (6089), 640-643 (1986).
- Singh, H., Sen, R., Baltimore, D., Sharp, P. A. A nuclear factor that binds to a conserved sequence motif in transcriptional control elements of immunoglobulin genes. Nature. 319 (6049), 154-158 (1986).
- Weirauch, M. T., et al. Determination and inference of eukaryotic transcription factor sequence specificity. Cell. 158 (6), 1431-1443 (2014).
- Ward, L. D., Kellis, M. HaploReg: a resource for exploring chromatin states, conservation, and regulatory motif alterations within sets of genetically linked variants. Nucleic Acids Res. 40 (Database issue), D930-D934 (2012).
- Boyle, A. P., et al. Annotation of functional variation in personal genomes using RegulomeDB. Genome Res. 22 (9), 1790-1797 (2012).
- Hume, M. A., Barrera, L. A., Gisselbrecht, S. S., Bulyk, M. L. UniPROBE, update 2015: new tools and content for the online database of protein-binding microarray data on protein-DNA interactions. Nucleic Acids Res. 43 (Database issue), D117-D122 (2015).
- Mathelier, A., et al. JASPAR 2014: an extensively expanded and updated open-access database of transcription factor binding profiles. Nucleic Acids Res. 42 (Database issue), 142-147 (2014).
- Smith, M. F., Delbary-Gossart, S. Electrophoretic Mobility Shift Assay (EMSA). Methods Mol Med. 50, 249-257 (2001).
- Franza, B. R., Josephs, S. F., Gilman, M. Z., Ryan, W., Clarkson, B. Characterization of cellular proteins recognizing the HIV enhancer using a microscale DNA-affinity precipitation assay. Nature. 330 (6146), 391-395 (1987).
- . BCA Protein Assay Kit: User Guide Available from: https://tools.thermofisher.com/content/sfs/manuals/MAN0011430_Pierce_BCA_Protein_Asy_UG.pdf (2014)
- Wijeratne, A. B., et al. Phosphopeptide separation using radially aligned titania nanotubes on titanium wire. ACS Appl Mater Interfaces. 7 (21), 11155-11164 (2015).
- Silva, J. M., McMahon, M. The Fastest Western in Town: A Contemporary Twist on the Classic Western Blot Analysis. J. Vis. Exp. (84), (2014).
- Lu, X., et al. Lupus Risk Variant Increases pSTAT1 Binding and Decreases ETS1 Expression. Am J Hum Genet. 96 (5), 731-739 (2015).
- Ramana, C. V., Chatterjee-Kishore, M., Nguyen, H., Stark, G. R. Complex roles of Stat1 in regulating gene expression. Oncogene. 19 (21), 2619-2627 (2000).
- Fillebeen, C., Wilkinson, N., Pantopoulos, K. Electrophoretic Mobility Shift Assay (EMSA) for the Study of RNA-Protein Interactions: The IRE/IRP Example. J. Vis. Exp. (94), e52230 (2014).
- Heng, T. S., Painter, M. W. Immunological Genome Project, C. The Immunological Genome Project: networks of gene expression in immune cells. Nat Immunol. 9 (10), 1091-1094 (2008).
- Wu, C., et al. BioGPS: an extensible and customizable portal for querying and organizing gene annotation resources. Genome Biol. 10 (11), R130 (2009).
- Wu, C., Macleod, I., Su, A. I. BioGPS and MyGene.info: organizing online, gene-centric information. Nucleic Acids Res. 41 (Database issue), D561-D565 (2013).
- Wang, J., et al. Sequence features and chromatin structure around the genomic regions bound by 119 human transcription factors. Genome Res. 22 (9), 1798-1812 (2012).
- Holden, N. S., Tacon, C. E. Principles and problems of the electrophoretic mobility shift assay. J Pharmacol Toxicol Methods. 63 (1), 7-14 (2011).
- Xu, J., Liu, H., Park, J. S., Lan, Y., Jiang, R. Osr1 acts downstream of and interacts synergistically with Six2 to maintain nephron progenitor cells during kidney organogenesis. Development. 141 (7), 1442-1452 (2014).
- Yang, T. -. P., et al. Genevar: a database and Java application for the analysis and visualization of SNP-gene associations in eQTL studies. Bioinformatics. 26 (19), 2474-2476 (2010).
- Fort, A., et al. A liver enhancer in the fibrinogen gene cluster. Blood. 117 (1), 276-282 (2011).
- Solberg, N., Krauss, S. Luciferase assay to study the activity of a cloned promoter DNA fragment. Methods Mol Biol. 977, 65-78 (2013).
- Rahman, M., et al. A repressor element in the 5′-untranslated region of human Pax5 exon 1A. Gene. 263 (1-2), 59-66 (2001).
- Mali, P., et al. RNA-Guided Human Genome Engineering via Cas9. Science. 339 (6121), 823-826 (2013).

