Screening für Funktionelle nichtkodierenden genetischen Varianten Mit elektrophoretische Mobilitäts-Shift-Assay (EMSA) und DNA-Affinitätspräzipitation Assay (DAPA)
Summary
We present a strategic plan and protocol for identifying non-coding genetic variants affecting transcription factor (TF) DNA binding. A detailed experimental protocol is provided for electrophoretic mobility shift assay (EMSA) and DNA affinity precipitation assay (DAPA) analysis of genotype-dependent TF DNA binding.
Abstract
Population and family-based genetic studies typically result in the identification of genetic variants that are statistically associated with a clinical disease or phenotype. For many diseases and traits, most variants are non-coding, and are thus likely to act by impacting subtle, comparatively hard to predict mechanisms controlling gene expression. Here, we describe a general strategic approach to prioritize non-coding variants, and screen them for their function. This approach involves computational prioritization using functional genomic databases followed by experimental analysis of differential binding of transcription factors (TFs) to risk and non-risk alleles. For both electrophoretic mobility shift assay (EMSA) and DNA affinity precipitation assay (DAPA) analysis of genetic variants, a synthetic DNA oligonucleotide (oligo) is used to identify factors in the nuclear lysate of disease or phenotype-relevant cells. For EMSA, the oligonucleotides with or without bound nuclear factors (often TFs) are analyzed by non-denaturing electrophoresis on a tris-borate-EDTA (TBE) polyacrylamide gel. For DAPA, the oligonucleotides are bound to a magnetic column and the nuclear factors that specifically bind the DNA sequence are eluted and analyzed through mass spectrometry or with a reducing sodium dodecyl sulfate polyacrylamide gel electrophoresis (SDS-PAGE) followed by Western blot analysis. This general approach can be widely used to study the function of non-coding genetic variants associated with any disease, trait, or phenotype.
Introduction
Sequenzierung und Genotypisierung basierte Studien, einschließlich der genomweiten Assoziationsstudien (GWAS), Kandidaten-Locus Studien und tiefSequenzierungsUntersuchungen haben viele genetische Varianten identifiziert, die statistisch im Zusammenhang mit einer Krankheit, Zug oder Phänotyp. Im Gegensatz zu dem frühen Vorhersagen, die meisten dieser Varianten (85-93%) werden in nicht-codierenden Regionen und die Aminosäuresequenz von Proteinen nicht 1,2 ändern. Interpretieren der Funktion dieser nicht-kodierenden Varianten und Bestimmung der biologischen Mechanismen , um sie zu dem zugeordneten Krankheit verbindet, trait oder Phänotyp hat Herausforderung erwiesen 3-6. Wir haben eine allgemeine Strategie entwickelt, um die molekularen Mechanismen zu identifizieren, die Varianten zu einem wichtigen Zwischen Phänotyp verknüpfen – Genexpression. Diese Pipeline wird speziell zu identifizieren, die Modulation der TF-Bindung durch genetische Varianten entwickelt. Diese Strategie kombiniert Rechenansätze und Techniken der Molekularbiologie Ziel vorherzusagenbiologische Wirkungen von Kandidatenvarianten in silico, und überprüfen diese Prognosen empirisch (Abbildung 1).
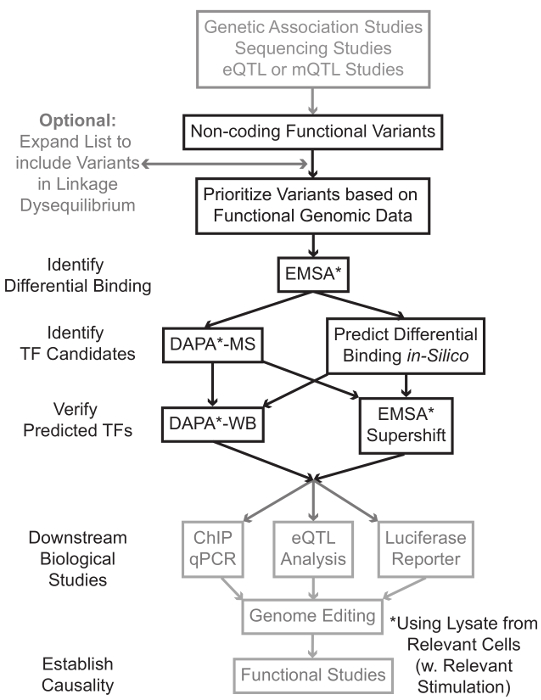
Abbildung 1:.. Strategischer Ansatz für die Analyse von nicht-kodierenden genetischen Varianten Schritte, die in der detaillierten Protokoll sind nicht mit diesem Manuskript assoziiert enthalten sind grau schattiert Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
In vielen Fällen ist es wichtig, durch eine Erweiterung der Liste der Varianten zu beginnen alle, die in hohen Gestänge-Ungleichgewichts (LD) mit jeder statistisch assoziiert Variante enthalten. LD ist ein Maß der nicht-zufällige Assoziation von Allelen an zwei verschiedenen chromosomalen Stellen, die durch die r 2 -Statistik 7 gemessen werden kann. r 2 ist ein Maß für die linKage Ungleichgewichts zwischen zwei Varianten, mit einem r 2 = 1 bezeichnet perfekte Verbindung zwischen zwei Varianten. Allelen in hohen LD gefunden auf dem Chromosom über Vorfahren Populationen zusammen entmischen. Aktuelle Genotypisierung Arrays enthalten nicht alle bekannten Varianten im menschlichen Genom. Stattdessen verwerten sie die LD innerhalb des menschlichen Genoms und sind eine Teilmenge der bekannten Varianten , die 8 für andere Varianten innerhalb einer bestimmten Region des LD als Stellvertreter fungieren. Somit kann mit einer bestimmten Krankheit assoziiert sein eine Variante ohne biologische Folge kann, da es in LD mit dem kausalen varianten der Variante mit einer sinnvollen biologischen Wirkung. Prozedural, wird empfohlen , die neueste Version der 1000 Genome Projekt 9 Variante Call – Dateien (VCF) in binäre Dateien zu konvertieren kompatibel mit PLINK 10,11, einem Open-Source – Tool für Genom – Assoziations – Analyse. Anschließend werden alle anderen genetischen Varianten mit LD r 2> 0,8 mit jedem Eingang genetischen variant als Kandidaten identifiziert werden. Es ist wichtig , die entsprechende Referenzpopulation für diese Schritt- zB zu verwenden, wenn eine Variante , bei Themen von europäischer Abstammung identifiziert wurde, Daten von Patienten mit ähnlicher Herkunft sollte für LD Expansion verwendet werden.
LD Expansion resultiert oft in Dutzenden von Kandidatenvarianten, und es ist wahrscheinlich, dass nur ein kleiner Bruchteil von diesen zu Krankheitsmechanismus beizutragen. Oft ist es nicht durchführbar, experimentell jede dieser Varianten einzeln untersuchen. Es ist daher sinnvoll, die Tausende von öffentlich verfügbaren funktionellen genomischen Datensätze als Filter zu nutzen, um die Varianten zu priorisieren. Zum Beispiel 12 das ENCODE – Konsortium hat Tausende von ChIP-seq Experimente beschreiben die Bindung von Transkriptionsfaktoren und Co-Faktoren, und Histonmarkierungen in einem breiten Spektrum von Kontexten, zusammen mit Chromatin Zugänglichkeit von Daten von Technologien wie DNase-seq 13, ATAC ausgeführt -SEQ 14 und FAIRE-seq 15. DatabAses und Web – Servern wie dem UCSC Genome Browser 16, Roadmap Epigenomics 17, Epigenome Blueprint 18, Cistrome 19 und ReMap 20 bieten freien Zugang zu Daten , die durch diese und andere experimentelle Techniken in einer Vielzahl von Zelltypen und Bedingungen hergestellt. Wenn es zu viele Varianten sind experimentell zu untersuchen, können diese Daten verwendet werden, die innerhalb wahrscheinlich regulatorischen Regionen in entsprechenden Zell- und Gewebetypen angeordnet zu priorisieren. Ferner wird in Fällen, in denen eine Variante für ein spezifisches Protein in einer ChIP-seq peak ist, können diese Daten Potential führt als der spezifischen TF (s) oder Cofaktoren schaffen, deren Bindung könnte beeinflussen.
Als nächstes werden die resultierenden priorisiert Varianten gescreent experimentell vorhergesagt Genotyp-abhängige Protein zu validieren Bindungs 21,22 mittels EMSA. EMSA misst die Veränderung in der Migration des oligo auf einem nicht-reduzierenden TBE-Gel. Fluoreszierend markiertes Oligo mit der inkubiertenKern Lysat, und die Bindung von Kernfaktoren wird verzögern die Bewegung des Oligo auf dem Gel. Auf diese Weise Oligo, die mehr Kernfaktoren gebunden hat, als ein stärkeres Fluoreszenzsignal beim Abtasten präsentieren wird. Bemerkenswert ist, erfordert keine EMSA Prognosen über die spezifischen Proteine, deren Bindung betroffen.
Sobald Varianten identifiziert werden, die innerhalb der erwarteten regulatorischen Regionen befinden und in der Lage sind unterschiedlich Bindung Kernfaktoren sind Berechnungsverfahren die spezifische TF (s), dessen vorherzusagen verwendet Bindung sie beeinflussen könnten. Wir bevorzugen CIS-BP 23,24, RegulomeDB 25, UNIProbe 26 zu verwenden, und JASPAR 27. Sobald Kandidaten TFs identifiziert werden, können diese Prognosen ausdrücklich Antikörper gegen diese TFs (EMSA-supershifts und DAPA-Westerns) getestet werden. Eine EMSA-Supershift beinhaltet die Zugabe eines TF-spezifischen Antikörpers an das Kern Lysat und Oligo. Ein positives Ergebnis in einem EMSA-Supershift ist represented als eine weitere Verschiebung der EMSA Band oder einem Verlust des Bandes (in Bezug 28 überprüft). In der komplementären DAPA, eine 5'-biotinylierten Oligokomplexes die Variante und das 20 Basenpaar-flankierenden Nukleotide enthalten, werden mit nuklearen Lysat von relevanten Zelltyp (en) inkubiert alle Kernfaktoren speziell zur Erfassung der Oligos binden. Die Oligokomplexes-nuclear factor-Komplex wird durch Streptavidin-Mikrokügelchen in einer magnetischen Säule immobilisiert. Die gebundenen Kernfaktoren werden direkt durch Elution 29,48 gesammelt. Bindungsvorhersagen können dann durch Western-Blot untersucht werden, um Antikörper, spezifisch für das Protein verwendet wird. In Fällen, in denen es keine offensichtlichen Prognosen oder zu viele Vorhersagen, die Elutionen von Variante Pull-downs der DAPA Experimente können zu einem Proteomik Kern gesendet werden, um Kandidaten TFs mit Massenspektrometrie identifizieren, die anschließend validiert werden können mit diesen zuvor beschriebenen Methoden.
In dem Rest des article wird das detaillierte Protokoll für EMSA und DAPA Analyse genetischer Varianten vorgesehen.
Protocol
Representative Results
Discussion
Obwohl Fortschritte in der Sequenzierung und Genotypisierung Technologien stark unsere Fähigkeit, mit der Krankheit genetische Varianten assoziiert zu identifizieren verbessert haben, unsere Fähigkeit, die Funktionsmechanismen von diesen Varianten betroffen zu verstehen, hinkt. Eine wichtige Ursache des Problems ist, dass viele krankheitsassoziierten Varianten in n angeordnet sind on-kodierenden Regionen des Genoms, die wahrscheinlich Einfluss auf schwieriger zu sagen voraus Mechanismen Genexpression steuern. Hi…
Disclosures
The authors have nothing to disclose.
Acknowledgements
We thank Erin Zoller, Jessica Bene, and Lindsey Hays for input and direction in protocol development. MTW was supported in part by NIH R21 HG008186 and a Trustee Award grant from the Cincinnati Children’s Hospital Research Foundation. ZHP was supported in part by T32 GM063483-13.
Materials
| Custom DNA Oligonucleotides | Integrated DNA Technologies | http://www.idtdna.com/site/order/oligoentry | |
| Potassium Chloride | Fisher Scientific | BP366-500 | KCl, for CE buffer |
| HEPES (1M) | Fisher Scientific | 15630-080 | For CE and NE buffer |
| EDTA (0.5M), pH 8.0 | Life Technologies | R1021 | For CE, NE, and annealing buffer |
| Sodium Chloride | Fisher Scientific | BP358-1 | NaCl, for NE buffer |
| Tris-HCl (1M), pH 8.0 | Invitrogen | BP1756-100 | For annealing buffer |
| Phosphate Buffered Saline (1X) | Fisher Scientific | MT21040CM | PBS, for cell wash |
| DL-Dithiothreitol solution (1M) | Sigma | 646563 | Reducing agent |
| PMSF | Thermo Scientific | 36978 | Protease Inhibitor |
| Phosphatase Inhibitor Cocktail | Thermo Scientific | 78420 | Prevents dephosphorylation of TFs |
| Nonidet P-40 Substitute | IBI Scientific | IB01140 | NP-40, for nuclear extraction |
| BCA Protein Assay Kit | Thermo Scientific | 23225 | For measuring protein concentration |
| Odyssey EMSA Buffer Kit | Licor | 829-07910 | Contains all necessary EMSA buffers |
| TBE Gels, 6%, 12 Wells | Invitrogen | EC6265BOX | For EMSA |
| TBE Buffer (10X) | Thermo Scientific | B52 | For EMSA |
| FactorFinder Starting Kit | Miltenyi Biotec | 130-092-318 | Contains all necessary DAPA buffers |
| Licor Odyssey CLx | Licor | Recommended scanner for DAPA/EMSA | |
| Antibiotic-Antimycotic | Gibco | 15240-062 | Contains 10,000 units/mL of penicillin, 10,000 µg/mL of streptomycin, and 25 µg/mL of Fungizone® Antimycotic |
| Fetal Bovine Serum | Gibco | 26140-079 | FBS, for culture media |
| RPMI 1640 Medium | Gibco | 22400-071 | Contains L-glutamine and 25mM HEPES |
References
- Hindorff, L. A., et al. Potential etiologic and functional implications of genome-wide association loci for human diseases and traits. Proc Natl Acad Sci U S A. 106 (23), 9362-9367 (2009).
- Maurano, M. T., et al. Systematic localization of common disease-associated variation in regulatory DNA. Science. 337 (6099), 1190-1195 (2012).
- Ward, L. D., Kellis, M. Interpreting noncoding genetic variation in complex traits and human disease. Nat Biotechnol. 30 (11), 1095-1106 (2012).
- Paul, D. S., Soranzo, N., Beck, S. Functional interpretation of non-coding sequence variation: concepts and challenges. Bioessays. 36 (2), 191-199 (2014).
- Zhang, F., Lupski, J. R. Non-coding genetic variants in human disease. Hum Mol Genet. , (2015).
- Lee, T. I., Young, R. A. Transcriptional regulation and its misregulation in disease. Cell. 152 (6), 1237-1251 (2013).
- Slatkin, M. Linkage disequilibrium–understanding the evolutionary past and mapping the medical future. Nat Rev Genet. 9 (6), 477-485 (2008).
- Bush, W. S., Moore, J. H. Chapter 11: Genome-wide association studies. PLoS Comput Biol. 8 (12), e1002822 (2012).
- 1000 Genomes Project Consortium. An integrated map of genetic variation from 1,092 human genomes. Nature. 491 (7422), 56-65 (2012).
- Chang, C. C., et al. Second-generation PLINK: rising to the challenge of larger and richer datasets. Gigascience. 4, 7 (2015).
- Purcell, S., et al. PLINK: a tool set for whole-genome association and population-based linkage analyses. Am J Hum Genet. 81 (3), 559-575 (2007).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Crawford, G. E., et al. Genome-wide mapping of DNase hypersensitive sites using massively parallel signature sequencing (MPSS). Genome Res. 16 (1), 123-131 (2006).
- Buenrostro, J. D., Giresi, P. G., Zaba, L. C., Chang, H. Y., Greenleaf, W. J. Transposition of native chromatin for fast and sensitive epigenomic profiling of open chromatin, DNA-binding proteins and nucleosome position. Nat Methods. 10 (12), 1213-1218 (2013).
- Giresi, P. G., Kim, J., McDaniell, R. M., Iyer, V. R., Lieb, J. D. FAIRE Formaldehyde-Assisted Isolation of Regulatory Elements) isolates active regulatory elements from human chromatin. Genome Res. 17 (6), 877-885 (2007).
- Kent, W. J., et al. The human genome browser at UCSC. Genome Res. 12 (6), 996-1006 (2002).
- Roadmap Epigenomics Consortium. Integrative analysis of 111 reference human epigenomes. Nature. 518 (7539), 317-330 (2015).
- Martens, J. H., Stunnenberg, H. G. BLUEPRINT: mapping human blood cell epigenomes. Haematologica. 98 (10), 1487-1489 (2013).
- Liu, T., et al. Cistrome: an integrative platform for transcriptional regulation studies. Genome Biol. 12 (8), R83 (2011).
- Griffon, A., et al. Integrative analysis of public ChIP-seq experiments reveals a complex multi-cell regulatory landscape. Nucleic Acids Res. 43 (4), e27 (2015).
- Staudt, L. M., et al. A lymphoid-specific protein binding to the octamer motif of immunoglobulin genes. Nature. 323 (6089), 640-643 (1986).
- Singh, H., Sen, R., Baltimore, D., Sharp, P. A. A nuclear factor that binds to a conserved sequence motif in transcriptional control elements of immunoglobulin genes. Nature. 319 (6049), 154-158 (1986).
- Weirauch, M. T., et al. Determination and inference of eukaryotic transcription factor sequence specificity. Cell. 158 (6), 1431-1443 (2014).
- Ward, L. D., Kellis, M. HaploReg: a resource for exploring chromatin states, conservation, and regulatory motif alterations within sets of genetically linked variants. Nucleic Acids Res. 40 (Database issue), D930-D934 (2012).
- Boyle, A. P., et al. Annotation of functional variation in personal genomes using RegulomeDB. Genome Res. 22 (9), 1790-1797 (2012).
- Hume, M. A., Barrera, L. A., Gisselbrecht, S. S., Bulyk, M. L. UniPROBE, update 2015: new tools and content for the online database of protein-binding microarray data on protein-DNA interactions. Nucleic Acids Res. 43 (Database issue), D117-D122 (2015).
- Mathelier, A., et al. JASPAR 2014: an extensively expanded and updated open-access database of transcription factor binding profiles. Nucleic Acids Res. 42 (Database issue), 142-147 (2014).
- Smith, M. F., Delbary-Gossart, S. Electrophoretic Mobility Shift Assay (EMSA). Methods Mol Med. 50, 249-257 (2001).
- Franza, B. R., Josephs, S. F., Gilman, M. Z., Ryan, W., Clarkson, B. Characterization of cellular proteins recognizing the HIV enhancer using a microscale DNA-affinity precipitation assay. Nature. 330 (6146), 391-395 (1987).
- . BCA Protein Assay Kit: User Guide Available from: https://tools.thermofisher.com/content/sfs/manuals/MAN0011430_Pierce_BCA_Protein_Asy_UG.pdf (2014)
- Wijeratne, A. B., et al. Phosphopeptide separation using radially aligned titania nanotubes on titanium wire. ACS Appl Mater Interfaces. 7 (21), 11155-11164 (2015).
- Silva, J. M., McMahon, M. The Fastest Western in Town: A Contemporary Twist on the Classic Western Blot Analysis. J. Vis. Exp. (84), (2014).
- Lu, X., et al. Lupus Risk Variant Increases pSTAT1 Binding and Decreases ETS1 Expression. Am J Hum Genet. 96 (5), 731-739 (2015).
- Ramana, C. V., Chatterjee-Kishore, M., Nguyen, H., Stark, G. R. Complex roles of Stat1 in regulating gene expression. Oncogene. 19 (21), 2619-2627 (2000).
- Fillebeen, C., Wilkinson, N., Pantopoulos, K. Electrophoretic Mobility Shift Assay (EMSA) for the Study of RNA-Protein Interactions: The IRE/IRP Example. J. Vis. Exp. (94), e52230 (2014).
- Heng, T. S., Painter, M. W. Immunological Genome Project, C. The Immunological Genome Project: networks of gene expression in immune cells. Nat Immunol. 9 (10), 1091-1094 (2008).
- Wu, C., et al. BioGPS: an extensible and customizable portal for querying and organizing gene annotation resources. Genome Biol. 10 (11), R130 (2009).
- Wu, C., Macleod, I., Su, A. I. BioGPS and MyGene.info: organizing online, gene-centric information. Nucleic Acids Res. 41 (Database issue), D561-D565 (2013).
- Wang, J., et al. Sequence features and chromatin structure around the genomic regions bound by 119 human transcription factors. Genome Res. 22 (9), 1798-1812 (2012).
- Holden, N. S., Tacon, C. E. Principles and problems of the electrophoretic mobility shift assay. J Pharmacol Toxicol Methods. 63 (1), 7-14 (2011).
- Xu, J., Liu, H., Park, J. S., Lan, Y., Jiang, R. Osr1 acts downstream of and interacts synergistically with Six2 to maintain nephron progenitor cells during kidney organogenesis. Development. 141 (7), 1442-1452 (2014).
- Yang, T. -. P., et al. Genevar: a database and Java application for the analysis and visualization of SNP-gene associations in eQTL studies. Bioinformatics. 26 (19), 2474-2476 (2010).
- Fort, A., et al. A liver enhancer in the fibrinogen gene cluster. Blood. 117 (1), 276-282 (2011).
- Solberg, N., Krauss, S. Luciferase assay to study the activity of a cloned promoter DNA fragment. Methods Mol Biol. 977, 65-78 (2013).
- Rahman, M., et al. A repressor element in the 5′-untranslated region of human Pax5 exon 1A. Gene. 263 (1-2), 59-66 (2001).
- Mali, P., et al. RNA-Guided Human Genome Engineering via Cas9. Science. 339 (6121), 823-826 (2013).

