Screening för funktionell icke-kodande genetiska varianter Använda Elektro Mobility Shift Assay (EMSA) och DNA-affinitet Nederbörd analys (DAPA)
Summary
We present a strategic plan and protocol for identifying non-coding genetic variants affecting transcription factor (TF) DNA binding. A detailed experimental protocol is provided for electrophoretic mobility shift assay (EMSA) and DNA affinity precipitation assay (DAPA) analysis of genotype-dependent TF DNA binding.
Abstract
Population and family-based genetic studies typically result in the identification of genetic variants that are statistically associated with a clinical disease or phenotype. For many diseases and traits, most variants are non-coding, and are thus likely to act by impacting subtle, comparatively hard to predict mechanisms controlling gene expression. Here, we describe a general strategic approach to prioritize non-coding variants, and screen them for their function. This approach involves computational prioritization using functional genomic databases followed by experimental analysis of differential binding of transcription factors (TFs) to risk and non-risk alleles. For both electrophoretic mobility shift assay (EMSA) and DNA affinity precipitation assay (DAPA) analysis of genetic variants, a synthetic DNA oligonucleotide (oligo) is used to identify factors in the nuclear lysate of disease or phenotype-relevant cells. For EMSA, the oligonucleotides with or without bound nuclear factors (often TFs) are analyzed by non-denaturing electrophoresis on a tris-borate-EDTA (TBE) polyacrylamide gel. For DAPA, the oligonucleotides are bound to a magnetic column and the nuclear factors that specifically bind the DNA sequence are eluted and analyzed through mass spectrometry or with a reducing sodium dodecyl sulfate polyacrylamide gel electrophoresis (SDS-PAGE) followed by Western blot analysis. This general approach can be widely used to study the function of non-coding genetic variants associated with any disease, trait, or phenotype.
Introduction
Sekvensering och genotypning baserade studier, inklusive Genomvid associationsstudier (GWAS), kandidat locus studier och djupsekvense studier har identifierat många genetiska varianter som statistiskt är associerade med en sjukdom, drag eller fenotyp. I motsats till tidigare prognoser, de flesta av dessa varianter (85-93%) är belägna i icke-kodande regioner och ändrar inte aminosyrasekvensen av proteiner 1,2. Tolka funktion av dessa icke-kodande varianter och bestämma de biologiska mekanismerna som förbinder dem till associerad sjukdom, har drag, eller fenotyp visat utmanande 3-6. Vi har utvecklat en övergripande strategi för att identifiera de molekylära mekanismer som länkar varianter till en viktig mellan fenotyp – genuttryck. Denna pipeline är särskilt utformade för att identifiera moduleringen av TF bindning av genetiska varianter. Denna strategi kombinerar beräkningsmetoder och molekylärbiologiska tekniker som syftar till att förutsägabiologiska effekter av kandidat varianter i silico och verifiera dessa förutsägelser empiriskt (Figur 1).
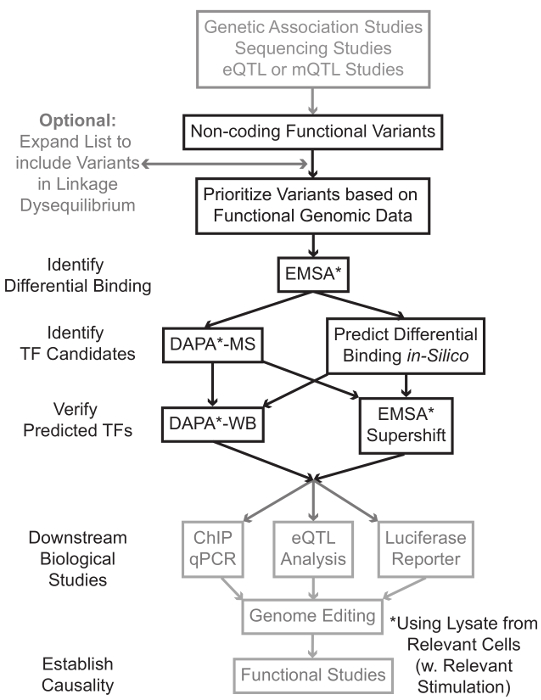
Figur 1:.. Strategi för analys av icke-kodande genetiska varianter Steg som inte ingår i den detaljerade protokoll i samband med detta manuskript är skuggade i grått Klicka här för att se en större version av denna siffra.
I många fall är det viktigt att börja med att utöka listan över varianter att omfatta alla de i hög linkage-ojämvikt (LD) med varje statistiskt samband variant. LD är ett mått på icke-slumpmässigt association av alleler vid två olika kromosomala positioner, som kan mätas av r 2 statistik 7. r 2 är ett mått på linKage obalans mellan två varianter, med en r 2 = 1 betecknar perfekt koppling mellan två varianter. Alleler i hög LD befinns samtidigt segregera på kromosomen över släkt- befolkningar. Nuvarande genotypning arrayer inkluderar inte alla kända varianter i det mänskliga genomet. Istället utnyttjar LD inom det mänskliga genomet och inkludera en delmängd av de kända varianter som fungerar som ombud för andra varianter inom en viss region i LD 8. Således kan en variant utan någon biologisk konsekvens vara associerad med en särskild sjukdom eftersom det är i LD med orsaks variant-varianten med en meningsfull biologisk effekt. Procedurmässigt är det rekommenderat att konvertera den senaste versionen av 1000 genomen projektet 9 variant samtals filer (VCF) till binära filer som är kompatibla med Plink 10,11, ett open-source verktyg för hela genomet associationsanalys. Därefter alla andra genetiska varianter med LD r 2> 0,8 med varje ingång genetisk variant kan identifieras som kandidater. Det är viktigt att använda den tillämpliga referenspopulationen för denna steg t ex om en variant identifierades i frågor av europeisk härkomst, data från patienter med liknande härkomst ska användas för LD expansion.
LD expansionen resulterar ofta i dussintals kandidat varianter, och det är troligt att endast en liten del av dessa bidrar till sjukdomsmekanismen. Ofta är det omöjligt att experimentellt undersöka var och en av dessa varianter individuellt. Det är därför lämpligt att utnyttja de tusentals offentligt tillgängliga funktionsgenomiska datamängder som ett filter för att prioritera varianter. Till exempel har ENCODE konsortiet 12 utfört tusentals Chip-punkter experiment som beskriver bindningen av TF och co-faktorer, och histon märken i en rad olika sammanhang, tillsammans med kromatin tillgänglighetsdata från teknik såsom DNas-punkter 13, ATAC -seq 14, och FAIRE-seq 15. DatabALL och webbservrar som UCSC Genome Browser 16, färdplan Epigenomics 17, Blueprint Epigenome 18, Cistrome 19, och mappa 20 ger fri tillgång till data som produceras av dessa och andra experimentella metoder inom ett brett spektrum av celltyper och villkor. När det finns för många varianter att undersöka experimentellt, kan dessa data användas för att prioritera de som ligger inom troliga reglerande regioner inom relevanta cell- och vävnadstyper. Vidare, i de fall där en variant inom en chip-punkter topp för ett specifikt protein, kan dessa uppgifter ger potentiella kunder som den specifika TF (s) eller co-faktorer vars bindning kan påverka.
Därefter de resulterande prioriterade varianter screenas experimentellt att validera förutspått genotyp beroende proteinbindning med hjälp av EMSA 21,22. EMSA mäter förändringar i migreringen av oligo på en icke-reducerande TBE-gel. Fluorescensmärkt oligo inkuberas mednukleära lysat, och bindning av nukleära faktorer kommer att fördröja rörelsen av oligo på gelén. På detta sätt, oligo som har bunden fler nukleära faktorer kommer att presentera som en starkare fluorescerande signal vid scanning. Noterbart är inte EMSA inte kräva förutsägelser om de specifika proteiner vars bindning kommer att påverkas.
När varianter identifieras som är belägna inom förutsedda regulatoriska regioner och är i stånd att differentiellt bindande kärn faktorer, är beräkningsmetoder som används för att förutsäga specifika TF (s) vars bindande de kan påverka. Vi föredrar att använda CIS-BP 23,24, RegulomeDB 25 UniProbe 26, och Jaspar 27. När kandidat TF har identifierats, kan dessa förutsägelser specifikt testas med hjälp av antikroppar mot dessa TF (EMSA-supershifts och DAPA-Westerns). En EMSA-supershift innebär tillsättning av en TF-specifik antikropp mot den nukleära lysat och oligo. Ett positivt resultat i en EMSA-supershift är represented som en ytterligare förskjutning i EMSA-bandet, eller en förlust av bandet (översikt i referens 28). I den komplementära DAPA är en 5'-biotinylerad oligo duplex innehållande varianten och 20 baspar som flankerar nukleotider inkuberas med kärn lysat från relevant celltyp (s) för att fånga några nukleära faktorer som specifikt binder oligos. Oligo duplex-nukleär faktor komplexet immobiliseras genom streptavidin mikropärlor i en magnetisk kolonn. De bundna nukleära faktorer samlas direkt genom eluering 29,48. Bindande prognoser kan sedan bedömas av en Western blöt med användning av antikroppar som är specifika för proteinet. I de fall där det inte finns några uppenbara förutsägelser, eller alltför många förutsägelser, de elueringar från variantrullgardinsmenyerna av DAPA experiment kan skickas till en proteomik kärna för att identifiera kandidat TF använder masspektrometri, som därefter kan valideras med hjälp av dessa tidigare beskrivna metoder.
I återstoden av article är den detaljerade protokoll för EMSA och DAPA analys av genetiska varianter tillhandahålls.
Protocol
Representative Results
Discussion
Även framsteg inom sekvensering och genotypning teknik har förbättrats avsevärt vår förmåga att identifiera genetiska varianter i samband med sjukdom, är vår förmåga att förstå de funktionella mekanismer påverkas av dessa varianter släpar. En viktig källa till problemet är att många sjukdomsassocierade varianter finns i n på-kodande regioner av genomet, vilket sannolikt påverkar hårdare mot förutse mekanismer som styr genuttryck. Här presenterar vi ett protokoll baserat på EMSA och DAPA tek…
Disclosures
The authors have nothing to disclose.
Acknowledgements
We thank Erin Zoller, Jessica Bene, and Lindsey Hays for input and direction in protocol development. MTW was supported in part by NIH R21 HG008186 and a Trustee Award grant from the Cincinnati Children’s Hospital Research Foundation. ZHP was supported in part by T32 GM063483-13.
Materials
| Custom DNA Oligonucleotides | Integrated DNA Technologies | http://www.idtdna.com/site/order/oligoentry | |
| Potassium Chloride | Fisher Scientific | BP366-500 | KCl, for CE buffer |
| HEPES (1M) | Fisher Scientific | 15630-080 | For CE and NE buffer |
| EDTA (0.5M), pH 8.0 | Life Technologies | R1021 | For CE, NE, and annealing buffer |
| Sodium Chloride | Fisher Scientific | BP358-1 | NaCl, for NE buffer |
| Tris-HCl (1M), pH 8.0 | Invitrogen | BP1756-100 | For annealing buffer |
| Phosphate Buffered Saline (1X) | Fisher Scientific | MT21040CM | PBS, for cell wash |
| DL-Dithiothreitol solution (1M) | Sigma | 646563 | Reducing agent |
| PMSF | Thermo Scientific | 36978 | Protease Inhibitor |
| Phosphatase Inhibitor Cocktail | Thermo Scientific | 78420 | Prevents dephosphorylation of TFs |
| Nonidet P-40 Substitute | IBI Scientific | IB01140 | NP-40, for nuclear extraction |
| BCA Protein Assay Kit | Thermo Scientific | 23225 | For measuring protein concentration |
| Odyssey EMSA Buffer Kit | Licor | 829-07910 | Contains all necessary EMSA buffers |
| TBE Gels, 6%, 12 Wells | Invitrogen | EC6265BOX | For EMSA |
| TBE Buffer (10X) | Thermo Scientific | B52 | For EMSA |
| FactorFinder Starting Kit | Miltenyi Biotec | 130-092-318 | Contains all necessary DAPA buffers |
| Licor Odyssey CLx | Licor | Recommended scanner for DAPA/EMSA | |
| Antibiotic-Antimycotic | Gibco | 15240-062 | Contains 10,000 units/mL of penicillin, 10,000 µg/mL of streptomycin, and 25 µg/mL of Fungizone® Antimycotic |
| Fetal Bovine Serum | Gibco | 26140-079 | FBS, for culture media |
| RPMI 1640 Medium | Gibco | 22400-071 | Contains L-glutamine and 25mM HEPES |
References
- Hindorff, L. A., et al. Potential etiologic and functional implications of genome-wide association loci for human diseases and traits. Proc Natl Acad Sci U S A. 106 (23), 9362-9367 (2009).
- Maurano, M. T., et al. Systematic localization of common disease-associated variation in regulatory DNA. Science. 337 (6099), 1190-1195 (2012).
- Ward, L. D., Kellis, M. Interpreting noncoding genetic variation in complex traits and human disease. Nat Biotechnol. 30 (11), 1095-1106 (2012).
- Paul, D. S., Soranzo, N., Beck, S. Functional interpretation of non-coding sequence variation: concepts and challenges. Bioessays. 36 (2), 191-199 (2014).
- Zhang, F., Lupski, J. R. Non-coding genetic variants in human disease. Hum Mol Genet. , (2015).
- Lee, T. I., Young, R. A. Transcriptional regulation and its misregulation in disease. Cell. 152 (6), 1237-1251 (2013).
- Slatkin, M. Linkage disequilibrium–understanding the evolutionary past and mapping the medical future. Nat Rev Genet. 9 (6), 477-485 (2008).
- Bush, W. S., Moore, J. H. Chapter 11: Genome-wide association studies. PLoS Comput Biol. 8 (12), e1002822 (2012).
- 1000 Genomes Project Consortium. An integrated map of genetic variation from 1,092 human genomes. Nature. 491 (7422), 56-65 (2012).
- Chang, C. C., et al. Second-generation PLINK: rising to the challenge of larger and richer datasets. Gigascience. 4, 7 (2015).
- Purcell, S., et al. PLINK: a tool set for whole-genome association and population-based linkage analyses. Am J Hum Genet. 81 (3), 559-575 (2007).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Crawford, G. E., et al. Genome-wide mapping of DNase hypersensitive sites using massively parallel signature sequencing (MPSS). Genome Res. 16 (1), 123-131 (2006).
- Buenrostro, J. D., Giresi, P. G., Zaba, L. C., Chang, H. Y., Greenleaf, W. J. Transposition of native chromatin for fast and sensitive epigenomic profiling of open chromatin, DNA-binding proteins and nucleosome position. Nat Methods. 10 (12), 1213-1218 (2013).
- Giresi, P. G., Kim, J., McDaniell, R. M., Iyer, V. R., Lieb, J. D. FAIRE Formaldehyde-Assisted Isolation of Regulatory Elements) isolates active regulatory elements from human chromatin. Genome Res. 17 (6), 877-885 (2007).
- Kent, W. J., et al. The human genome browser at UCSC. Genome Res. 12 (6), 996-1006 (2002).
- Roadmap Epigenomics Consortium. Integrative analysis of 111 reference human epigenomes. Nature. 518 (7539), 317-330 (2015).
- Martens, J. H., Stunnenberg, H. G. BLUEPRINT: mapping human blood cell epigenomes. Haematologica. 98 (10), 1487-1489 (2013).
- Liu, T., et al. Cistrome: an integrative platform for transcriptional regulation studies. Genome Biol. 12 (8), R83 (2011).
- Griffon, A., et al. Integrative analysis of public ChIP-seq experiments reveals a complex multi-cell regulatory landscape. Nucleic Acids Res. 43 (4), e27 (2015).
- Staudt, L. M., et al. A lymphoid-specific protein binding to the octamer motif of immunoglobulin genes. Nature. 323 (6089), 640-643 (1986).
- Singh, H., Sen, R., Baltimore, D., Sharp, P. A. A nuclear factor that binds to a conserved sequence motif in transcriptional control elements of immunoglobulin genes. Nature. 319 (6049), 154-158 (1986).
- Weirauch, M. T., et al. Determination and inference of eukaryotic transcription factor sequence specificity. Cell. 158 (6), 1431-1443 (2014).
- Ward, L. D., Kellis, M. HaploReg: a resource for exploring chromatin states, conservation, and regulatory motif alterations within sets of genetically linked variants. Nucleic Acids Res. 40 (Database issue), D930-D934 (2012).
- Boyle, A. P., et al. Annotation of functional variation in personal genomes using RegulomeDB. Genome Res. 22 (9), 1790-1797 (2012).
- Hume, M. A., Barrera, L. A., Gisselbrecht, S. S., Bulyk, M. L. UniPROBE, update 2015: new tools and content for the online database of protein-binding microarray data on protein-DNA interactions. Nucleic Acids Res. 43 (Database issue), D117-D122 (2015).
- Mathelier, A., et al. JASPAR 2014: an extensively expanded and updated open-access database of transcription factor binding profiles. Nucleic Acids Res. 42 (Database issue), 142-147 (2014).
- Smith, M. F., Delbary-Gossart, S. Electrophoretic Mobility Shift Assay (EMSA). Methods Mol Med. 50, 249-257 (2001).
- Franza, B. R., Josephs, S. F., Gilman, M. Z., Ryan, W., Clarkson, B. Characterization of cellular proteins recognizing the HIV enhancer using a microscale DNA-affinity precipitation assay. Nature. 330 (6146), 391-395 (1987).
- . BCA Protein Assay Kit: User Guide Available from: https://tools.thermofisher.com/content/sfs/manuals/MAN0011430_Pierce_BCA_Protein_Asy_UG.pdf (2014)
- Wijeratne, A. B., et al. Phosphopeptide separation using radially aligned titania nanotubes on titanium wire. ACS Appl Mater Interfaces. 7 (21), 11155-11164 (2015).
- Silva, J. M., McMahon, M. The Fastest Western in Town: A Contemporary Twist on the Classic Western Blot Analysis. J. Vis. Exp. (84), (2014).
- Lu, X., et al. Lupus Risk Variant Increases pSTAT1 Binding and Decreases ETS1 Expression. Am J Hum Genet. 96 (5), 731-739 (2015).
- Ramana, C. V., Chatterjee-Kishore, M., Nguyen, H., Stark, G. R. Complex roles of Stat1 in regulating gene expression. Oncogene. 19 (21), 2619-2627 (2000).
- Fillebeen, C., Wilkinson, N., Pantopoulos, K. Electrophoretic Mobility Shift Assay (EMSA) for the Study of RNA-Protein Interactions: The IRE/IRP Example. J. Vis. Exp. (94), e52230 (2014).
- Heng, T. S., Painter, M. W. Immunological Genome Project, C. The Immunological Genome Project: networks of gene expression in immune cells. Nat Immunol. 9 (10), 1091-1094 (2008).
- Wu, C., et al. BioGPS: an extensible and customizable portal for querying and organizing gene annotation resources. Genome Biol. 10 (11), R130 (2009).
- Wu, C., Macleod, I., Su, A. I. BioGPS and MyGene.info: organizing online, gene-centric information. Nucleic Acids Res. 41 (Database issue), D561-D565 (2013).
- Wang, J., et al. Sequence features and chromatin structure around the genomic regions bound by 119 human transcription factors. Genome Res. 22 (9), 1798-1812 (2012).
- Holden, N. S., Tacon, C. E. Principles and problems of the electrophoretic mobility shift assay. J Pharmacol Toxicol Methods. 63 (1), 7-14 (2011).
- Xu, J., Liu, H., Park, J. S., Lan, Y., Jiang, R. Osr1 acts downstream of and interacts synergistically with Six2 to maintain nephron progenitor cells during kidney organogenesis. Development. 141 (7), 1442-1452 (2014).
- Yang, T. -. P., et al. Genevar: a database and Java application for the analysis and visualization of SNP-gene associations in eQTL studies. Bioinformatics. 26 (19), 2474-2476 (2010).
- Fort, A., et al. A liver enhancer in the fibrinogen gene cluster. Blood. 117 (1), 276-282 (2011).
- Solberg, N., Krauss, S. Luciferase assay to study the activity of a cloned promoter DNA fragment. Methods Mol Biol. 977, 65-78 (2013).
- Rahman, M., et al. A repressor element in the 5′-untranslated region of human Pax5 exon 1A. Gene. 263 (1-2), 59-66 (2001).
- Mali, P., et al. RNA-Guided Human Genome Engineering via Cas9. Science. 339 (6121), 823-826 (2013).

