Elektroforetik Hareketlilik Assay (EMSA) ve Shift kullanarak Fonksiyonel Olmayan kodlama Genetik Türevleri için tarama DNA afinite Yağış Deneyi (DAPA)
Summary
We present a strategic plan and protocol for identifying non-coding genetic variants affecting transcription factor (TF) DNA binding. A detailed experimental protocol is provided for electrophoretic mobility shift assay (EMSA) and DNA affinity precipitation assay (DAPA) analysis of genotype-dependent TF DNA binding.
Abstract
Population and family-based genetic studies typically result in the identification of genetic variants that are statistically associated with a clinical disease or phenotype. For many diseases and traits, most variants are non-coding, and are thus likely to act by impacting subtle, comparatively hard to predict mechanisms controlling gene expression. Here, we describe a general strategic approach to prioritize non-coding variants, and screen them for their function. This approach involves computational prioritization using functional genomic databases followed by experimental analysis of differential binding of transcription factors (TFs) to risk and non-risk alleles. For both electrophoretic mobility shift assay (EMSA) and DNA affinity precipitation assay (DAPA) analysis of genetic variants, a synthetic DNA oligonucleotide (oligo) is used to identify factors in the nuclear lysate of disease or phenotype-relevant cells. For EMSA, the oligonucleotides with or without bound nuclear factors (often TFs) are analyzed by non-denaturing electrophoresis on a tris-borate-EDTA (TBE) polyacrylamide gel. For DAPA, the oligonucleotides are bound to a magnetic column and the nuclear factors that specifically bind the DNA sequence are eluted and analyzed through mass spectrometry or with a reducing sodium dodecyl sulfate polyacrylamide gel electrophoresis (SDS-PAGE) followed by Western blot analysis. This general approach can be widely used to study the function of non-coding genetic variants associated with any disease, trait, or phenotype.
Introduction
Sıralama ve genom çapında ilişkilendirme çalışmaları (GWAS), aday odağı çalışmaları da dahil olmak üzere genotiplendirme dayalı çalışmalar, ve derin sıralama çalışmaları, istatistiksel bir hastalık, özellik veya fenotip ile ilişkili birçok genetik varyantlar belirledik. Erken tahminler aksine, bu varyantlar (85-93%) en kodlayıcı olmayan bölgelerinde bulunur ve protein 1,2 amino asit dizisini değiştirmezler. Bu kodlayıcı olmayan varyantları işlevini yorumlama ve ilişkili hastalık onları bağlayan biyolojik mekanizmaları belirleyen özellik veya fenotip 3-6 zorlu kanıtlanmıştır. gen ifadesinin – Biz önemli bir ara fenotip için varyantları bağlantı moleküler mekanizmaları tanımlamak için genel bir strateji geliştirdik. Bu boru hattı, özellikle genetik varyantlar bağlama TF modülasyonu belirlemek için tasarlanmıştır. Bu strateji tahmin etmek amaçlanmıştır hesaplama yaklaşımları ve moleküler biyoloji teknikleri birleştirenBiyolojik siliko aday varyantları etkileri ve doğrulamak için şu tahminler ampirik (Şekil 1).
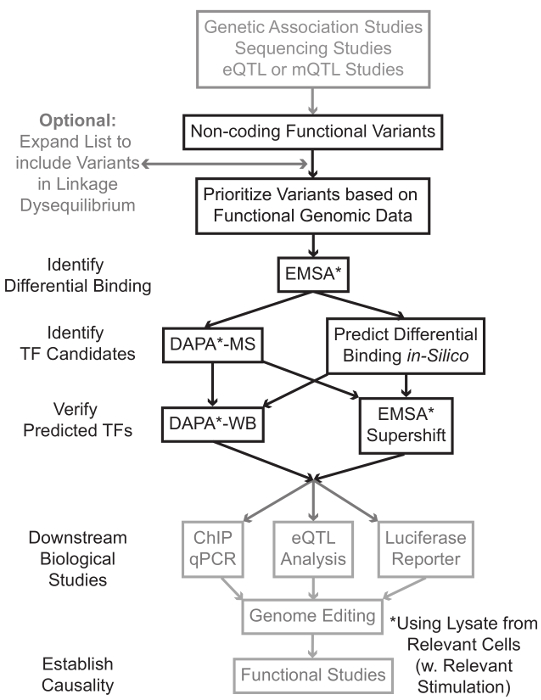
Şekil 1:.. Gri gölgeli bu yazının ilişkili detaylı protokolde yer almayan kodlayıcı olmayan genetik varyantları Adımlar analizi için stratejik yaklaşım bu rakamın daha büyük bir versiyonunu görmek için lütfen buraya tıklayınız.
Bir çok durumda, her bir istatistiksel bağlantılı varyantı ile, yüksek bir bağlantı-dengesizliği (LD) tüm bu dahil varyantların listesini genişleterek başlaması önemlidir. LD R2 istatistik 7 ile ölçülebilir iki farklı kromozom konumlarda allel rastgele olmayan federasyonu, bir ölçüsüdür. R2 lin bir ölçüsüdüriki modeli arasında bir r 2 = 1 belirten mükemmel bağlantı ile iki modeli arasındaki kage dengesizlik. yüksek LD alleller atalarının popülasyonlar arasında kromozom üzerindeki ortak ayırmak bulunmuştur. Güncel genotiplendirme dizileri insan genomu bilinen tüm varyasyonları dahil değildir. Bunun yerine, onlar insan genomunun içinde LD istismar ve LD 8 belirli bir bölgedeki diğer varyantları için temsilci olarak vekaleten oy bilinen çeşitleri bir alt kümesini içerir. anlamlı bir biyolojik etki ile nedensel varyantı-varyantı ile LD çünkü Böylece herhangi bir biyolojik sonucu olmayan bir varyant, belirli bir hastalık ile ilişkili olabilir. Prosedürel, 1,000 genomları son sürümü plink 10,11, tüm genom dernek analizi için bir açık kaynak aracı ile uyumlu ikili dosyaları içine 9 varyant çağrı dosyalarını (vcf) proje dönüştürmek için tavsiye edilir. Daha sonra, her giriş genetik va ile LD r 2> 0.8 ile diğer tüm genetik varyantlarRiant adayları olarak tespit edilebilir. Bir varyant Avrupa kökenli kişilerde, benzer kökenli konularda veri LD genişlemesi için kullanılması gerektiğini tespit edilmiştir eğer, bu üvey örneğin için uygun referans nüfusu kullanmak önemlidir.
LD genişletme genellikle aday türevleri onlarca neden olur ve sadece küçük bir fraksiyonu bir hastalık mekanizmasına katkıda bulunması olasıdır. Genellikle, deneysel bireysel bu varyantların her incelemek için olanaksız olduğunu. Varyantlar öncelik filtre olarak kamuya açık fonksiyonel genomik veri setleri binlerce kaldıraç nedenle faydalıdır. Örneğin, şifreleme konsorsiyum 12, örneğin, DNase seq 13 ATAC olarak teknolojilerden kromatin erişilebilirlik verileri ile birlikte, bağlamlarda çeşitli yonga-DİZ TFs bağlanmasını tarif eden deneyler ve ko-faktörler, ve histon işaretleri binlerce yürüttü -DİZ 14 ve FAIRE seq 15. databBöyle UCSC Genom Tarayıcı 16, Yol Haritası Epigenomics 17, Blueprint epigenom 18 Cistrome 19 ve eşleştirmek 20 olarak ases ve web sunucuları hücre tipleri ve koşullar geniş bir yelpazede bu ve diğer deneysel tekniklerle üretilen verilere ücretsiz erişim sağlar. Deneysel olarak incelemek için pek çok çeşidi vardır, bu veri, ilgili hücre ve doku tiplerinde olası düzenleyici bölgeler içinde yer alan bu öncelik için kullanılabilir. Bundan başka, bir varyantı belirli bir protein için bir çip DİZİ zirve içinde olduğu durumlarda, bu veriler, belirli bir TF (ler) olan ya da etki olabilir bağlama ko-faktör olarak potansiyel neden sağlayabilir.
Sonraki, öncelik ortaya çıkan varyantları EMSA 21,22 kullanarak bağlama tahmin genotip-bağımlı protein doğrulamak için deneysel taranmaktadır. EMSA bir indirgeyici olmayan TBE jeli üzerinde oligo geçiş değişimi ölçer. Floresan etiketli oligo kuluçkalanırNükleer lizat ve nükleer faktörlerin bağlayıcı jel üzerinde oligo hareketini geciktirir. Bu şekilde, tarama sırasında daha güçlü bir floresan sinyal olarak sunacak daha fazla nükleer faktörler bağlı olan oligo içinde. Özellikle, EMSA bağlayıcı etkilenir spesifik protein ilgili tahminlerde gerektirmez.
varyantlar tahmin düzenleyici bölgeler içinde yer alan ve farklı şekilde bağlayıcı nükleer faktörlerin yeteneğine sahip olduğu tespit edildikten sonra, hesaplama yöntemleri olan onlar etkileyebilecek bağlayıcı belirli TF (ler) tahmin etmek istihdam edilmektedir. Biz BDT-BP 23,24, RegulomeDB 25, UniProbe 26 ve Jaspar 27 kullanmayı tercih eder. Aday TF'ler belirlendikten sonra, bu öngörüler özellikle bu TFs (EMSA-Süper kaymalar ve DAPA-Batılıların) karşı antikorlar kullanılarak test edilebilir. Bir EMSA-süperkayma nükleer lizat ve oligo bir TF-özgü antikorun eklenmesini kapsamaktadır. Bir EMSA-süperkayma bir pozitif sonuç repr olduğunuEMSA bandında bir kayma ya da grubun bir kayıp olarak esented (referans 28'de incelenmiştir). Tamamlayıcı DAPA, varyant ve yan nükleotidlerini 20 baz çifti ihtiva eden bir 5'-biyotinlenmiş oligo dupleks spesifik oligolar bağlama nükleer faktörü yakalamak için uygun bir hücre tipinde (ler) den, nükleer lizatı ile inkübe edilir. Oligo dubleks nükleer faktör kompleksi manyetik sütunda mikroboncuklarm streptavidin tesbit edilir. Bağlı nükleer faktörler elüsyon 29,48 doğrudan tahsil edilir. Bağlanma tahminler daha sonra proteine özel antikorlar kullanılarak bir Western blot ile tespit edilebilir. sonradan kullanılarak valide edilebilir hiçbir belirgin tahminler, ya da çok fazla tahminler, kütle-spektrometresi kullanılarak aday TFs belirlemek için bir proteomik çekirdeğe gönderilebilir DAPA deneyleri varyant aşağı açılır menüden elüsyon olduğu durumlarda, bu daha önce açıklanan yöntemleri.
articl kalanındae, genetik varyantları EMSA ve DAPA analizi için detaylı bir protokol sağlanır.
Protocol
Representative Results
Discussion
sıralama ve genotiplendirme teknolojilerindeki gelişmeler büyük ölçüde hastalıkla ilgili genetik varyantları tanımlamak için kapasitemizi gelişmiş olmasına rağmen, bu varyantlar etkilenen fonksiyonel mekanizmaları anlamak için yeteneğimizi kalıyor. Sorunun ana kaynağı olasılıkla gen ekspresyonunu kontrol zor-arası tahmin mekanizmaları etkileyen ilgili kodlama genomunun bölgeler birçok hastalıkla ilişkili varyantları n yer olmasıdır. Burada, biz büyük olasılıkla çok kodlayıcı …
Disclosures
The authors have nothing to disclose.
Acknowledgements
We thank Erin Zoller, Jessica Bene, and Lindsey Hays for input and direction in protocol development. MTW was supported in part by NIH R21 HG008186 and a Trustee Award grant from the Cincinnati Children’s Hospital Research Foundation. ZHP was supported in part by T32 GM063483-13.
Materials
| Custom DNA Oligonucleotides | Integrated DNA Technologies | http://www.idtdna.com/site/order/oligoentry | |
| Potassium Chloride | Fisher Scientific | BP366-500 | KCl, for CE buffer |
| HEPES (1M) | Fisher Scientific | 15630-080 | For CE and NE buffer |
| EDTA (0.5M), pH 8.0 | Life Technologies | R1021 | For CE, NE, and annealing buffer |
| Sodium Chloride | Fisher Scientific | BP358-1 | NaCl, for NE buffer |
| Tris-HCl (1M), pH 8.0 | Invitrogen | BP1756-100 | For annealing buffer |
| Phosphate Buffered Saline (1X) | Fisher Scientific | MT21040CM | PBS, for cell wash |
| DL-Dithiothreitol solution (1M) | Sigma | 646563 | Reducing agent |
| PMSF | Thermo Scientific | 36978 | Protease Inhibitor |
| Phosphatase Inhibitor Cocktail | Thermo Scientific | 78420 | Prevents dephosphorylation of TFs |
| Nonidet P-40 Substitute | IBI Scientific | IB01140 | NP-40, for nuclear extraction |
| BCA Protein Assay Kit | Thermo Scientific | 23225 | For measuring protein concentration |
| Odyssey EMSA Buffer Kit | Licor | 829-07910 | Contains all necessary EMSA buffers |
| TBE Gels, 6%, 12 Wells | Invitrogen | EC6265BOX | For EMSA |
| TBE Buffer (10X) | Thermo Scientific | B52 | For EMSA |
| FactorFinder Starting Kit | Miltenyi Biotec | 130-092-318 | Contains all necessary DAPA buffers |
| Licor Odyssey CLx | Licor | Recommended scanner for DAPA/EMSA | |
| Antibiotic-Antimycotic | Gibco | 15240-062 | Contains 10,000 units/mL of penicillin, 10,000 µg/mL of streptomycin, and 25 µg/mL of Fungizone® Antimycotic |
| Fetal Bovine Serum | Gibco | 26140-079 | FBS, for culture media |
| RPMI 1640 Medium | Gibco | 22400-071 | Contains L-glutamine and 25mM HEPES |
References
- Hindorff, L. A., et al. Potential etiologic and functional implications of genome-wide association loci for human diseases and traits. Proc Natl Acad Sci U S A. 106 (23), 9362-9367 (2009).
- Maurano, M. T., et al. Systematic localization of common disease-associated variation in regulatory DNA. Science. 337 (6099), 1190-1195 (2012).
- Ward, L. D., Kellis, M. Interpreting noncoding genetic variation in complex traits and human disease. Nat Biotechnol. 30 (11), 1095-1106 (2012).
- Paul, D. S., Soranzo, N., Beck, S. Functional interpretation of non-coding sequence variation: concepts and challenges. Bioessays. 36 (2), 191-199 (2014).
- Zhang, F., Lupski, J. R. Non-coding genetic variants in human disease. Hum Mol Genet. , (2015).
- Lee, T. I., Young, R. A. Transcriptional regulation and its misregulation in disease. Cell. 152 (6), 1237-1251 (2013).
- Slatkin, M. Linkage disequilibrium–understanding the evolutionary past and mapping the medical future. Nat Rev Genet. 9 (6), 477-485 (2008).
- Bush, W. S., Moore, J. H. Chapter 11: Genome-wide association studies. PLoS Comput Biol. 8 (12), e1002822 (2012).
- 1000 Genomes Project Consortium. An integrated map of genetic variation from 1,092 human genomes. Nature. 491 (7422), 56-65 (2012).
- Chang, C. C., et al. Second-generation PLINK: rising to the challenge of larger and richer datasets. Gigascience. 4, 7 (2015).
- Purcell, S., et al. PLINK: a tool set for whole-genome association and population-based linkage analyses. Am J Hum Genet. 81 (3), 559-575 (2007).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Crawford, G. E., et al. Genome-wide mapping of DNase hypersensitive sites using massively parallel signature sequencing (MPSS). Genome Res. 16 (1), 123-131 (2006).
- Buenrostro, J. D., Giresi, P. G., Zaba, L. C., Chang, H. Y., Greenleaf, W. J. Transposition of native chromatin for fast and sensitive epigenomic profiling of open chromatin, DNA-binding proteins and nucleosome position. Nat Methods. 10 (12), 1213-1218 (2013).
- Giresi, P. G., Kim, J., McDaniell, R. M., Iyer, V. R., Lieb, J. D. FAIRE Formaldehyde-Assisted Isolation of Regulatory Elements) isolates active regulatory elements from human chromatin. Genome Res. 17 (6), 877-885 (2007).
- Kent, W. J., et al. The human genome browser at UCSC. Genome Res. 12 (6), 996-1006 (2002).
- Roadmap Epigenomics Consortium. Integrative analysis of 111 reference human epigenomes. Nature. 518 (7539), 317-330 (2015).
- Martens, J. H., Stunnenberg, H. G. BLUEPRINT: mapping human blood cell epigenomes. Haematologica. 98 (10), 1487-1489 (2013).
- Liu, T., et al. Cistrome: an integrative platform for transcriptional regulation studies. Genome Biol. 12 (8), R83 (2011).
- Griffon, A., et al. Integrative analysis of public ChIP-seq experiments reveals a complex multi-cell regulatory landscape. Nucleic Acids Res. 43 (4), e27 (2015).
- Staudt, L. M., et al. A lymphoid-specific protein binding to the octamer motif of immunoglobulin genes. Nature. 323 (6089), 640-643 (1986).
- Singh, H., Sen, R., Baltimore, D., Sharp, P. A. A nuclear factor that binds to a conserved sequence motif in transcriptional control elements of immunoglobulin genes. Nature. 319 (6049), 154-158 (1986).
- Weirauch, M. T., et al. Determination and inference of eukaryotic transcription factor sequence specificity. Cell. 158 (6), 1431-1443 (2014).
- Ward, L. D., Kellis, M. HaploReg: a resource for exploring chromatin states, conservation, and regulatory motif alterations within sets of genetically linked variants. Nucleic Acids Res. 40 (Database issue), D930-D934 (2012).
- Boyle, A. P., et al. Annotation of functional variation in personal genomes using RegulomeDB. Genome Res. 22 (9), 1790-1797 (2012).
- Hume, M. A., Barrera, L. A., Gisselbrecht, S. S., Bulyk, M. L. UniPROBE, update 2015: new tools and content for the online database of protein-binding microarray data on protein-DNA interactions. Nucleic Acids Res. 43 (Database issue), D117-D122 (2015).
- Mathelier, A., et al. JASPAR 2014: an extensively expanded and updated open-access database of transcription factor binding profiles. Nucleic Acids Res. 42 (Database issue), 142-147 (2014).
- Smith, M. F., Delbary-Gossart, S. Electrophoretic Mobility Shift Assay (EMSA). Methods Mol Med. 50, 249-257 (2001).
- Franza, B. R., Josephs, S. F., Gilman, M. Z., Ryan, W., Clarkson, B. Characterization of cellular proteins recognizing the HIV enhancer using a microscale DNA-affinity precipitation assay. Nature. 330 (6146), 391-395 (1987).
- . BCA Protein Assay Kit: User Guide Available from: https://tools.thermofisher.com/content/sfs/manuals/MAN0011430_Pierce_BCA_Protein_Asy_UG.pdf (2014)
- Wijeratne, A. B., et al. Phosphopeptide separation using radially aligned titania nanotubes on titanium wire. ACS Appl Mater Interfaces. 7 (21), 11155-11164 (2015).
- Silva, J. M., McMahon, M. The Fastest Western in Town: A Contemporary Twist on the Classic Western Blot Analysis. J. Vis. Exp. (84), (2014).
- Lu, X., et al. Lupus Risk Variant Increases pSTAT1 Binding and Decreases ETS1 Expression. Am J Hum Genet. 96 (5), 731-739 (2015).
- Ramana, C. V., Chatterjee-Kishore, M., Nguyen, H., Stark, G. R. Complex roles of Stat1 in regulating gene expression. Oncogene. 19 (21), 2619-2627 (2000).
- Fillebeen, C., Wilkinson, N., Pantopoulos, K. Electrophoretic Mobility Shift Assay (EMSA) for the Study of RNA-Protein Interactions: The IRE/IRP Example. J. Vis. Exp. (94), e52230 (2014).
- Heng, T. S., Painter, M. W. Immunological Genome Project, C. The Immunological Genome Project: networks of gene expression in immune cells. Nat Immunol. 9 (10), 1091-1094 (2008).
- Wu, C., et al. BioGPS: an extensible and customizable portal for querying and organizing gene annotation resources. Genome Biol. 10 (11), R130 (2009).
- Wu, C., Macleod, I., Su, A. I. BioGPS and MyGene.info: organizing online, gene-centric information. Nucleic Acids Res. 41 (Database issue), D561-D565 (2013).
- Wang, J., et al. Sequence features and chromatin structure around the genomic regions bound by 119 human transcription factors. Genome Res. 22 (9), 1798-1812 (2012).
- Holden, N. S., Tacon, C. E. Principles and problems of the electrophoretic mobility shift assay. J Pharmacol Toxicol Methods. 63 (1), 7-14 (2011).
- Xu, J., Liu, H., Park, J. S., Lan, Y., Jiang, R. Osr1 acts downstream of and interacts synergistically with Six2 to maintain nephron progenitor cells during kidney organogenesis. Development. 141 (7), 1442-1452 (2014).
- Yang, T. -. P., et al. Genevar: a database and Java application for the analysis and visualization of SNP-gene associations in eQTL studies. Bioinformatics. 26 (19), 2474-2476 (2010).
- Fort, A., et al. A liver enhancer in the fibrinogen gene cluster. Blood. 117 (1), 276-282 (2011).
- Solberg, N., Krauss, S. Luciferase assay to study the activity of a cloned promoter DNA fragment. Methods Mol Biol. 977, 65-78 (2013).
- Rahman, M., et al. A repressor element in the 5′-untranslated region of human Pax5 exon 1A. Gene. 263 (1-2), 59-66 (2001).
- Mali, P., et al. RNA-Guided Human Genome Engineering via Cas9. Science. 339 (6121), 823-826 (2013).

