조정 나노 기공을 통해 나노 입자 전좌 속도를 통해 제타 전위의 측정 : 예를 들어 DNA 변성 입자를 사용하여
Summary
여기에서는 개개의 나노 입자의 제타 포텐셜을 결정하는데 사용될 수있다 입자 전위 속도의 측정을 통해 나노 입자의 표면 화학을 특징 저항 펄스 감지 기술에 통합 된 폴리 우레탄 동조 나노 기공을 사용한다.
Abstract
저항 펄스 센서 (RPS)로 총칭 알려진 나노 기공 기술, 탐지, 정량화 및 단백질 분자와 나노 입자의 특성을 사용하고있다. 조정 가능한 저항 펄스 센서 (TRPS)는 실시간으로 변경 될 수있는 조정 가능한 공극을 포함 RPS 비교적 최근에 적응된다. 그들은 DNA 농도 및 구조의 함수로서 가변 기공 멤브레인을 통과 여기 우리 DNA 변성 나노 입자의 전위의 시간을 모니터링하는 TRPS를 사용하여 (즉, 단일 쇄 이중 가닥 DNA)이다.
TRPS는 전계인가시 안정한 이온 전류를 설정 엘라스토머 기공 막에 의해 분리 된 두개의 Ag / AgCl을 전극에 기초한다. 각종 광학 기반 입자 특성 기술과 달리, TRPS는 복합 샘플 용이하게 분석 할 수 있도록하는 샘플 모집단 중에서 개개의 입자를 특성화 할 수있다. 여기, 우리는 제타 전위 측정을 보여공지 된 표준 입자 전위 속도 통해 따라서 이러한 분석의 제타 전위 측정 결과 피검 전좌 회 샘플링이 적용.
뿐만 아니라 평균 제타 전위 값을 획득하는 등, 샘플은 모든 예를 들어, 샘플 인구 분포를 통해 주어진 샘플에 대한 자세한 정보를 나타내는 입자에 의해 입자의 관점을 사용하여 측정하고 있습니다. 이러한 가운데,이 방법은 모두 의료 및 환경 분야에 대한 감지 애플리케이션 내에서 가능성을 보여줍니다.
Introduction
기능화 나노 입자는 의료 및 환경 분야 모두에서 바이오 센서로 점점 더 인기를 끌고있다. 능력은 예를 들어 표적 약물 전달 시스템 (1) 및 감시 DNA 단백질 상호 작용 2-4 유용 증명, DNA와 나노 입자의 표면 화학을 변경한다. 점점 일반적인 나노 입자의 특성은 생물 검정에서 활용 및 치료제의 전달에 초상 자성 5된다. 초상 자성 입자 (SPPS)를 식별하고 복잡한 혼합물에서 특정 분석을 제거하는데 매우 유용하며, 하나의 자석의 간단한 사용하여 수행 할 수 있습니다. 일단 제거 된 분석 결합 된 입자는 특징과 목적에 맞는 분석 할 수 있습니다.
나노 입자의 검출 및 특성화에 사용되는 이전의 방법은 달리 광자 상관 분광법으로 알려진 동적 광산란 (DLS) 등의 광학 기법을 포함한다. 안녕하세요 있지만GH 처리량 기술, DLS이 평균을 기반으로 기술되고 그리고 전문 소프트웨어의 추가없이 복합 시료를 분석하는 경우에 한정되어, 더 큰 입자는 -6,7- 완전히 알려지지 작은 입자의 일부를 남겨, 훨씬 더 우세 신호를 생성한다. 입자가 입자의 특성화 기술은 따라서 더욱 바람직한 나노 입자 및 나노 입자의 작용 화 시스템을 분석한다.
RPS 기반 기술은 시료에 전기장을인가하고, 합성 또는 생물학적 나노 기공을 통해 입자의 반송기구를 감시를 기반으로한다. RPS에 기초한 비교적 최근 나노 검출 및 특성화 기법은 가변 저항 펄스 센서 (TRPS) 8-16이다. TRPS는 엘라스토머, 가변 기공 막에 의해 분리 된 두 개의 전극 시스템이다. 파장 가변 세공 방법은 트랜스를 통해 측정되는 도형 (17)의 크기의 범위의 분석을 허용기공을 통해 포트 메커니즘. 동조 기공 종전에는 투과형 전자 분광법 (TEM) (10)과 같은 다른 기술들에 비교 결과를 생성하는 작은 입자의 검출 (70 내지 95 nm의 직경)에 사용되어왔다. 전기장이인가되면, 이온 전류가 관찰되는 입자 / 분자가 공극을 통과 할 때, 이들은 일시적 '봉쇄 이벤트'로 정의 될 수있는 전류의 감소를 일으키는 원인이되는 기공을 차단. 샘플 내의 각각의 입자 특성을 개별적으로 봉쇄 진폭, Δ에 기초 할 수 있도록 각 차단 이벤트는 하나의 입자의 대표 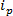 및 반값 폭, FWHM뿐만 아니라 다른 차단 특성. 그들은 나노 기공을 통과 할 때 개개의 입자를 분석하는 것은 입자 아몬 크기의 TRPS가 성공적으로 효과적으로 범위를 구별 할 수있는 복합 샘플에 유리하다하나의 샘플을 GST. 조정 저항 펄스 감지 크기 10, 단일 실행에서 동시에 제타 전위 12, 18, 20, 22 및 농도 (15) 측정을 완료하고 여전히 비슷한 샘플을 차별화 따라서 할 수있는 경우에 그 표면 전하 (19)에 의해하지 같은 크기; 대안 사이징 기술을 통해 장점.
및 반값 폭, FWHM뿐만 아니라 다른 차단 특성. 그들은 나노 기공을 통과 할 때 개개의 입자를 분석하는 것은 입자 아몬 크기의 TRPS가 성공적으로 효과적으로 범위를 구별 할 수있는 복합 샘플에 유리하다하나의 샘플을 GST. 조정 저항 펄스 감지 크기 10, 단일 실행에서 동시에 제타 전위 12, 18, 20, 22 및 농도 (15) 측정을 완료하고 여전히 비슷한 샘플을 차별화 따라서 할 수있는 경우에 그 표면 전하 (19)에 의해하지 같은 크기; 대안 사이징 기술을 통해 장점.
제타 전위는 전단 (20)의면에 정전 전위로 정의되며, 그들은 기공 (19)로 이동할 때 입자 속도로부터 계산된다. 개별 입자의 제타 전위 측정 따라서 전위기구 및 솔루션의 나노 입자 시스템, 응용 프로그램의 범위에 대한 나노 입자 분석 디자인의 미래를위한 가치있는 정보의 행동에 대한 통찰력을 제공합니다. 이러한 자연의 입자에 의해 입자 분석은 또한 오 자세한 내용은 허용 표본 집단 사이에 확산 및 제타 전위 값의 분포를 탐험 할 수 있습니다용액 N의 반응 속도 (예를 들어, 이중 가닥 DNA를 단일 가닥) 및 입자 안정성이 달성된다.
여기서 우리는 감지하고 수정되지 않은 및 DNA 변성 SPP 표면 모두를 특징 짓는 기술을 설명합니다. 여기에 기술 된 프로토콜은 무기 나노 입자와 생체의 범위에 적용 할 수 있지만,이 때문에 애플리케이션들은 광범위한 DNA 변성 표면을 사용하는 절차를 보여준다. 이 기술은 사용자에 따라서 입자 전좌 기공 시스템을 통해 속도 및 제타 전위에 기초하여, 나노 입자 표면 상에 단일 가닥 및 이중 가닥 DNA 표적을 구별 할 수있다.
Protocol
Representative Results
Discussion
제타 전위의 계산 Arjmandi 외. 21 일에 관련된 보정 기반 방법을 사용 하였다. 입자의 전위의 길이가 나노 기공이 규칙적 원추형 구멍의 전체에 걸쳐 평균 전계 입자의 속도를 이용하여,인가 전압의 함수로서 측정된다 이송있다. 전기 영동 이동성 감지 영역 길이의 제곱 L 곱한 전압에 대한 (T는 봉쇄 기간 임) 1 / T의 유도체이다. 감지 영역을 통해 다수의 기준 포인트의 평균…
Disclosures
The authors have nothing to disclose.
Acknowledgements
저자는 그들의 지원을 위해 Izon 과학 (주) 감사합니다. 이 연구는 연구 (PCIG11-GA-2012-321836 Nano4Bio)에 대한 유럽위원회 (European Commission)에 의해 지원되었다.
Materials
| Phosphate buffered Saline (PBS) | Sigma Aldrich, UK | P4417 | 1 tablet dissolved in 200 mL deionised water to make buffer solution. |
| Tween-20 | Sigma Aldrich, UK | P1379 | 0.05% (v/v) in PBS buffer as a surfactant |
| Carboxyl polystyrene nanoparticles | Bangs Laboratories, US | CPC200 | Nominal diamter of 220 nm, raw concentration of 1E12 particles/mL, specific surface charge of 86 µeq/g (equivalent to a surface charge density of 3.2E19 C/nm^2. |
| Streptavidin coated nanoparticles | Ademtech, France | 3121 | Batch had binding capacity of 4352 pmol/mg (188 nM theoretical DNA binding capacity) at a raw concentration of 1.1E11 particles/mL. |
| Biotinylated oligonucleotides | Sigma Aldrich, UK | VC00001 | Supplier spec: Reverse Phase 1 purification (0.05 Scale); Biotin modification at 3' end; Lyophilised powders reconstituted to 100 µM using deionised water, and diluted as required. Sequences: CP 5'ATGGTTAAACCTCAC TACGCGTGGC[Btn]3' |
| Standard olignonucleotides | Sigma Aldrich, UK | VC00001 | Supplier spec: Reverse Phase 1 purification (0.05 Scale); Lyophilised powders reconstituted to 100 µM using deionised water, and diluted as required. Sequences of DNA targets: Fully complementary – 5'GCCACGCGTAGTGAGGTTTAACCAT3', Middle binding – 5'GTAGTGAGGT3', End binding – 5'GTTTAACCAT3', Partially complementary overhanging – 5'GTGAGGTTTAACCAT TTTTTTTTTTTTTTT3'. |
| Izon qNano | Izon Science, NZ | Inherent pressure on system of 47 Pa, | |
| Izon Variable Pressure Module (VPM) | Izon Science, NZ | Each 'cm' of pressure is equivalent to approximately 1000 Pa. | |
| Polyurethane nanopore membranes | Izon Science, NZ | NP150 | Analyte size range 60-480 nm, pore diameter of calculated to be 799 nm at a 45 mm stretch. |
| Magrack 6 | GE Healthcare, UK | 28-9489-64 | |
| Sonic Bath | Fisher Scientific, UK | 10692353 | 80 Watts |
| Vortexer | IKA, Germany | 0003365000 | |
| Rotary Wheel | Labnet International, US | H5500-230 V |
References
- Alexander, C. M., Maye, M. M., Dabrowiak, J. C. DNA-capped nanoparticles designed for doxorubicin drug delivery. Chem Commun. 47 (12), 3418-3420 (2011).
- Billinge, E. R., Platt, M. Aptamer based dispersion assay using tunable resistive pulse sensing (TRPS). Anal Methods. 7 (20), 8534-8538 (2015).
- Bulyk, M. L. Protein Binding Microarrays for the Characterization of Protein-DNA Interactions. Adv Biochem Eng Biotechnol. 104, 65-85 (2007).
- Platt, M., Rowe, W., Knowles, J., Day, P. J., Kell, D. B. Analysis of aptamer sequence activity relationships. Integr Biol. 1 (1), 116-122 (2009).
- Ruiz-Hernández, E., Baeza, A., Vallet-Regí, M. Smart Drug Delivery through DNA/Magnetic Nanoparticle Gates. ACS Nano. 5 (2), 1259-1266 (2011).
- Murdock, R. C., Braydich-stolle, L., Schrand, A. M., Schlager, J. J., Hussain, S. M. Characterization of Nanomaterial Dispersion in Solution Prior to In Vitro Exposure Using Dynamic Light Scattering Technique. Toxicol Sci. 101 (2), 239-253 (2008).
- Hupfield, S., Holsaeter, A. M., Skar, M., Frantzen, C. B., Brandl, M. Liposome size analysis by dynamic/static light scattering upon size exclusion-/field flow fractionation. J Nanosci Nanotechnol. 6 (7), 3025-3031 (2006).
- Roberts, G. S., et al. Tunable pores for measuring concentrations of synthetic and biological nanoparticle dispersions. Biosens Bioelectron. 31 (1), 17-25 (2012).
- Roberts, G. S., Kozak, D., Anderson, W., Broom, M. F., Vogel, R., Trau, M. Tunable nano/micropores for particle detection and discrimination: scanning ion occlusion spectroscopy. Small. 6 (23), 2653-2658 (2010).
- Vogel, R., et al. Quantitative sizing of nano/microparticles with a tunable elastomeric pore sensor. Anal Chem. 83 (9), 3499-3506 (2011).
- Booth, M. A., Vogel, R., Curran, J. M., Harbison, S., Travas-Sejdic, J. Detection of target-probe oligonucleotide hybridization using synthetic nanopore resistive pulse sensing. Biosens Bioelectron. 45, 136-140 (2013).
- Kozak, D., Anderson, W., Vogel, R., Chen, S. Simultaneous size and ζ-potential measurements of individual nanoparticles in dispersion using size-tunable pore sensors. ACS Nano. 6 (8), 6990-6997 (2012).
- Kozak, D., Anderson, W., Vogel, R., Trau, M. Advances in Resistive Pulse Sensors: Devices bridging the void between molecular and microscopic detection. Nano Today. 6 (5), 531-545 (2011).
- Weatherall, E., Willmott, G. R. Applications of tunable resistive pulse sensing. Analyst. 140, 3318-3334 (2015).
- Willmott, G. R., et al. Use of tunable nanopore blockade rates to investigate colloidal dispersions. J Phys Condens Matter. 22 (45), 454116 (2010).
- Blundell, E. L. C. J., Mayne, L. J., Billinge, E. R., Platt, M. Emergence of tunable resistive pulse sensing as a biosensor. Anal Methods. 7, 7055-7066 (2015).
- Platt, M., Willmott, G. R., Lee, G. U. Resistive Pulse Sensing of Analyte-Induced Multicomponent Rod Aggregation Using Tunable Pores. Small. 8 (15), 2436-2444 (2012).
- Vogel, R., Anderson, W., Eldridge, J., Glossop, B., Willmott, G. A variable pressure method for characterizing nanoparticle surface charge using pore sensors. Anal Chem. 84 (7), 3125-3131 (2012).
- Blundell, E. L. C. J., Vogel, R., Platt, M. Particle-by-Particle Charge Analysis of DNA-Modified Nanoparticles Using Tunable Resistive Pulse Sensing. Langmuir. 32 (4), (2016).
- Hunter, R. J. . Zeta Potential in Colloid Science: Principles and Applications. , (1981).
- Arjmandi, N., Van Roy, W., Lagae, L., Borghs, G. Measuring the electric charge and zeta potential of nanometer-sized objects using pyramidal-shaped nanopores. Anal Chem. 84 (20), 8490-8496 (2012).
- Bacri, L., et al. Dynamics of colloids in single solid-state nanopores. J Phys Chem B. 115 (12), 2890-2898 (2011).
- Cabello-Aguilar, S., et al. Dynamics of polymer nanoparticles through a single artificial nanopore with a high-aspect-ratio. Soft Matter. 10 (42), 8413-8419 (2014).
- Billinge, E. R., Muzard, J., Platt, M. Tunable resistive pulse sensing as a tool to monitor analyte induced particle aggregation. Nanomater Nanosci. 1 (1), 11 (2013).
- Li, J., Fan, C., Pei, H., Shi, J., Huang, Q. Smart Drug Delivery Nanocarriers with Self-Assembled DNA Nanostructures. Adv Mater. 25 (32), 4386-4396 (2013).
- Billinge, E. R., Broom, M., Platt, M. Monitoring aptamer-protein interactions using tunable resistive pulse sensing. Anal Chem. 86 (2), 1030-1037 (2014).
- Gold, L., et al. Aptamer-Based Multiplexed Proteomic Technology for Biomarker Discovery. PLoS One. 5 (12), e15004 (2010).
- Park, S. -. J., Taton, T. A., Mirkin, C. A. Array-Based Electrical Detection of DNA with Nanoparticle Probes. Science. 295 (5559), 1503-1506 (2002).

