Fluoreszenzanisotropiemessungen als Werkzeug Protein-Protein-Interaktionen zu untersuchen
Summary
Protein-Wechselwirkungen sind im Herzen von einer Funktion der Zelle. Kalorimetrische und spektroskopischen Techniken werden allgemein zu charakterisieren sie verwendet. Hier beschreiben wir Fluoreszenzanisotropie als Werkzeug, um die Wechselwirkung zwischen dem Protein in dem Shwachman-Diamant-Syndrom (SBDS) und dem Elongationsfaktor-like 1 GTPase (EFL1) mutiert zu studieren.
Abstract
Protein-Protein-Interaktionen spielen eine wesentliche Rolle in der Funktion eines lebenden Organismus. Sobald eine Interaktion identifiziert und validiert ist es notwendig, sie auf die strukturelle und mechanistische Ebene zu charakterisieren. Mehrere biochemische und biophysikalische Methoden existieren für solche Zwecke. Unter ihnen ist Fluoreszenzanisotropie eine leistungsfähige Technik insbesondere verwendet, wenn die Fluoreszenzintensität eines Fluorophor-markierten Proteins konstant bleibt auf Protein-Protein-Interaktion. In dieser Technik wird ein Fluorophor markiertes Protein ist mit vertikal polarisiertem Licht einer geeigneten Wellenlänge angeregt, die selektiv eine Untergruppe der Fluorophore entsprechend ihrer relativen Orientierung mit dem ankommenden Strahl erregt. Die resultierende Emission hat auch eine Direktionalität , deren Beziehung in der vertikalen und horizontalen Ebenen definiert Anisotropie (r) wie folgt: r = (I VV -I VH) / (I + 2I VV VH), wobei I VV und I <sub> VH sind die Fluoreszenzintensitäten der vertikalen und horizontalen Komponenten, respectively. Fluoreszenzanisotropie ist empfindlich gegenüber der Rotationsdiffusion eines Fluorophors, nämlich die scheinbare Molekülgröße eines Fluorophors an ein Protein gebunden, die auf Protein-Protein-Wechselwirkung verändert wird. In dem vorliegenden Text ist die Verwendung von Fluoreszenzanisotropie als Werkzeug Protein-Protein-Interaktionen zu studieren, wurde beispielhaft die Bindung zwischen dem Protein in dem Shwachman-Diamant-Syndrom (SBDS) und den Elongationsfaktor like-1-GTPase (EFL1) mutiert zu adressieren. Herkömmlicherweise Markierung eines Proteins mit einem Fluorophor ist auf der Thiol-Gruppen (Cystein) oder in den Aminogruppen (die N-terminale Amin oder Lysin) ausgeführt des Proteins. Allerdings besitzt SBDS mehrere Cysteine und Lysine, die nicht ortsgerichtete Kennzeichnung es erlauben. Als alternative Technik, dem Farbstoff 4 ', 5'-bis (1,3,2 dithioarsolan-2-yl) fluorescein wurde verwendet, spezifisch ein Motiv Tetracysteinmotiv zu etikettieren, Cys-Cys-Pro-Gly-Cys-Cys, genetisch in dem C-Terminus des rekombinanten Proteins SBDS konstruiert. Fitting der experimentellen Daten bereitgestellt quantitative und mechanistische Information über den Bindungsmodus zwischen diesen Proteinen.
Introduction
Zellen enthalten eine Vielzahl von Biomakromolekülen, die ständig miteinander in Wechselwirkung treten. Diese Vereinigung führt zu Komplexen, die in den zellulären Wege, die für ihre Funktion in der Signaltransduktion, die Regulation der Genexpression und der Zellmigration unter anderem teilnehmen. Alle Protein-Protein-Wechselwirkungen, die in einer Zelle auftreten, umfassen ein Netzwerk wie das Interaktom bekannt. In Saccharomyces cerevisiae mehr als 70% ihrer Proteine wurden Interaktionspartner 1 gezeigt zu haben. Das Verständnis der Interaktom einer Zelle und deren Funktionen liefern relevante Informationen über die Komplexität und Vielfalt der Lebewesen. Verschiedene Methoden wurden Protein-Protein-Interaktionen beschrieben zu identifizieren und zu charakterisieren. Verschiedene Hochdurchsatzverfahren, wie Hefe – Zwei-Hybrid – 2 – Protein-Fragment Komplementierung Assays 3, Affinitätsreinigung 4 gekoppelt mit der Massenspektrometrie und Protein microarrays verwendet 5,6 eine Interaktion zu identifizieren. Einmal identifiziert, ist es notwendig, sie zu überprüfen und diese auf einer Fall-zu-Fall-Basis variieren. Typischerweise umfassen diese Versuche die Wechselwirkung selbst auf der Ebene der einzelnen Mitglieder des Wechselwirkungspaares zu stören, beispielsweise durch Gendeletion oder Überexpression eines der Proteine, und dann auf Änderungen in den Eigenschaften oder der Funktion des anderen Elements bei der Suche zellulärer Ebene. Anschließend werden biophysikalische Techniken 7 verwendet , um die Protein-Protein – Wechselwirkung auf molekularer Ebene zu charakterisieren. Zu diesem Zweck sind die Struktur von Protein-Komplexe, die durch Röntgenkristallographie bestimmt, kernmagnetische Resonanz und Kryo-Elektronenmikroskopie während Kalorimetrie und Fluoreszenzspektroskopie verwendet werden, um quantitativ und sie mechanistisch beschreiben.
In dieser Arbeit wurde Fluoreszenzanisotropie als eine Technik verwendet, um die Wechselwirkung zwischen dem GTPase EFL1 und der SBD zu charakterisierenS-Protein. Diese Proteine beteiligen sich an der Synthese von Ribosomen durch die Freisetzung von eukaryotischen Initiationsfaktor Förderung 6 von der Oberfläche der 60S ribosomalen Untereinheit 8. Das SBDS Protein wird in einer Krankheit als Shwachman-Diamond – Syndrom 9 und wirkt als ein Guaninnukleotid – Austauschfaktor für EFL1 abnehm seine Affinität für Guanosindiphosphat 10,11 bekannt mutiert. Krankheit Mutationen in SBDS die Interaktion mit EFL1 abzuschaffen und somit ihre Aktivierung verhindern.
Fluoreszenzanisotropie wird in biologischen Anwendungen zu untersuchen Protein-Peptid- oder Protein-Nukleinsäure-Wechselwirkungen verwendet. Sie basiert auf dem Prinzip, dass ein Fluorophor mit polarisiertem Licht führt zu einer teilweise polarisierte Emission angeregt. Fluoreszenzanisotropie wird durch Gleichung 1 definiert:
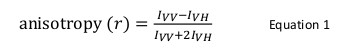
wo ich VV und ich VH sind dieFluoreszenzintensitäten des vertikal (VV) und horizontal (VH) polarisierte Emission , wenn die Probe angeregt wird , mit vertikal polarisiertem Licht 12. Fluoreszenzanisotropie ist empfindlich gegenüber Faktoren, die die Geschwindigkeit der Rotationsdiffusion des Fluorophors beeinflussen und somit abhängig von der Temperatur, der Viskosität der Lösung und die scheinbare Molekülgröße des Fluorophors. Die scheinbare Grße eines Proteins ein Fluorophor enthält, erhöht, wenn es mit einem anderen Protein und eine solche Änderung in Wechselwirkung kann dann als eine Änderung der Anisotropie ausgewertet werden. Genauer gesagt, der Wert und kleiner I VH Wert mit einem großen I VV und daher eine relativ große Anisotropie wird zeigen müssen ein Fluorophor, die sich langsam in Lösung relativ zu seiner Fluoreszenzlebensdauer dreht. Für Fluorophore, die sich schnell in Bezug auf ihre Fluoreszenzlebensdauer im Trockner, ich VV und ich VH wird wird ähnlich und ihre Anisotropie sein klein sein 12 </sup> (Abbildung 1). Darüber hinaus ist für eine gute Anisotropie Signal-Rausch-Messung ist es notwendig, ein Fluorophor mit einer Fluoreszenzlebensdauer ähnlich der Rotationskorrelationszeit des Moleküls von Interesse zu haben. Andernfalls ist es nicht möglich, genau den Unterschied in der Anisotropie zwischen dem freien Protein aufnehmen, und dass in der Anlage. Zum Beispiel kann die Anisotropie einer fluoreszierenden Sonde mit einer Lebensdauer der Nähe von 4 nsec, wie beispielsweise Fluorescein oder Rhodamin zu einer niedermolekularen Verbindung gebunden von 100 Da beträgt 0,05. Die Bindung an ein Molekül von 160 kDa seine Anisotropiewertes auf 0,29 zu erhöhen; ein Unterschied, der genau gemessen werden kann. Im Gegensatz dazu ging es um die gleiche Fluoreszenzsonde in einer Bindungsreaktion, deren Anstieg in Molekülgröße variiert von 65 bis 1.000 kDa führt nur in einer Anisotropie Änderung von 0,28 bis 0,3 ist, die zu klein ist, genau gemessen werden. In diesem Szenario wäre eine Sonde mit einer Lebensdauer von 400 ns mehr geeignet 12.
<pclass = "jove_content">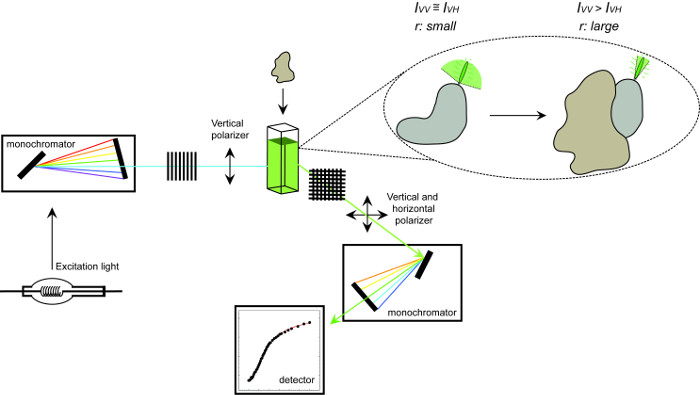
Abbildung 1 : Schematische Darstellung der Einrichtung zur Messung der Fluoreszenz – Anisotropie und die Prozedur. Schematische Darstellung der verwendeten Ausrüstung verwendet , um eine Protein-Protein – Wechselwirkung Experiment die Messung der Fluoreszenz – Anisotropie auszuführen. Fluorophore , die schnelle Anzeige geringe Anisotropie im Trockner , die bei der Bindung an einen Wechselwirkungspartner erhöht. Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
Fluoreszenzanwendungen erfordern das Vorhandensein eines Fluorophors in einem der untersuchten Moleküle. Zur Untersuchung von Protein-Protein-Wechselwirkungen gibt es drei Arten von Fluorophore: 1) die Tryptophan-Reste in den Proteinen, 2) chemisch gebunden Fluorophore und 3) fluoreszierende Fusionspartner wie das grün fluoreszierende Protein (GFP) und dessen derivativen. Die meisten Proteine haben Tryptophanreste auf seine Struktur und damit die einfachste Weg, um eine Interaktion zu messen, ist durch die Änderungen in dem entsprechenden Fluoreszenzspektren Überwachung oder durch Veränderungen in der Fluoreszenzintensität der Tryptophanreste zu überwachen. Jedoch können Tryptophanreste in beiden Proteinen vorhanden sein, die Analyse erschwert wird. Auf der anderen Seite, für ein Fluorophor aufgrund einer Wechselwirkung muss seine fluoreszierenden Eigenschaften ändern sie auf oder in der Nähe der Bindungsstelle lokalisiert werden und es könnte mit der Interaktion selbst stören. Dies erfordert besondere Aufmerksamkeit, wenn sperrige Fluorophore wie GFP verwenden. Wenn keine dieser Fluorophore können für Bindungsstudien verwendet werden, ist es notwendig, dann, um extrinsische Fluorophore an die eine der beteiligten Proteine einzuführen. Viele chemisch synthetisierte Fluorophore existieren und können kovalent an Proteine über ihre reaktiven Gruppen, wie den Amingruppen (Seitenkette von Lysinen oder N-Terminus) und die Thiolgruppen in Cystein gebunden sein. Fluorophore Derivate mit Isothiocyanat und Succinimidylester reagieren mit Amidgruppen während iodoacetamide und Maleimid sind thiolreaktives Gruppen 13. Die am häufigsten verwendeten Farbstoffe in Fluoreszenzanwendungen verwendet werden, sind Derivate des Fluorescein und Rhodamin grüne Farbstoffe, Cumarine, BODIPY Fluorophore und Alexa Fluor Farbstoffe. Eine detaillierte Liste der im Handel erhältlichen Fluorophore und ihre Verwendung finden Sie in Referenzen 14,15 gefunden werden. Für eine erfolgreiche Markierung muss die reaktive Gruppe auf der Oberfläche des Proteins exponiert werden, aber aufgrund der großen Anzahl von reaktiven funktionellen Gruppen typischerweise in Polypeptiden ist es sehr schwer ortsspezifische Modifikation zu erhalten. Das Protein von Interesse in dieser Studie SBDS, enthält 5 freie Cysteine und 33 Lysine, die in mehreren Standorten Kennzeichnung führen kann. Uneinheitliche Kennzeichnung kann die Bindung beeinflussen und Datenanalyse komplizieren als unterschiedliche Fluorophormoleküle unterschiedlichen Fluoreszenzintensitätssignale bei der Bindung auslösen kann. ÖVErcome dieses Problem haben wir den FlAsH Fluorophore, 4 ', 5'-Bis (1,3,2 dithioarsolan-2-yl) fluorescein orts direkt das SBDS Protein-Label. Dies ist ein arsenoxide Farbstoff mit einer hohen Affinität für vier beabstandete Cysteine in einem Motiv kennen als Flash-tag der Sequenz CCXXCC aus wobei X für jede Aminosäure außer Cystein ist 16,17. Dieses Motiv wird Tetracysteinmotiv gentechnologisch zusammen mit einem geeigneten Linker an den N- oder C-Terminus des Proteins hinzugefügt, um die Störung des Gesamtfaltung des Proteins zu verhindern. Das Paar , das aus FlAsH Farbstoff und FlAsH-Tag wurde ursprünglich auf ortsspezifische Label Proteine entwickelt in lebenden Zellen 17 , sondern es kann auch gereinigte Proteine verwendet werden , in vitro zu beschriften , wie sie hier veranschaulicht wird. Zusätzlich haben enzymatische Strategien auch 18 ortsspezifische Funktionalisierung von Proteinen zu ermöglichen , entwickelt worden.
In diesem Manuskript beschreiben wir die Nützlichkeit von Fluoreszenzanisotropiemessungen einsa Werkzeug Protein-Protein-Wechselwirkungen zu untersuchen. Die Bindung kann durch einfache Inspektion der Bindungskurvenform bewertet werden, während quantitative Informationen können aus der Anpassung der experimentellen Daten erhalten werden.
Protocol
Representative Results
Discussion
Die meisten biochemischen Experimenten mit Proteinen erfordern nicht nur reines Protein, sondern auch große Mengen von ihnen unabhängig von der verwendeten Technik. Aus diesem Grund werden die Proteine für diese Art von Versuchen verwendet werden, durch die heterologe Expression erhalten wird, wie es der Fall vorgestellt hier war. Florescence Spektroskopie erfordert die Anwesenheit eines Fluorophors in der untersuchten Moleküls. Aromatische Reste darstellen, die intrinsischen Fluorophoren eines Proteins jedoch…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Authors acknowledge the financial support from CONACyT project numbers 167359 and 177138, and from DGAPA-UNAM project number IN201615.
Materials
| 0.5 mm Glass beads | Biospec Products | 11079105 | |
| Tris Base | Formedium | TRIS01 | Ultra pure |
| Glycerol | Sigma-Aldrich | G5516 | |
| dye 4’, 5’-bis(1,3,2 dithioarsolan-2-yl) fluorescein | ThermoFischer Scientific | LC6090 | This kit contains the dye to label a FlAsH tag |
| Ampiciline | IBI Shelton Scientific, Inc | IB02040 | |
| D(+)-Glucose Anhydrous | Formedium | GLU03 | |
| D(+)-Galactose | Formedium | GAL03 | |
| L-Leucine | Formedium | DOC0157 | |
| L-Tryptofan | Formedium | DOC0189 | |
| Bezamidine hydrochloride | Sigma-Aldrich | B6506-5G | |
| PMSF | Gold Biotechnology, Inc | P-470-25 | Phenylmethylsulfonyl fluoride |
| NaCl | Formedium | NAC02 | Sodium Chloride |
| Glycerol | Tecsiquim, S.A. de C.V. | GT1980-6 | |
| MgCl2 | Merck Millipore Corporation | 1725711000 | Magnesium Chloride |
| Imidazole | Sigma-Aldrich | I2399-500G | |
| 2-Mercaptoethanol | Sigma-Aldrich | M6250-100ML | |
| K2HPO4 | Sigma-Aldrich | P3786-500G | Potassium phosphate dibasic |
| NaH2PO4 | Sigma-Aldrich | S3139-500G | Sodium phosphate monobasic |
| Yeast nitrogen base without amino acids | Formedium | CYN0410 | |
| Yeast extract | Formedium | YEM03 | Micro Granulated |
| L-Tyroisne | Formedium | DOC0193 | |
| Adenine sulphate | Formedium | DOC0230 | |
| Casamino acids | Formedium | CAS03 | |
| Tryptone | IBI Shelton Scientific, Inc | IB49182 | |
| IPTG | Formedium | IPTG025 | |
| Name of Material | Company | Catalog Number | Comments/Description |
| Filtration units | Merck Millipore Corporation | UFC901096 | Amicon Ultra-15, membrana PLGC Ultracel-PL, 10 kDa |
| Membrane Filter | Merck Millipore Corporation | GSWP04700 | Membrane Filter, mixed cellulose esters, Hydrophilic, 0.22 µm, 47 mm, white, plain |
| Ni2+ affinity column | QIAGEN | 30760 | Cartridge pre-filled with 5 ml Ni-NTA Superflow |
| Strong Sulfopropyl cation exchanger column | GE Healthcare Life Science | 17-5157-01 | HiTrap SP Sepharose FF 5 ml |
| Size Exclusion column | GE Healthcare Life Science | 28989335 | HiLoad 16/600 Superdex 200 PG |
| Fluorescence cell | Hellma Analytics | 111-057-40 | |
| Name of Equipment | Company | Catalog Number | Comments/Description |
| Spectrophotometer | Agilent Technologies | G6860AA | Cary 60 UV-Vis |
| Shaker | ThermoFischer Scientific | SHKA4000-7 | MaxQ 4000 Benchtop temperature range Ambient-15° to 60°C |
| Centrifuge | ThermoFischer Scientific | 75004271 | Heraeus Megafuge 16R |
| FPLC | Pharmacia Biotech | Discontinued | FPLC system conductivity UV-MM II monitor P500 pump fraction |
| Spectrofluorometer | Olis | No applicable | Olis DM 45 with Polarization Toolbox |
References
- Krogan, N. J., et al. Global landscape of protein complexes in the yeast Saccharomyces cerevisiae. Nature. 440 (7084), 637-643 (2006).
- Fields, S., Song, O. A novel genetic system to detect protein-protein interactions. Nature. 340, 245-246 (1989).
- Michnick, S. W., Hien Ear, P., Landry, C., Malleshaiah, M. K., Messier, V. A toolkit of protein-fragment complementation assays for studying and dissecting large-scale and dynamic protein-protein interactions in living cells. Methods in Enzymology. 470, 336-366 (2010).
- Puig, O., et al. The tandem affinity purification (TAP) method: a general procedure of protein complex purification. Methods. 24, 218-229 (2001).
- Dwane, S., Kiely, P. A. Tools used to study how protein complexes are assembled in signaling cascades. Bioeng Bugs. 2 (5), 247-259 (2011).
- Snider, J., et al. Fundamentals of protein interaction network mapping. Mol Syst Biol. 11 (12), 848 (2015).
- Fersht, A., Baldwin, R. L. . Structure and mechanism in protein science: a guide to enzyme catalysis and protein folding. , 191-214 (2002).
- Menne, T. F., et al. The Shwachman-Bodian-Diamond syndrome protein mediates translational activation of ribosomes in yeast. Nat Genet. 39 (4), 486-495 (2007).
- Boocock, G. R., et al. Mutations in SBDS are associated with Shwachman-Diamond syndrome. Nat Genet. 33 (1), 97-101 (2003).
- Garcia-Marquez, A., Gijsbers, A., de la Mora, E., Sanchez-Puig, N. Defective Guanine Nucleotide Exchange in the Elongation Factor-like 1 (EFL1) GTPase by Mutations in the Shwachman-Diamond Syndrome Protein. J Biol Chem. 290 (29), 17669-17678 (2015).
- Gijsbers, A., Garcia-Marquez, A., Luviano, A., Sanchez-Puig, N. Guanine nucleotide exchange in the ribosomal GTPase EFL1 is modulated by the protein mutated in the Shwachman-Diamond syndrome. Biochem Biophys Res Commun. 437 (3), 349-354 (2013).
- Lakowicz, J. R. . Principles of fluorescence spectroscopy. , (2010).
- Nishigaki, T., Treviño, C. L., Gòmez, I. . Tools to understand protein-protein interactions. 37, 1-14 (2012).
- Johnson, I. . The Molecular Probes Handbook: A Guide to Fluorescent Probes and Labeling Technologies. , (2010).
- Sabnis, R. W. . Handbook of Fluorescent Dyes and Probes. , (2015).
- Adams, S. R., et al. New biarsenical ligands and tetracysteine motifs for protein labeling in vitro and in vivo: synthesis and biological applications. J Am Chem Soc. 124 (21), 6063-6076 (2002).
- Griffin, B. A., Adams, S. R., Tsien, R. Y. Specific covalent labeling of recombinant protein molecules inside live cells. Science. 281 (5374), 269-272 (1998).
- Rashidian, M., Dozier, J. K., Distefano, M. D. Enzymatic labeling of proteins: techniques and approaches. Bioconjug Chem. 24 (8), 1277-1294 (2013).
- Maniatis, T., Fritsch, E. F., Sambrook, J. . Molecular cloning: A laboratory manual. , (2001).
- Neuhoff, V., Arold, N., Taube, D., Ehrhardt, W. Improved staining of proteins in polyacrylamide gels including isoelectric focusing gels with clear background at nanogram sensitivity using Coomassie Brilliant Blue G-250 and R-250. Electrophoresis. 9 (6), 255-262 (1988).
- West, R. W., Chen, S. M., Putz, H., Butler, G., Banerjee, M. GAL1-GAL10 divergent promoter region of Saccharomyces cerevisiae contains negative control elements in addition to functionally separate and possibly overlapping upstream activating sequences. Genes Dev. 1 (10), 1118-1131 (1987).
- Ito, H., Fukuda, Y., Murata, K., Kimura, A. Transformation of intact yeast cells treated with alkali cations. J Bacteriol. 153 (1), 163-168 (1983).
- Eftink, M. R. Fluorescence methods for studying equilibrium macromolecule-ligand interactions. Methods Enzymol. 278, 221-257 (1997).
- Han, H., et al. Binding of Substrates to the Central Pore of the Vps4 ATPase Is Autoinhibited by the Microtubule Interacting and Trafficking (MIT) Domain and Activated by MIT Interacting Motifs (MIMs). J Biol Chem. 290 (21), 13490-13499 (2015).
- Sanchez-Puig, N., Veprintsev, D. B., Fersht, A. R. Binding of natively unfolded HIF-1alpha ODD domain to p53. Mol Cell. 17 (1), 11-21 (2005).
- Trusch, F., et al. The N-terminal Region of the Ubiquitin Regulatory X (UBX) Domain-containing Protein 1 (UBXD1) Modulates Interdomain Communication within the Valosin-containing Protein p97. J Biol Chem. 290 (49), 29414-29427 (2015).
- Kamp, F., Beyer, K. Binding of alpha-synuclein affects the lipid packing in bilayers of small vesicles. The Journal of Biological Chemsitry. 281, 9251-9259 (2006).
- Bujalowski, W. M., Jezewska, M. J. Fluorescence Intensity, Anisotropy, and Transient Dynamic Quenching Stopped-Flow Kinetics. Spectroscopic Methods of Analysis. 875, 105-133 (2012).
- Asano, N., et al. Direct interaction between EFL1 and SBDS is mediated by an intrinsically disordered insertion domain. Biochem Biophys Res Commun. 443 (4), 1251-1256 (2014).

