Anisotropia di fluorescenza come strumento per studiare le interazioni proteina-proteina
Summary
interazioni proteina sono al centro della funzione di una cellula. tecniche calorimetriche e spettroscopiche sono comunemente usati per caratterizzare loro. Qui si descrive anisotropia di fluorescenza come strumento per studiare l'interazione tra la proteina mutata nella sindrome di Shwachman-Diamond (SBDS) e il fattore di allungamento-simile 1 GTPase (EFL1).
Abstract
Le interazioni proteina-proteina svolgono un ruolo essenziale nella funzione di un organismo vivente. Una volta che l'interazione è stata identificata e convalidata è necessario caratterizzare al livello strutturale e meccanicistica. Esistono diversi metodi biochimici e biofisici per tale scopo. Tra questi, anisotropia di fluorescenza è una tecnica potente particolarmente utilizzata quando l'intensità di fluorescenza di una proteina fluoroforo marcato rimane costante mediante interazione proteina-proteina. In questa tecnica, una proteina fluoroforo marcato è eccitato con luce polarizzata verticalmente di opportuna lunghezza d'onda che eccita selettivamente un sottoinsieme dei fluorofori in base al loro orientamento rispetto al fascio in ingresso. L'emissione risultante ha anche una direzionalità cui rapporto in piani verticali e orizzontali definisce anisotropia (r) come segue: r = (I VV -I VH) / (I + 2I VV VH), dove Vv e <sub> VH sono le intensità di fluorescenza dei componenti verticale ed orizzontale, rispettivamente. Anisotropia di fluorescenza è sensibile alla diffusione di rotazione di un fluoroforo, vale a dire la dimensione molecolare apparente di un fluoroforo attaccato ad una proteina, che viene modificato mediante interazione proteina-proteina. Nel testo attuale, l'uso di anisotropia di fluorescenza come strumento per studiare le interazioni proteina-proteina è stato esemplificato per affrontare il legame tra la proteina mutata nella sindrome del Shwachman-Diamond (SBDS) e il fattore di allungamento like-1 GTPase (EFL1). Convenzionalmente, l'etichettatura di una proteina con un fluoroforo viene effettuata sui gruppi tiolici (cisteina) o dei gruppi amminici (N-terminali amminici o lisina) della proteina. Tuttavia, SBDS possiede diversi cisteine e lisine che non consentono sito-etichettatura di esso. In una tecnica alternativa, il colorante 4 ', 5'-bis (1,3,2 dithioarsolan-2-il) fluoresceina è stato usato per etichettare specificamente un motivo tetracysteine, Cys-Cys-Pro-Gly-Cys-Cys, geneticamente nel C-terminale della proteina ricombinante SBDS. Montaggio dei dati sperimentali fornito informazioni quantitative e meccanicistica della modalità di legame tra queste proteine.
Introduction
Le cellule contengono una moltitudine di biomacromolecole che interagiscono costantemente con l'altro. Questa associazione dà luogo a complessi che partecipano alle vie cellulari responsabili per il loro funzionamento nella trasduzione del segnale, regolazione dell'espressione genica e la migrazione delle cellule, tra gli altri. Tutte le interazioni proteina-proteina che si verificano in una cella comprendono una rete nota come dell'interattoma. In Saccharomyces cerevisiae hanno dimostrato più del 70% delle proteine di avere partner interagenti 1. Comprendere la interattoma di una cella e le loro funzioni di fornire informazioni pertinenti sulla complessità e la diversità degli organismi viventi. Diverse metodologie sono state descritte per identificare e caratterizzare le interazioni proteina-proteina. Diverso alto attraverso metodi messi come il lievito doppio ibrido 2, saggi di complementazione proteine frammento 3, affinità di purificazione 4 accoppiata alla spettrometria di massa e microarra proteineys vengono utilizzati per identificare una interazione 5,6. Una volta identificato, è necessario convalidare e puo variare caso per caso. Tipicamente, questi esperimenti comportano interrompere stessa interazione a livello dei singoli membri della coppia interazione, ad esempio, dalla delezione gene o sovraespressione di una delle proteine, e quindi cerca di cambiamenti nelle proprietà o funzione di un altro membro del livello cellulare. Successivamente, tecniche biofisiche 7 sono utilizzati per caratterizzare l'interazione proteina-proteina a livello molecolare. A tal fine, la struttura di complessi proteici sono determinati mediante cristallografia a raggi X, risonanza magnetica nucleare e la microscopia crioelettronica mentre calorimetria e spettroscopia di fluorescenza sono utilizzati per descriverli quantitativamente e meccanicamente.
In questo lavoro, anisotropia di fluorescenza è stato utilizzato come tecnica per caratterizzare l'interazione tra la GTPasi EFL1 e SBDS proteine. Queste proteine partecipano nella sintesi dei ribosomi promuovendo il rilascio di fattore di inizio eucariotico 6 dalla superficie 60S ribosomiale subunità 8. La proteina SBDS è mutato in una malattia nota come Shwachman-Diamond Sindrome 9 e agisce come un fattore di scambio guanina nucleotide per EFL1 diminuendo la sua affinità per guanosindifosfato 10,11. mutazioni malattia in SBDS abolire l'interazione con EFL1 e quindi impediscono la sua attivazione.
Fluorescenza anisotropia è comunemente usato in applicazioni biologiche per studiare la proteina-peptide o interazioni degli acidi nucleici proteine. Si basa sul principio che un fluoroforo eccitato con risultati luce polarizzata in una emissione parzialmente polarizzata. Anisotropia di fluorescenza è definito dall'equazione 1:
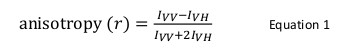
dove ho vv e mi VH sono ilintensità di fluorescenza della verticale (VV) e in orizzontale (VH) polarizzati emissione quando il campione è eccitato con luce polarizzata verticalmente 12. Anisotropia di fluorescenza è sensibile a fattori che influenzano la velocità di diffusione rotazionale del fluoroforo e dipende dalla temperatura, la viscosità della soluzione e la dimensione molecolare apparente del fluoroforo così. La dimensione apparente di una proteina contenente un fluoroforo aumenta quando interagisce con un'altra proteina e tale modifica può essere valutato come un cambiamento di anisotropia. Più specificamente, un fluoroforo che ruota lentamente in soluzione rispetto alla sua durata fluorescente avrà un valore I VV e piccolo valore I VH e pertanto presentano una relativamente grande anisotropia. Per fluorofori che scendono rapidamente rispetto al loro ciclo di vita fluorescente, ho Vv e VH saranno simili e il loro valore anisotropia sarà piccolo 12 </sup> (Figura 1). Inoltre, per un buon segnale anisotropia di misura del rumore, è necessario avere un fluoroforo con una vita di fluorescenza simile al tempo di correlazione rotazionale della molecola di interesse. Altrimenti, non è possibile registrare accuratamente la differenza di anisotropia tra la proteina libero e che nel complesso. Ad esempio, l'anisotropia di una sonda fluorescente con una durata quasi 4 nsec come fluoresceina o rodamina collegati a un composto a basso peso molecolare di 100 Da è 0.05. Associazione a una molecola di 160 kDa aumenterà il suo valore anisotropia a 0,29; una differenza che può essere misurata con precisione. Al contrario, la stessa sonda fluorescente coinvolto in una reazione di legame cui incremento di peso molecolare varia da 65 a 1000 kDa comporterà solo un cambiamento anisotropia di 0,28 e 0,3, che è troppo piccola per essere misurata con precisione. In questo scenario, una sonda con una durata di 400 nanosecondi sarebbe più adatto 12.
<pclass = "jove_content">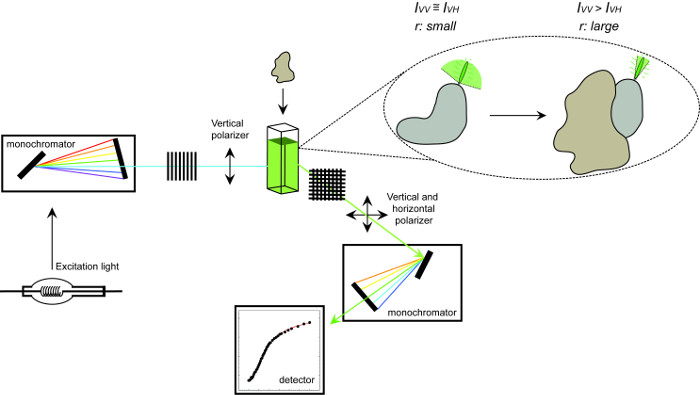
Figura 1. Rappresentazione schematica delle apparecchiature utilizzate per misurare anisotropia di fluorescenza e la procedura. Rappresentazione schematica dell'attrezzatura utilizzata per eseguire una anisotropia di fluorescenza misurazione proteina-proteina interazione esperimento. Fluorofori che scendono visualizzazione rapida piccolo anisotropia che aumenta dopo il legame a un partner di interazione. Cliccate qui per vedere una versione più grande di questa figura.
applicazioni fluorescenza richiedono la presenza di un fluoroforo in una qualsiasi delle molecole studiate. Per studiare le interazioni proteina-proteina ci sono tre tipi di fluorofori: 1) i residui di triptofano presenti nelle proteine, 2) fluorofori allegate chimicamente e 3) partner di fusione fluorescenti come la proteina fluorescente verde (GFP) e la sua Derivative. La maggior parte delle proteine hanno residui di triptofano sulla sua struttura, così il modo più semplice per misurare un'interazione è monitorando i cambiamenti nel corrispondente spettri di fluorescenza o monitorando i cambiamenti nell'intensità di fluorescenza dei residui di triptofano. Tuttavia, residui di triptofano possono essere presenti in entrambe le proteine complicare l'analisi. D'altra parte, per un fluoroforo per modificare le proprietà fluorescenti dovuto all'interazione deve essere situato sopra o vicino al sito di legame e potrebbe interferire con l'interazione stessa. Questo richiede particolare attenzione quando si utilizza fluorofori ingombranti come GFP. Se nessuno di questi fluorofori può essere utilizzato per studi di legame è necessario, quindi, di introdurre fluorofori estrinseci a una delle proteine coinvolte. Molti fluorofori sintetizzati chimicamente esistono e possono essere covalentemente attaccati alle proteine attraverso i loro gruppi reattivi come i gruppi amminici (catena laterale di lisine o N-terminale) ei gruppi tiolici in cisteina. Fderivati luorophore con isotiocianato e succinimidyl esteri reagiscono con gruppi ammidici mentre Iodoacetamide e maleimmide sono gruppi tiolo-reattivi 13. I coloranti più comuni utilizzati in applicazioni in fluorescenza sono derivati del fluoresceina ei coloranti verdi rodamina, cumarine, fluorofori BODIPY e coloranti Alexa Fluor. Un elenco dettagliato dei fluorofori disponibili in commercio e il loro uso può essere trovato in riferimenti 14,15. Per l'etichettatura di successo, il gruppo reattivo deve essere esposto sulla superficie della proteina, ma a causa del gran numero di gruppi funzionali reattivi tipicamente presenti in polipeptidi è molto difficile ottenere modificazioni sito-specifica. La proteina di interesse in questo studio, SBDS, contiene 5 cisteine libere e 33 lisine che possono provocare l'etichettatura più siti. l'etichettatura non uniforme può influenzare il legame e complicherà l'analisi dei dati come diverse molecole fluorofori possono suscitare diversi segnali di intensità fluorescente su vincolante. a Overcome questo problema, abbiamo usato il fluoroforo flash, 4 ', 5'-bis (1,3,2 dithioarsolan-2-il) fluoresceina all'etichetta sito-diretta la proteina SBDS. Questo è un colorante arsenoxide con alta affinità per quattro cisteine distanziati in un motivo conosciuto come FlAsH-tag costituito dalla CCXXCC sequenza in cui X è un qualsiasi amminoacido diverso cisteina 16,17. Questo motivo tetracysteine viene aggiunto N- o C-terminale della proteina mediante ingegneria genetica insieme ad un linker opportuno evitare l'interruzione della piega complessiva della proteina. La coppia che consiste di Flash tintura e Flash-tag è stato originariamente progettato per le proteine di etichette site-specific nelle cellule viventi 17, ma può anche essere usato per etichettare le proteine purificate in vitro come è esemplificato qui. Inoltre, sono stati sviluppati strategie enzimatici per consentire funzionalizzazione site-specific di proteine 18.
In questo manoscritto descriviamo l'utilità di anisotropia di fluorescenza unTool SA per studiare le interazioni proteina-proteina. Binding può essere valutata semplice ispezione della forma vincolante curva mentre le informazioni quantitative può essere ottenuta dalla misura dei dati sperimentali.
Protocol
Representative Results
Discussion
Maggior parte degli esperimenti biochimici con proteine richiedono non solo proteina pura, ma anche grandi quantità di loro, indipendentemente dalla tecnica utilizzata. Per questo motivo, le proteine utilizzate per questo tipo di esperimenti sono ottenuti mediante espressione eterologa, come era il caso qui presentato. spettroscopia Florescence richiede la presenza di un fluoroforo nella molecola studiata. residui aromatici costituiscono i fluorofori intrinseci di una proteina, tuttavia, utilizzando il segn…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Authors acknowledge the financial support from CONACyT project numbers 167359 and 177138, and from DGAPA-UNAM project number IN201615.
Materials
| 0.5 mm Glass beads | Biospec Products | 11079105 | |
| Tris Base | Formedium | TRIS01 | Ultra pure |
| Glycerol | Sigma-Aldrich | G5516 | |
| dye 4’, 5’-bis(1,3,2 dithioarsolan-2-yl) fluorescein | ThermoFischer Scientific | LC6090 | This kit contains the dye to label a FlAsH tag |
| Ampiciline | IBI Shelton Scientific, Inc | IB02040 | |
| D(+)-Glucose Anhydrous | Formedium | GLU03 | |
| D(+)-Galactose | Formedium | GAL03 | |
| L-Leucine | Formedium | DOC0157 | |
| L-Tryptofan | Formedium | DOC0189 | |
| Bezamidine hydrochloride | Sigma-Aldrich | B6506-5G | |
| PMSF | Gold Biotechnology, Inc | P-470-25 | Phenylmethylsulfonyl fluoride |
| NaCl | Formedium | NAC02 | Sodium Chloride |
| Glycerol | Tecsiquim, S.A. de C.V. | GT1980-6 | |
| MgCl2 | Merck Millipore Corporation | 1725711000 | Magnesium Chloride |
| Imidazole | Sigma-Aldrich | I2399-500G | |
| 2-Mercaptoethanol | Sigma-Aldrich | M6250-100ML | |
| K2HPO4 | Sigma-Aldrich | P3786-500G | Potassium phosphate dibasic |
| NaH2PO4 | Sigma-Aldrich | S3139-500G | Sodium phosphate monobasic |
| Yeast nitrogen base without amino acids | Formedium | CYN0410 | |
| Yeast extract | Formedium | YEM03 | Micro Granulated |
| L-Tyroisne | Formedium | DOC0193 | |
| Adenine sulphate | Formedium | DOC0230 | |
| Casamino acids | Formedium | CAS03 | |
| Tryptone | IBI Shelton Scientific, Inc | IB49182 | |
| IPTG | Formedium | IPTG025 | |
| Name of Material | Company | Catalog Number | Comments/Description |
| Filtration units | Merck Millipore Corporation | UFC901096 | Amicon Ultra-15, membrana PLGC Ultracel-PL, 10 kDa |
| Membrane Filter | Merck Millipore Corporation | GSWP04700 | Membrane Filter, mixed cellulose esters, Hydrophilic, 0.22 µm, 47 mm, white, plain |
| Ni2+ affinity column | QIAGEN | 30760 | Cartridge pre-filled with 5 ml Ni-NTA Superflow |
| Strong Sulfopropyl cation exchanger column | GE Healthcare Life Science | 17-5157-01 | HiTrap SP Sepharose FF 5 ml |
| Size Exclusion column | GE Healthcare Life Science | 28989335 | HiLoad 16/600 Superdex 200 PG |
| Fluorescence cell | Hellma Analytics | 111-057-40 | |
| Name of Equipment | Company | Catalog Number | Comments/Description |
| Spectrophotometer | Agilent Technologies | G6860AA | Cary 60 UV-Vis |
| Shaker | ThermoFischer Scientific | SHKA4000-7 | MaxQ 4000 Benchtop temperature range Ambient-15° to 60°C |
| Centrifuge | ThermoFischer Scientific | 75004271 | Heraeus Megafuge 16R |
| FPLC | Pharmacia Biotech | Discontinued | FPLC system conductivity UV-MM II monitor P500 pump fraction |
| Spectrofluorometer | Olis | No applicable | Olis DM 45 with Polarization Toolbox |
References
- Krogan, N. J., et al. Global landscape of protein complexes in the yeast Saccharomyces cerevisiae. Nature. 440 (7084), 637-643 (2006).
- Fields, S., Song, O. A novel genetic system to detect protein-protein interactions. Nature. 340, 245-246 (1989).
- Michnick, S. W., Hien Ear, P., Landry, C., Malleshaiah, M. K., Messier, V. A toolkit of protein-fragment complementation assays for studying and dissecting large-scale and dynamic protein-protein interactions in living cells. Methods in Enzymology. 470, 336-366 (2010).
- Puig, O., et al. The tandem affinity purification (TAP) method: a general procedure of protein complex purification. Methods. 24, 218-229 (2001).
- Dwane, S., Kiely, P. A. Tools used to study how protein complexes are assembled in signaling cascades. Bioeng Bugs. 2 (5), 247-259 (2011).
- Snider, J., et al. Fundamentals of protein interaction network mapping. Mol Syst Biol. 11 (12), 848 (2015).
- Fersht, A., Baldwin, R. L. . Structure and mechanism in protein science: a guide to enzyme catalysis and protein folding. , 191-214 (2002).
- Menne, T. F., et al. The Shwachman-Bodian-Diamond syndrome protein mediates translational activation of ribosomes in yeast. Nat Genet. 39 (4), 486-495 (2007).
- Boocock, G. R., et al. Mutations in SBDS are associated with Shwachman-Diamond syndrome. Nat Genet. 33 (1), 97-101 (2003).
- Garcia-Marquez, A., Gijsbers, A., de la Mora, E., Sanchez-Puig, N. Defective Guanine Nucleotide Exchange in the Elongation Factor-like 1 (EFL1) GTPase by Mutations in the Shwachman-Diamond Syndrome Protein. J Biol Chem. 290 (29), 17669-17678 (2015).
- Gijsbers, A., Garcia-Marquez, A., Luviano, A., Sanchez-Puig, N. Guanine nucleotide exchange in the ribosomal GTPase EFL1 is modulated by the protein mutated in the Shwachman-Diamond syndrome. Biochem Biophys Res Commun. 437 (3), 349-354 (2013).
- Lakowicz, J. R. . Principles of fluorescence spectroscopy. , (2010).
- Nishigaki, T., Treviño, C. L., Gòmez, I. . Tools to understand protein-protein interactions. 37, 1-14 (2012).
- Johnson, I. . The Molecular Probes Handbook: A Guide to Fluorescent Probes and Labeling Technologies. , (2010).
- Sabnis, R. W. . Handbook of Fluorescent Dyes and Probes. , (2015).
- Adams, S. R., et al. New biarsenical ligands and tetracysteine motifs for protein labeling in vitro and in vivo: synthesis and biological applications. J Am Chem Soc. 124 (21), 6063-6076 (2002).
- Griffin, B. A., Adams, S. R., Tsien, R. Y. Specific covalent labeling of recombinant protein molecules inside live cells. Science. 281 (5374), 269-272 (1998).
- Rashidian, M., Dozier, J. K., Distefano, M. D. Enzymatic labeling of proteins: techniques and approaches. Bioconjug Chem. 24 (8), 1277-1294 (2013).
- Maniatis, T., Fritsch, E. F., Sambrook, J. . Molecular cloning: A laboratory manual. , (2001).
- Neuhoff, V., Arold, N., Taube, D., Ehrhardt, W. Improved staining of proteins in polyacrylamide gels including isoelectric focusing gels with clear background at nanogram sensitivity using Coomassie Brilliant Blue G-250 and R-250. Electrophoresis. 9 (6), 255-262 (1988).
- West, R. W., Chen, S. M., Putz, H., Butler, G., Banerjee, M. GAL1-GAL10 divergent promoter region of Saccharomyces cerevisiae contains negative control elements in addition to functionally separate and possibly overlapping upstream activating sequences. Genes Dev. 1 (10), 1118-1131 (1987).
- Ito, H., Fukuda, Y., Murata, K., Kimura, A. Transformation of intact yeast cells treated with alkali cations. J Bacteriol. 153 (1), 163-168 (1983).
- Eftink, M. R. Fluorescence methods for studying equilibrium macromolecule-ligand interactions. Methods Enzymol. 278, 221-257 (1997).
- Han, H., et al. Binding of Substrates to the Central Pore of the Vps4 ATPase Is Autoinhibited by the Microtubule Interacting and Trafficking (MIT) Domain and Activated by MIT Interacting Motifs (MIMs). J Biol Chem. 290 (21), 13490-13499 (2015).
- Sanchez-Puig, N., Veprintsev, D. B., Fersht, A. R. Binding of natively unfolded HIF-1alpha ODD domain to p53. Mol Cell. 17 (1), 11-21 (2005).
- Trusch, F., et al. The N-terminal Region of the Ubiquitin Regulatory X (UBX) Domain-containing Protein 1 (UBXD1) Modulates Interdomain Communication within the Valosin-containing Protein p97. J Biol Chem. 290 (49), 29414-29427 (2015).
- Kamp, F., Beyer, K. Binding of alpha-synuclein affects the lipid packing in bilayers of small vesicles. The Journal of Biological Chemsitry. 281, 9251-9259 (2006).
- Bujalowski, W. M., Jezewska, M. J. Fluorescence Intensity, Anisotropy, and Transient Dynamic Quenching Stopped-Flow Kinetics. Spectroscopic Methods of Analysis. 875, 105-133 (2012).
- Asano, N., et al. Direct interaction between EFL1 and SBDS is mediated by an intrinsically disordered insertion domain. Biochem Biophys Res Commun. 443 (4), 1251-1256 (2014).

