Opdræt og dobbelt-strenget RNA-medieret Gene Knockdown i Skjul Beetle,<em> Dermestes maculatus</em
Summary
Her præsenterer vi protokoller for opdræt en mellemliggende-kim bille, Dermestes maculatus (D. maculatus) i laboratoriet. Vi deler også protokoller for embryonale og forældreorlov RNAi og metoder til at analysere embryonale fænotyper at studere genfunktion i denne art.
Abstract
Advances in genomics have raised the possibility of probing biodiversity at an unprecedented scale. However, sequence alone will not be informative without tools to study gene function. The development and sharing of detailed protocols for the establishment of new model systems in laboratories, and for tools to carry out functional studies, is thus crucial for leveraging the power of genomics. Coleoptera (beetles) are the largest clade of insects and occupy virtually all types of habitats on the planet. In addition to providing ideal models for fundamental research, studies of beetles can have impacts on pest control as they are often pests of households, agriculture, and food industries. Detailed protocols for rearing and maintenance of D. maculatus laboratory colonies and for carrying out dsRNA-mediated interference in D. maculatus are presented. Both embryonic and parental RNAi procedures-including apparatus set up, preparation, injection, and post-injection recovery-are described. Methods are also presented for analyzing embryonic phenotypes, including viability, patterning defects in hatched larvae, and cuticle preparations for unhatched larvae. These assays, together with in situ hybridization and immunostaining for molecular markers, make D. maculatus an accessible model system for basic and applied research. They further provide useful information for establishing procedures in other emerging insect model systems.
Introduction
I 1998 Fire og Mello rapporterede, at dobbeltstrenget RNA (dsRNA) kan fremkalde hæmning af gen funktion i Caenorhabditis elegans 1. Denne reaktion udløst af dsRNA blev opkaldt RNA-interferens (RNAi), og sådan RNAi-medieret gen silencing blev rapporteret at være bevaret i dyr, planter og svampe 2-7. I planter og nogle dyr, RNAi fungerer systemisk, hvilket betyder, at virkningen kan sprede sig til andre celler / væv, hvor dsRNA ikke er direkte indført (gennemgået i 8-10). Forskere har gjort brug af denne endogene cellulære RNAi respons ved at designe dsRNA'er at målrette gener af interesse, og dermed vælte gen funktion uden direkte at manipulere genomet (revideret i 11-14).
RNAi er et stærkt værktøj til funktionelle undersøgelser på grund af følgende fordele. Første, selv med minimal gensekvensinformation, et gen kan målrettes ved hjælp af RNAi. Dette er især vigtigt for studies af ikke-modelorganismer mangler genomiske eller transkriptom data. For det andet, i organismer hvor RNAi respons er robust systemisk, RNAi-medieret gen knockdown kan udføres på næsten alle udviklingstrin. Denne funktion er meget nyttig til undersøgelse af funktionen af pleiotrope gener. For det tredje, i nogle tilfælde spredes RNAi effekter til gonaderne og afkom, således at fænotyper hos afkommet 15,16. Dette fænomen, kendt som forældrenes RNAi (pRNAi), er særligt fordelagtigt for gener påvirker fosterudviklingen, så talrige afkom af en enkelt indsprøjtet forælder kan undersøges uden direkte manipulation af æg. Af disse grunde, pRNAi er den foretrukne fremgangsmåde. Men hvis pRNAi er ineffektiv, for eksempel for gener, der er nødvendige for oogenesen derefter embryonale RNAi (eRNAi) skal anvendes. For det fjerde kan RNAi anvendes til at frembringe hvad der svarer til en allelisk serie ved, at mængden af dsRNA leverede kan varieres over et område for at frembringe svage til stærke defekter. En sådan graduering af fænotyper kan være nyttigt for forståelsen genfunktioner når genet er involveret i en kompleks proces og / eller fuldstændigt tab af funktion er dødelig. Femte, levering af dsRNA er generelt let og muligt, især i dyr, der viser robuste systemiske RNAi svar. dsRNA kan indføres ved mikroinjektion 1,5, fodring / indtagelse 17,18, iblødsætning, 19,20 og virus / bakterie-medieret levering 21,22. Sjette, i modsætning til nogle genmålretning / redigering metoder, er der ingen grund til at screene for organismer, der bærer mutationen eller foretage genetiske krydsninger at generere homozygoter ved brug RNAi. Derfor sammenlignet med mange andre teknikker til at studere genfunktion, RNAi er hurtig, billig, og kan anvendes til store skærme 23-25.
Den brede anvendelighed af RNAi tilvejebringer midler til at udføre funktionelle studier i en lang række organismer, udvide række arter til rådighed til undersøgelse beyond de traditionelle modelsystemer for hvilke der er udviklet genetiske værktøjer. For eksempel er undersøgelser med anvendelse af ikke-modelsystemer forpligtet til at give indblik i udviklingen af gener og gen-netværk ved at sammenligne funktionerne i ortologer fra arter, der repræsenterer forskellige udviklingsprojekter tilstande eller udstilles distinkte morfologiske træk 26-29. Disse typer af undersøgelser vil give en bedre forståelse af den biologiske mangfoldighed, med konsekvenser for både anvendt forskning og grundforskning.
Som den største dyregruppe på planeten, insekter giver en stor mulighed for at udforske de mekanismer, der ligger mangfoldighed. Derudover insekter er generelt små, har korte livscyklus, høj frugtbarhed, og er lette at opdrætte i laboratoriet. I de seneste to årtier har RNAi blevet anvendt med succes i insekter spænder ordrer, herunder Diptera (sande fluer) 5, Lepidoptera (sommerfugle og møl) 30, Coleoptera (biller) 16,31, Hymenoptera (sawfløgne, hvepse, myrer og bier) 32, Hemiptera (sande bugs), Isoptera (termitter) 34, Blattodea (kakerlakker) 35, Orthoptera (fårekyllinger, græshopper, græshopper og katydids) 36 og Phthiraptera (lus) 37. Vellykket anvendelse af RNAi har givet funktionelle data for studier af mønstre i tidlig embryogenese (anterior-posterior akse 32, dorsal-ventral akse 28, segmentering 26,38), kønsbestemmelse 39,40, kitin / neglebånd biosyntese 41, ecdyson signalering 42, social adfærd 43, og mere. RNAi metoder udviklet til forskellige insektarter kan være af yderligere fordel ved, at de sandsynligvis vil være nyttige til skadedyrsbekæmpelse (gennemgået i 44-46). RNAi effekter vil være genspecifikke samt artsspecifik, så længe ikke-konserverede regioner valgt til målretning. For gavnlige insektarter som honningbier og silkeorme, rettet mod gener vitale for overlevelsevira eller parasitter at kontrollere infektion kan tilvejebringe en ny strategi til at beskytte disse arter 47,48.
Dermestes maculatus (D. maculatus), almindelig betegnelse skjul bille, fordeles over hele verden med undtagelse af Antarktis. Som holometabolous insekt, D. maculatus livscyklus omfatter embryonale, larve, puppe og voksne stadier (Figur 1). Fordi det feeds på kødet, er D. maculatus bruges i museer at skeletonize døde dyr og retsmedicinske Entomologer kan bruge det til at vurdere dødstidspunktet 49,50. D. maculatus feeds på animalske produkter, herunder kroppe, tørret kød, ost og den pupper / kokoner af andre insekter og forårsager dermed skade på husholdninger, opbevarede fødevarer, og silke, ost, og kødbranchen 51,52. Anvendelse RNAi i denne bille kunne give en effektiv og miljøvenlig måde at minimere dens økonomiske virkninger. Vores laboratorium har brugt D. maculatus som en ny model insekt at studere segmentering 53. Ud over at være modtagelig for lab opdræt, D. maculatus er af interesse for grundforskning, da det er et mellemprodukt-kim udvikler, hvilket gør det til et nyttigt art at studere overgangen mellem kort og langt kim udvikling.
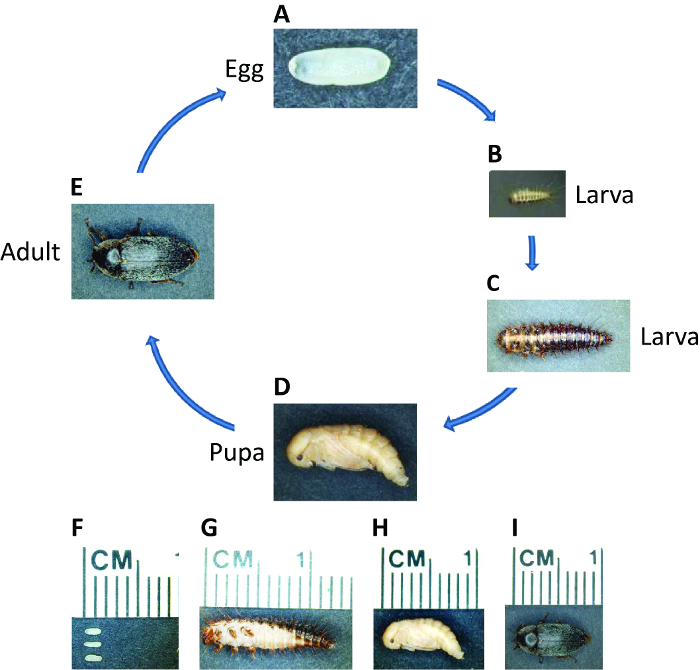
Figur 1: Life Cycle of D. maculatus. Fotografier af D. maculatus på forskellige livsstadier, som angivet. Den livscyklus fra æg til voksen tager tre uger ved 30 ° C, men længere ved lavere temperaturer. (A, F) frisk fastsat embryoner er hvide til lys gul og ovale, ca. 1,5 mm i længde. Embryogenese tager ~ 55 timer ved 30 ° C. (B, C og G) Larver har mørke pigmenterede striber og er dækket med setae. Larver gå gennem flere instars afhængig af miljøet, og deres længde kan strække sig op til over 1 cm. (D, H) </strong> Unge pupper er lys gul. Pupation tager ~ 5 – 7 dage ved 30 ° C. (E, I) Kort efter eclosion synes mørk pigmentering i den voksne bille krop. Voksne kan leve op til flere måneder og en kvindelig kan lægge hundredvis af embryoner i hendes levetid. Klik her for at se en større version af dette tal.
Tidligere viste vi, at RNAi er effektivt til at vælte genfunktion i D. maculatus 53. Her vores erfaring opdræt D. maculatus kolonier i laboratoriet deles sammen med trin-for-trin protokoller for både embryonale og forældreorlov RNAi set-up, injektion, efter injektion pleje, og fænotypisk analyse. De dsRNA-medieret gen Knockdown og analysemetoder indført her ikke kun give detaljerede oplysninger til adressering spørgsmål i D. maculatus, men også have potentiel betydning for anvendelse af RNAi i andre ikke-model bille / insektarter.
Protocol
Representative Results
Discussion
Mens et lille antal avancerede modelsystemer (mus, fluer, orme) blev udviklet i løbet af det 20. århundrede, har det 21. århundrede set en bølge af nye dyr, der udvikles i laboratorier i hele verden. Disse nye systemer give forskerne til at løse sammenlignende, evolutionære spørgsmål, der ikke kan sonderet kun at bruge de 'standard' modelsystemer. Denne implementering af nye modeller kræver den hurtige udvikling af metoder til lab dyrkning, gen-identifikation, og funktionelle tilga…
Disclosures
The authors have nothing to disclose.
Acknowledgements
We thank Drs. Alison Heffer and Yong Lu for setting up the microinjection apparatus and sharing their invaluable knowledge and experience with insect RNAi. This work was supported by the National Institutes of Health (R01GM113230 to L.P.).
Materials
| Dermestes maculatus live beetles | Our lab or Carolina Biological Supply | #144168 | Our lab strain was verified by COI barcoding; strain variation from Carolina cannot be ruled out |
| Wet cat food | Fancy Feast | Chunks of meat with gravy. Can buy at most pet food and grocery stores | |
| Dry dog food | Purina Puppy Chow | Can buy at most pet food and grocery stores | |
| Insect cage (size medium, 30.5x19x20.3 cm) | Exo Terra | PT2260 | For colony maintenance. Can use larger cage if needed |
| Insect cage (size mini, 17.8×10.2×12.7 cm) | Exo Terra | PT2250 | For embryo collection |
| Petri dish | VWR | 89038-968 | |
| Cotton ball | Fisher | 22-456-883 | |
| Megascript T7 transcription kit | Fisher | AM1334 | For 40 reactions |
| Pneumatic pump | WPI | PV830 | |
| Capillary holder | WPI | ||
| Micromanipulator | NARISHIGE | MN-151 | |
| Black filter paper (90 mm) | VWR | 28342-010 | |
| Food coloring (green) | McCormick | ||
| Borosilicate glass capillary | Hilgenberg | 1406119 | |
| Needle puller (micropipette puller) | Sutter Instrument Co. | P-97 | |
| Microscope glass slide | WorldWide Life Sciences Division | 41351157 | |
| Sealing film (Parafilm M) | Fisher | 13-374-12 | |
| Model 801 Syringe (10 µl ) | Hamilton | 7642-01 | |
| Needle (32-gauge) | Hamilton | 7762-05 | |
| Fixation Solution (Pampel's) | BioQuip Products, Inc. | 1184C | Toxic, needs to be handled in fume hood |
| Forcep (DUMONT #5) | Fine Science Tools | 11252-30 | |
| Cover slip (24X50 mm, No. 1.5) | Globe Scientific | 1415-15 | |
| Eppendorf Femtotips Microloader pipette tip | Fisher | E5242956003 | |
| Dissecting microscopy for embryo injection | Leica | M420 | |
| Dissecting microscopy for larval phenotypic visualization | Zeiss | SteREO Discover. V12 | |
| DIC microscopy | Zeiss | AXIO Imager. M1 | |
References
- Fire, A., et al. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature. 391, 806-811 (1998).
- Wianny, F., Zernicka-Goetz, M. Specific interference with gene function by double-stranded RNA in early mouse development. Nat Cell Biol. 2, 70-75 (2000).
- Svoboda, P., Stein, P., Hayashi, H., Schultz, R. M. Selective reduction of dormant maternal mRNAs in mouse oocytes by RNA interference. Development. 127, 4147-4156 (2000).
- Zimmermann, T. S., et al. RNAi-mediated gene silencing in non-human primates. Nature. 441, 111-114 (2006).
- Kennerdell, J. R., Carthew, R. W. Use of dsRNA-mediated genetic interference to demonstrate that frizzled and frizzled 2 act in the wingless pathway. Cell. 95, 1017-1026 (1998).
- Cogoni, C., et al. Transgene silencing of the al-1 gene in vegetative cells of Neurospora is mediated by a cytoplasmic effector and does not depend on DNA-DNA interactions or DNA methylation. EMBO J. 15, 3153-3163 (1996).
- Napoli, C., Lemieux, C., Jorgensen, R. Introduction of a Chimeric Chalcone Synthase Gene into Petunia Results in Reversible Co-Suppression of Homologous Genes in trans. Plant Cell. 2, 279-289 (1990).
- van Roessel, P., Brand, A. H. Spreading silence with Sid. Genome Biol. 5, 208 (2004).
- Grishok, A. RNAi mechanisms in Caenorhabditis elegans. FEBS Lett. 579, 5932-5939 (2005).
- Jose, A. M., Hunter, C. P. Transport of sequence-specific RNA interference information between cells. Annu Rev Genet. 41, 305-330 (2007).
- Hannon, G. J. RNA interference. Nature. 418, 244-251 (2002).
- Hammond, S. M., Caudy, A. A., Hannon, G. J. Post-transcriptional gene silencing by double-stranded RNA. Nat Rev Genet. 2, 110-119 (2001).
- Dorsett, Y., Tuschl, T. siRNAs: applications in functional genomics and potential as therapeutics. Nat Rev Drug Discov. 3, 318-329 (2004).
- Agrawal, N., et al. RNA interference: biology, mechanism, and applications. Microbiol Mol Biol Rev. 67, 657-685 (2003).
- Grishok, A., Tabara, H., Mello, C. C. Genetic requirements for inheritance of RNAi in C. elegans. Science. 287, 2494-2497 (2000).
- Bucher, G., Scholten, J., Klingler, M. Parental RNAi in Tribolium (Coleoptera). Curr Biol. 12, 85-86 (2002).
- Timmons, L., Fire, A. Specific interference by ingested dsRNA. Nature. 395, 854 (1998).
- Turner, C. T., et al. RNA interference in the light brown apple moth, Epiphyas postvittana (Walker) induced by double-stranded RNA feeding. Insect Mol Biol. 15, 383-391 (2006).
- Tabara, H., Grishok, A., Mello, C. C. RNAi in C. elegans: soaking in the genome sequence. Science. 282, 430-431 (1998).
- Eaton, B. A., Fetter, R. D., Davis, G. W. Dynactin is necessary for synapse stabilization. Neuron. 34, 729-741 (2002).
- Travanty, E. A., et al. Using RNA interference to develop dengue virus resistance in genetically modified Aedes aegypti. Insect Biochem Mol Biol. 34, 607-613 (2004).
- Whitten, M. M., et al. Symbiont-mediated RNA interference in insects. Proc Biol Sci. 283, (2016).
- Schmitt-Engel, C., et al. The iBeetle large-scale RNAi screen reveals gene functions for insect development and physiology. Nat Commun. 6, 7822 (2015).
- Dönitz, J., et al. iBeetle-Base: a database for RNAi phenotypes in the red flour beetle Tribolium castaneum. Nucleic Acids Res. 43, 720-725 (2015).
- Ulrich, J., et al. Large scale RNAi screen in Tribolium reveals novel target genes for pest control and the proteasome as prime target. BMC Genomics. 16, 674 (2015).
- Choe, C. P., Miller, S. C., Brown, S. J. A pair-rule gene circuit defines segments sequentially in the short-germ insect Tribolium castaneum. Proc. Natl. Acad. Sci. U. S. A. 103, 6560-6564 (2006).
- Angelini, D. R., Kaufman, T. C. Functional analyses in the hemipteran Oncopeltus fasciatus reveal conserved and derived aspects of appendage patterning in insects. Dev Biol. 271, 306-321 (2004).
- Lynch, J. A., Peel, A. D., Drechsler, A., Averof, M., Roth, S. EGF signaling and the origin of axial polarity among the insects. Curr Biol. 20, 1042-1047 (2010).
- Tenlen, J. R., McCaskill, S., Goldstein, B. RNA interference can be used to disrupt gene function in tardigrades. Dev Genes Evol. 223, 171-181 (2013).
- Quan, G. X., Kanda, T., Tamura, T. Induction of the white egg 3 mutant phenotype by injection of the double-stranded RNA of the silkworm white gene. Insect Mol Biol. 11, 217-222 (2002).
- Brown, S. J., Mahaffey, J. P., Lorenzen, M. D., Denell, R. E., Mahaffey, J. W. Using RNAi to investigate orthologous homeotic gene function during development of distantly related insects. Evol Dev. 1, 11-15 (1999).
- Lynch, J. A., Brent, A. E., Leaf, D. S., Pultz, M. A., Desplan, C. Localized maternal orthodenticle patterns anterior and posterior in the long germ wasp Nasonia. Nature. 439, 728-732 (2006).
- Liu, P. Z., Kaufman, T. C. hunchback is required for suppression of abdominal identity, and for proper germband growth and segmentation in the intermediate germband insect Oncopeltus fasciatus. Development. 131, 1515-1527 (2004).
- Zhou, X., Wheeler, M. M., Oi, F. M., Scharf, M. E. RNA interference in the termite Reticulitermes flavipes through ingestion of double-stranded RNA. Insect Biochem Mol Biol. 38, 805-815 (2008).
- Ciudad, L., Piulachs, M. D., Bellés, X. Systemic RNAi of the cockroach vitellogenin receptor results in a phenotype similar to that of the Drosophila yolkless mutant. FEBS J. 273, 325-335 (2006).
- Mito, T., et al. Non-canonical functions of hunchback in segment patterning of the intermediate germ cricket Gryllus bimaculatus. Development. 132, 2069-2079 (2005).
- Yoon, K. S., et al. Brief exposures of human body lice to sublethal amounts of ivermectin over-transcribes detoxification genes involved in tolerance. Insect Mol Biol. 20, 687-699 (2011).
- Rosenberg, M. I., Brent, A. E., Payre, F., Desplan, C. Dual mode of embryonic development is highlighted by expression and function of Nasonia pair-rule genes. Elife. 3, 01440 (2014).
- Hasselmann, M., et al. Evidence for the evolutionary nascence of a novel sex determination pathway in honeybees. Nature. 454, 519-522 (2008).
- Shukla, J. N., Palli, S. R. Sex determination in beetles: production of all male progeny by parental RNAi knockdown of transformer. Sci Rep. 2, 602 (2012).
- Arakane, Y., et al. The Tribolium chitin synthase genes TcCHS1 and TcCHS2 are specialized for synthesis of epidermal cuticle and midgut peritrophic matrix. Insect Mol Biol. 14, 453-463 (2005).
- Cruz, J., Mané-Padròs, D., Bellés, X., Martìn, D. Functions of the ecdysone receptor isoform-A in the hemimetabolous insect Blattella germanica revealed by systemic RNAi in vivo. Dev Biol. 297, 158-171 (2006).
- Guidugli, K. R., et al. Vitellogenin regulates hormonal dynamics in the worker caste of a eusocial insect. FEBS Lett. 579, 4961-4965 (2005).
- Zhang, H., Li, H. C., Miao, X. X. Feasibility, limitation and possible solutions of RNAi-based technology for insect pest control. Insect Sci. 20, 15-30 (2013).
- Huvenne, H., Smagghe, G. Mechanisms of dsRNA uptake in insects and potential of RNAi for pest control: a review. J Insect Physiol. 56, 227-235 (2010).
- Price, D. R., Gatehouse, J. A. RNAi-mediated crop protection against insects. Trends Biotechnol. 26, 393-400 (2008).
- Paldi, N., et al. Effective gene silencing in a microsporidian parasite associated with honeybee (Apis mellifera) colony declines. Appl Environ Microbiol. 76, 5960-5964 (2010).
- Kanginakudru, S., et al. Targeting ie-1 gene by RNAi induces baculoviral resistance in lepidopteran cell lines and in transgenic silkworms. Insect Mol Biol. 16, 635-644 (2007).
- Magni, P. A., Voss, S. C., Testi, R., Borrini, M., Dadour, I. R. A Biological and Procedural Review of Forensically Significant Dermestes Species (Coleoptera: Dermestidae). J Med Entomol. 52, 755-769 (2015).
- Zanetti, N. I., Visciarelli, E. C., Centeno, N. D. The Effect of Temperature and Laboratory Rearing Conditions on the Development of Dermestes maculatus (Coleoptera: Dermestidae). J Forensic Sci. , (2015).
- Veer, V., Negi, B. K., Rao, K. M. Dermestid beetles and some other insect pests associated with stored silkworm cocoons in India, including a world list of dermestid species found attacking this commodity. Journal of Stored Products Research. 32, 69-89 (1996).
- Xiang, J., Forrest, I. S., Pick, L. Dermestes maculatus: an intermediate-germ beetle model system for evo-devo. Evodevo. 6, 32 (2015).
- Fontenot, E. A., Arthur, F. H., Hartzer, K. L. Oviposition of Dermestes maculatus DeGeer, the hide beetle, as affected by biological and environmental conditions. Journal of Stored Products Research. 64, 154-159 (2015).
- Heffer, A., Grubbs, N., Mahaffey, J., Pick, L. The evolving role of the orphan nuclear receptor ftz-f1, a pair-rule segmentation gene. Evol Dev. 15, 406-417 (2013).
- Heffer, A., Shultz, J. W., Pick, L. Surprising flexibility in a conserved Hox transcription factor over 550 million years of evolution. Proc. Natl. Acad. Sci. U. S. A. 107, 18040-18045 (2010).
- Wilson, M. J., Dearden, P. K. Pair-rule gene orthologues have unexpected maternal roles in the honeybee (Apis mellifera). PLoS One. 7, 46490 (2012).
- Dawes, R., Dawson, I., Falciani, F., Tear, G., Akam, M. Dax, a locust Hox gene related to fushi-tarazu but showing no pair-rule expression. Development. 120, 1561-1572 (1994).
- Erezyilmaz, D. F., Kelstrup, H. C., Riddiford, L. M. The nuclear receptor E75A has a novel pair-rule-like function in patterning the milkweed bug, Oncopeltus fasciatus. Dev Biol. 334, 300-310 (2009).
- Stuart, J. J., Brown, S. J., Beeman, R. W., Denell, R. E. A deficiency of the homeotic complex of the beetle Tribolium. Nature. 350, 72-74 (1991).
- Aranda, M., Marques-Souza, H., Bayer, T., Tautz, D. The role of the segmentation gene hairy in Tribolium. Dev Genes Evol. 218, 465-477 (2008).
- Mito, T., et al. even-skipped has gap-like, pair-rule-like, and segmental functions in the cricket Gryllus bimaculatus, a basal, intermediate germ insect (Orthoptera). Dev Biol. 303, 202-213 (2007).
- Patel, N. H., Ball, E. E., Goodman, C. S. Changing role of even-skipped during the evolution of insect pattern formation. Nature. 357, 339-342 (1992).
- Nüsslein-Volhard, C., Wieschaus, E. Mutations affecting segment number and polarity in Drosophila. Nature. 287, 795-801 (1980).
- Jürgens, G., Wieschaus, E., Nüsslein-Volhard, C., Kluding, H. Mutations affecting the pattern of the larval cuticle in Drosophila melanogaster. II. Zygotic loci on the third chromosome. Wilhelm Roux’s archives of developmental biology. 193, 283-295 (1984).
- Wakimoto, B. T., Kaufman, T. C. Analysis of larval segmentation in lethal genotypes associated with the Aantennapedia gene complex in Drosophila melanogaster. Dev. Biol. 81, 51-64 (1981).
- Yu, Y., et al. The nuclear hormone receptor Ftz-F1 is a cofactor for the Drosophila homeodomain protein Ftz. Nature. 385, 552-555 (1997).
- Nüsslein-Volhard, C., Wieschaus, E., Kluding, H. Mutations affecting the pattern of the larval cuticle in Drosophila melanogaster. I.Zygotic loci on the second chromosome. Wilhelm Roux’s archives of developmental biology. 193, 267-282 (1984).
- Wieschaus, E., Nüsslein-Volhard, C., Jürgens, G. Mutations affecting the pattern of the larval cuticle in Drosophila melanogaster. III.Zygotic loci on the X-chromosome and fourth chromosome. ‘Wilhelm Roux’s archives of developmental biology. 193, 296-307 (1984).
- Guichet, A., et al. The nuclear receptor homologue Ftz-F1 and the homeodomain protein Ftz are mutually dependent cofactors. Nature. 385, 548-552 (1997).
- Fontenot, E. A., Arthur, F. H., Hartzer, K. L. Effect of diet and refugia on development of Dermestes maculatus DeGeer reared in a laboratory. J Pest Sci. 88, 113-119 (2014).
- Yang, Y., et al. Biodegradation and Mineralization of Polystyrene by Plastic-Eating Mealworms: Part 1. Chemical and Physical Characterization and Isotopic Tests. Environ Sci Technol. 49, 12080-12086 (2015).
- Kitzmann, P., Schwirz, J., Schmitt-Engel, C., Bucher, G. RNAi phenotypes are influenced by the genetic background of the injected strain. BMC Genomics. 14, 5 (2013).
- Chandler, C. H., Chari, S., Tack, D., Dworkin, I. Causes and consequences of genetic background effects illuminated by integrative genomic analysis. Genetics. 196, 1321-1336 (2014).
- Montagutelli, X. Effect of the genetic background on the phenotype of mouse mutations. J Am Soc Nephrol. 11, 101-105 (2000).
- Doetschman, T. Influence of genetic background on genetically engineered mouse phenotypes. Methods Mol Biol. 530, 423-433 (2009).

