Conception expérimentale pour Laser Microdissection ARN-Seq: Leçons tirées d'une analyse du développement du maïs Feuille
Summary
Many developmentally important genes have cell- or tissue-specific expression patterns. This paper describes LM RNA-seq experiments to identify genes that are differentially expressed at the maize leaf blade-sheath boundary and in lg1-R mutants compared to wild-type. The experimental considerations discussed here apply to transcriptomic analyses of other developmental phenomena.
Abstract
Les gènes ayant des rôles importants dans le développement ont souvent des profils d'expression spatialement et / ou temporellement restreint. Souvent, ces transcrits de gènes ne sont pas détectés ou ne sont pas identifiés comme étant différentiellement exprimés (DE) dans l'analyse du transcriptome d'organes de la plante entière. Laser Microdissection ARN-Seq (LM ARN-Seq) est un outil puissant pour identifier les gènes qui sont DE dans des domaines spécifiques de développement. Cependant, le choix des domaines cellulaires pour microdissect et comparer, et la précision des microdissections sont cruciales pour le succès des expériences. Ici, deux exemples illustrent des considérations de conception pour les expériences de transcriptomique; une LM analyse de l' ARN-seq pour identifier les gènes qui sont DE selon l'axe proximal-distal maïs feuille, et une seconde expérience pour identifier les gènes qui sont dans DE liguleless1-R (lg1-R) mutants par rapport au type sauvage. Les éléments clés qui ont contribué au succès de ces expériences ont été détaillées histologiques et situ analyses d' hybridation de la région à analyser, la sélection des primordiums de feuilles à un stade de développement équivalent, l'utilisation de points de repère pour sélectionner des régions morphologiques pour microdissection, et microdissection des domaines précisément mesurés. Ce document fournit un protocole détaillé pour l'analyse des domaines de développement en LM ARN-Seq. Les données présentées ici montrent comment la région sélectionnée pour microdissection aura une incidence sur les résultats obtenus.
Introduction
La feuille de maïs est un modèle idéal pour étudier la formation des champs de développement au cours de la morphogenèse, car il a une frontière distincte entre la lame et la gaine qui se prête à la dissection génétique (Figure 1A). Pendant les premiers stades de développement de la feuille, une bande linéaire de cellules plus petites, la bande de preligule (PLB), subdivise l'ébauche de feuille en pré-lame et pré-gaine domaines. Une ligule frange semblable et oreillettes triangulaires se développent à partir de la PLB (Figure 1A, C, D). écrans génétiques ont identifié des mutations qui perturbent la limite lame-gaine. Par exemple, liguleless1 récessive (LG1) mutations supprimer la ligule et oreillettes 1, 2, 3, 4 (figure 1B). L'hybridation in situ a révélé que la transcription lg1 accumule au PLB et ligule émergents, ce qui en fait un excellent marqueur pour le développement ligule 5, 6 (figure 1E).
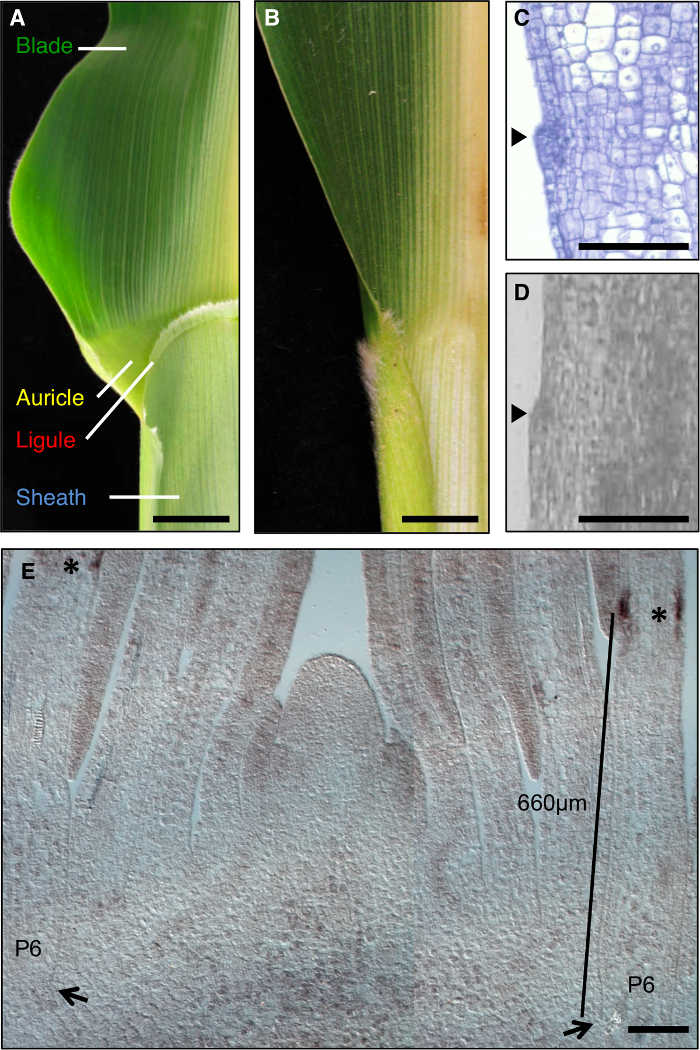
Figure 1: type sauvage et liguleless1-R feuilles de maïs. (A) région limite Blade-gaine de maturité des feuilles de type sauvage montrant les structures de ligule et oreillette. (B) de la région de la frontière Blade-gaine mature liguleless1-R feuille montrant l' absence de structures de ligule et oreillette. Feuilles en A et B ont été coupés en deux le long de la nervure médiane. (C) longitudinale coupe de type sauvage feuille primordium. L'échantillon a été traité et colorées pour l'analyse histologique. Initier la ligule ressort comme une bosse en saillie par rapport au plan de la lame (pointe de flèche). (D) sect longitudinaleion par type sauvage feuille primordium. L'échantillon a été traité pour LM, comme décrit dans le texte. Arrowhead indique lancer ligule. (E) LG1 hybridation in situ de la tige latérale en coupe longitudinale de sommet en. Les astérisques indiquent l' accumulation de transcription lg1 au PLB du primordium P6 feuille. Les flèches indiquent la base de P6 primordium. Bar indique la mesure de la base de l'ébauche à la PLB. Les barres d'échelle en A et B = 20 mm. Les barres d'échelle dans CE = 100 um. Ce chiffre a été modifié depuis la référence 6 (Droit d' auteur Société américaine des biologistes végétaux). S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
Dans cette étude, LM ARN-Seq a été utilisée pour identifier un ensemble de gènes qui sont exprimés de manière différentielle (DE) à la limite lame-gaine par rapport à d'autres parties du primordium foliaire et Ide gènes ntifier qui sont dans des mutants DE lg1-R par rapport aux frères et sœurs de type sauvage. LM ARN-Seq est une méthode de quantification de l' accumulation du transcrit dans des cellules spécifiques ou des domaines cellulaires 7. Les systèmes LM combinent un laser et un microscope avec un appareil photo numérique. tissus Sectionné est monté sur des lames et vu à travers le microscope. Le logiciel de LM comprend généralement des outils de dessin qui permettent à l'utilisateur d'esquisser une région sélectionnée pour microdissection. Les coupes laser le long de la ligne, et le tissu sélectionné est catapulté hors de la diapositive et dans un tube suspendu au-dessus de la diapositive. LM permet à l'utilisateur de microdissect domaines précis, y compris les couches de cellules spécifiques et même des cellules individuelles 8, 9. L'ARN peut alors être extrait du tissu microdissection. Par la suite, le composant ARN-Seq utilise le séquençage de la prochaine génération à la séquence des banques d' ADNc générées à partir de l'ARN extrait 10,= "xref"> 11.
Les principaux avantages de LM ARN-seq sont la capacité à quantifier l' accumulation des transcrits dans des domaines bien définis et la capacité de profiler l'ensemble transcriptome simultanément 7. La technique est particulièrement adaptée à sonder les événements précoces du développement, où la région d'intérêt est souvent microscopique. Des études antérieures ont utilisé LM combiné avec la technologie des biopuces pour étudier les processus de développement dans les plantes 9, 12, 13. ARN-Seq a l'avantage de quantifier les transcriptions à travers une large gamme dynamique, y compris les gènes de faible exprimés, et de l' information de séquence préalable est pas nécessaire 10, 11. En outre, LM ARN-Seq a le potentiel de mettre en évidence les gènes importants pour le développement qui peuvent manquer dans les écrans de mutagenèse en raison de la redondance génétique ou à la létalité de la perte-ofmutant de fonction.
Gènes importants pour le développement, tels que sheath1 étroite (NS1) et en forme de cuvette de cotyledon2 (CUC2), ont souvent des profils d'expression spécifiques d'un seul ou de quelques cellules 17, 18, 19, 20. Beaucoup sont exprimés seulement au cours des étapes précoces du développement et non pas dans l'organe mature. Lorsque des organes entiers ou de grands domaines sont analysés, ces transcrits spécifiques des cellules sont diluées et ne peuvent pas être détectées dans les analyses plus classiques. En permettant des analyses des domaines bien définis, LM ARN-Seq permet à ces gènes spécifiques de tissus à être identifiés et quantifiés.
Les facteurs cruciaux dans le succès des expériences décrites ici ont une analyse histologique approfondie qui a guidé la sélection de l'étape de développement approprié et le domaine de l'analyse, et MeasureMe précisent de domaines de cellules tissulaires pour LM. Pour veiller à ce que les domaines équivalents ont été échantillonnés pour toutes les répétitions, les tissus ont été recueillis à partir de primordiums foliaires au même stade de développement et les domaines microdissection ont été mesurés par rapport aux repères morphologiques telles que la ligule émergente (figure 2). Il est connu que certains gènes sont exprimés dans un gradient allant de la pointe à la base de la feuille. En mesurant les domaines précis, la variation due à l' échantillonnage à différents endroits le long de l'axe proximal-distal feuille a été maintenu à un minimum (figure 3A). Par microdissecting domaines de la même taille, la variation due à la différence de dilution des produits de transcription spécifiques de cellules a également été réduite (figure 3B). des sections longitudinales latérales du sommet de pousses ont été utilisées pour toutes microdissections. Ceux – ci sont des profilés qui sont perpendiculaires à l'axe de la nervure médiane marge (figure 4). En utilisant uniquement les sections qui incluent le SAM assure que les régions latérales équivalentes deprimordia foliaires sont analysés.
Dans les échantillons traités et sectionnés pour LM, le premier signe morphologique d'excroissance ligule est une bosse sur le côté adaxial en raison de divisions cellulaires périclines dans l'épiderme adaxiales (figure 1D, Figure 2). Il a été déterminé que la ligule émergente pourrait être identifié de manière fiable à plastochron 7 primordia foliaires de scène. Nous étions intéressés par les gènes exprimés dans toute la région, y compris le ligule ligule émergents et les cellules immédiatement distales qui formeront l'auricule. Afin de s'assurer que les pièces de tissus équivalents ont été faites, la bosse ligule a été utilisé comme point de repère morphologique et un rectangle de 100 um centrée sur la bosse ligule a été sélectionné pour LM (figure 2A, 2B). rectangles de taille équivalentes de pré-lame et pré-gaine ont été choisis parmi les mêmes primordia foliaires.
Les analyses des plantes mutantes ont présenté une liguleless challe différentnge; mutants lg1-R ne forment pas une ligule, donc cette fonctionnalité morphologique ne peut pas être utilisé pour sélectionner la région pour LM. Au lieu de cela, le domaine de l' accumulation de transcription lg1 dans de type sauvage primordiums foliaires a été déterminé, et une région qui engloberait ce domaine a été défini. Ces analyses préliminaires ont été effectuées sur des plants de la même plantation qui ont été utilisés pour l'analyse finale, puisque les travaux antérieurs ont montré que l'emplacement de la PLB varie en fonction des conditions de croissance. L'hybridation in situ a indiqué que les transcriptions LG1 accumulent dans la PLB de P6 primordiums foliaires (figure 1E). Nous avons choisi un domaine 400-900 um à partir de la base de la primordia feuille qui englobait le domaine d'expression lg1 (rectangles violets, figure 2A) et capturé ces régions équivalentes de type sauvage et les plantes lg1-R. Pour réduire au minimum la variation des conditions de fond et de croissance génétique lorsque l'on compare transcriptaset accumulation dans lg1-R et des plantes de type sauvage, ségrégation des familles de mutants et frères et sœurs de type sauvage ont été utilisés.
Protocol
Representative Results
Discussion
La conception expérimentale est un facteur critique dans des expériences d'ARN-seq. Les principales considérations sont le domaine précis (s) et le stade de développement (s) à analyser, et quelles comparaisons seront faites. Il est crucial de penser en termes de comparaisons, puisque la sortie est généralement une liste de gènes qui sont DE entre deux ou plusieurs conditions. Comme avec toutes les expériences, il est important de modifier qu'une seule variable à la fois. Par exemple, lorsque l'on…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Les auteurs remercient S. Merlu pour collaboration continue et de stimuler les discussions sur le développement ligule. Ce travail est soutenu par des subventions de la Fondation nationale des sciences MCB 1.052.051 et IOS-1848478.
Materials
| Diethyl pyrocarbonate | Sigma-Aldrich | 159220 | Used for RNase treatment of solutions |
| Razor blades | Electron Microscopy Sciences | 72000 | |
| RNase Zap | Sigma-Aldrich | R2020-250ML | RNase decontamination solution |
| Ethanol absolute 200 proof | Fisher Scientific | BP28184 | |
| Acetic acid, glacial | Sigma-Aldrich | A6283 | |
| Glass vials – 22mL | VWR | 470206-384 | |
| Xylenes, histological grade | Sigma-Aldrich | 534056-4L | |
| Paraplast plus | Sigma-Aldrich | P3683-1KG | |
| Disposable base molds, 15 x 15 x 5mm | VWR | 15154-072 | |
| Embedding rings | VWR | 15154-303 | |
| Silica gel packets | Electron Microscopy Sciences | 71206-01 | Desiccant for storage of paraffin blocks |
| Oven | Fisher Scientific | 15-103-0503 | Oven must maintain temperature of 60° C |
| Paraffin embedding station | Leica | EG1160 | |
| Microtome | Leica | RM2235 | |
| Slide warmer | Electron Microscopy Sciences | 71317-10 | |
| Coplin jars | Electron Microscopy Sciences | 70316-02 | |
| Laser microdissector | Zeiss | ||
| KIMWIPES™ Delicate Task Wipers | Kimberly-Clark Professional | 34120 | Lint-free wipes for wicking excess solutions from microscope slides |
| Membrane Slide 1.0 PEN | Zeiss | 415190-9041-000 | Slides for laser microdissection |
| Adhesive Cap 200 opaque | Zeiss | 415190-9181-000 | Tubes for laser microdissection |
| PicoPure RNA Isolation Kit | ThermoFisher Scientific | KIT0204 |
References
- Becraft, P. W., Bongard-Pierce, D. K., Sylvester, A. W., Poethig, R. S., Freeling, M. The liguleless-1 gene acts tissue specifically in maize leaf development. Dev Biol. 141 (1), 220-232 (1990).
- Sylvester, A. W., Cande, W. Z., Freeling, M. Division and differentiation during normal and liguleless-1 maize leaf development. Development. 110 (3), 985-1000 (1990).
- Moreno, M. A., Harper, L. C., Krueger, R. W., Dellaporta, S. L., Freeling, M. liguleless1 encodes a nuclear-localized protein required for induction of ligules and auricles during maize leaf organogenesis. Genes Dev. 11 (5), 616-628 (1997).
- Emerson, R. A. The inheritance of the ligule and auricle of corn leaves. Neb. Agr. Exp. Sta. An. Rep. 25, 81-85 (1912).
- Moon, J., Candela, H., Hake, S. The Liguleless narrow mutation affects proximal-distal signaling and leaf growth. Development. 140 (2), 405-412 (2013).
- Johnston, R., et al. Transcriptomic Analyses Indicate That Maize Ligule Development Recapitulates Gene Expression Patterns That Occur during Lateral Organ Initiation. Plant Cell. 26 (12), 4718-4732 (2014).
- Schmid, M. W., et al. A powerful method for transcriptional profiling of specific cell types in eukaryotes: laser-assisted microdissection and RNA sequencing. PLoS One. 7 (1), e29685 (2012).
- Kerk, N. M., Ceserani, T., Tausta, S. L., Sussex, I. M., Nelson, T. M. Laser capture microdissection of cells from plant tissues. Plant Physiol. 132 (1), 27-35 (2003).
- Nakazono, M., Qiu, F., Borsuk, L. A., Schnable, P. S. Laser-capture microdissection, a tool for the global analysis of gene expression in specific plant cell types: identification of genes expressed differentially in epidermal cells or vascular tissues of maize. Plant Cell. 15 (3), 583-596 (2003).
- Marioni, J. C., Mason, C. E., Mane, S. M., Stephens, M., Gilad, Y. RNA-seq: an assessment of technical reproducibility and comparison with gene expression arrays. Genome Res. 18 (9), 1509-1517 (2008).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nat Rev Genet. 10 (1), 57-63 (2009).
- Brooks, L., et al. Microdissection of shoot meristem functional domains. PLoS Genet. 5 (5), e1000476 (2009).
- Cai, S., Lashbrook, C. C. Stamen abscission zone transcriptome profiling reveals new candidates for abscission control: enhanced retention of floral organs in transgenic plants overexpressing Arabidopsis ZINC FINGER PROTEIN2. Plant Physiol. 146 (3), 1305-1321 (2008).
- Li, P., et al. The developmental dynamics of the maize leaf transcriptome. Nat Genet. 42 (12), 1060-1067 (2010).
- Eveland, A. L., et al. Regulatory modules controlling maize inflorescence architecture. Genome Res. 24 (3), 431-443 (2014).
- Takacs, E. M., et al. Ontogeny of the maize shoot apical meristem. Plant Cell. 24 (8), 3219-3234 (2012).
- Aida, M., Ishida, T., Tasaka, M. Shoot apical meristem and cotyledon formation during Arabidopsis embryogenesis: interaction among the CUP-SHAPED COTYLEDON and SHOOT MERISTEMLESS genes. Development. 126 (8), 1563-1570 (1999).
- Ishida, T., Aida, M., Takada, S., Tasaka, M. Involvement of CUP-SHAPED COTYLEDON genes in gynoecium and ovule development in Arabidopsis thaliana. Plant Cell Physiol. 41 (1), 60-67 (2000).
- Takada, S., Hibara, K., Ishida, T., Tasaka, M. The CUP-SHAPED COTYLEDON1 gene of Arabidopsis regulates shoot apical meristem formation. Development. 128 (7), 1127-1135 (2001).
- Nardmann, J., Ji, J., Werr, W., Scanlon, M. J. The maize duplicate genes narrow sheath1 and narrow sheath2 encode a conserved homeobox gene function in a lateral domain of shoot apical meristems. Development. 131 (12), 2827-2839 (2004).
- Bonner, W. A., Hulett, H. R., Sweet, R. G., Herzenberg, L. A. Fluorescence activated cell sorting. Rev Sci Instrum. 43 (3), 404-409 (1972).
- Birnbaum, K., et al. A gene expression map of the Arabidopsis root. Science. 302 (5652), 1956-1960 (2003).
- Brady, S. M., et al. A high-resolution root spatiotemporal map reveals dominant expression patterns. Science. 318 (5851), 801-806 (2007).
- Carter, A. D., Bonyadi, R., Gifford, M. L. The use of fluorescence-activated cell sorting in studying plant development and environmental responses. Int J Dev Biol. 57 (6-8), 545-552 (2013).
- Ruzin, S. E. . Plant microtechnique and microscopy. , (1999).
- Jackson, D., Veit, B., Hake, S. Expression of maize KNOTTED1 related homeobox genes in the shoot apical meristem predicts patterns of morphogenesis in the vegetative shoot. Development. 120, 405-413 (1994).
- Javelle, M., Marco, C. F., Timmermans, M. In situ hybridization for the precise localization of transcripts in plants. J Vis Exp. (57), e3328 (2011).
- Day, R. C., McNoe, L., Macknight, R. C. Evaluation of global RNA amplification and its use for high-throughput transcript analysis of laser-microdissected endosperm. Int J Plant Genomics. , 61028 (2007).

