レーザーマイクロダイセクションRNA-のSeqのための実験デザイン:トウモロコシ葉の開発の分析からの教訓
Summary
Many developmentally important genes have cell- or tissue-specific expression patterns. This paper describes LM RNA-seq experiments to identify genes that are differentially expressed at the maize leaf blade-sheath boundary and in lg1-R mutants compared to wild-type. The experimental considerations discussed here apply to transcriptomic analyses of other developmental phenomena.
Abstract
開発に重要な役割を持つ遺伝子は、しばしば、空間的および/または一時的に制限された発現パターンを有します。多くの場合、これらの遺伝子転写物は検出されないか、または差動で全体の植物器官のトランスクリプトーム解析に(DE)表現として識別されていません。レーザーマイクロダイセクションRNA-のSeq(LM RNA-のSeq)は、特定の発達のドメインにDEされる遺伝子を同定するための強力なツールです。しかし、顕微解剖と比較するための細胞ドメインの選択、およびmicrodissectionsの精度は、実験の成功に不可欠です。ここでは、2つの例は、トランスクリプトミクス実験のための設計上の考慮事項を説明します。 DEは、トウモロコシの葉の近位-遠位軸、及びliguleless1-R(LG1-R)でのDE遺伝子を同定するための第二の実験に沿っている遺伝子を同定するためのLM-RNA配列分析 変異体は、野生型と比較しました。これらの実験の成功に貢献の重要な要素は、組織学的およびSIで詳述しました。TUのハイブリダイゼーションは、同等の発生段階の葉原基の選択、分析されるべき領域の顕微解剖のための領域を選択するための形態学的なランドマークの使用を分析し、正確に測定されたドメインの顕微解剖。本稿では、LM、RNAのSeqによって発達ドメインの分析のための詳細なプロトコルを提供します。ここに示されたデータは、顕微解剖のために選択された領域が得られた結果にどのように影響するかを示しています。
Introduction
それは遺伝的解剖( 図1A)の影響を受けやすいブレードとシースとの間に明確な境界を持っているとして、トウモロコシの葉は、形態形成の間に発達フィールドの形成を研究するための理想的なモデルです。葉の開発、より小さなセルの線形バンド、preliguleバンド(PLB)の初期段階で、事前ブレードおよびプレシースドメインに葉原基を細分化します。フリンジのような小舌と三角耳介は、PLB( 図1A、C、D)から発生します。遺伝子スクリーニングは、ブレード・シース境界を崩壊させる変異を同定しました。例えば、劣性liguleless1(LG1)の変異は、小舌と耳介1、2、3、4( 図1B)を削除します。 in situハイブリダイゼーションLG1転写物がPに蓄積することを明らかにしましたそれ小舌の開発5、6のための優れたマーカー( 図1E)作りLBと新興小舌、。
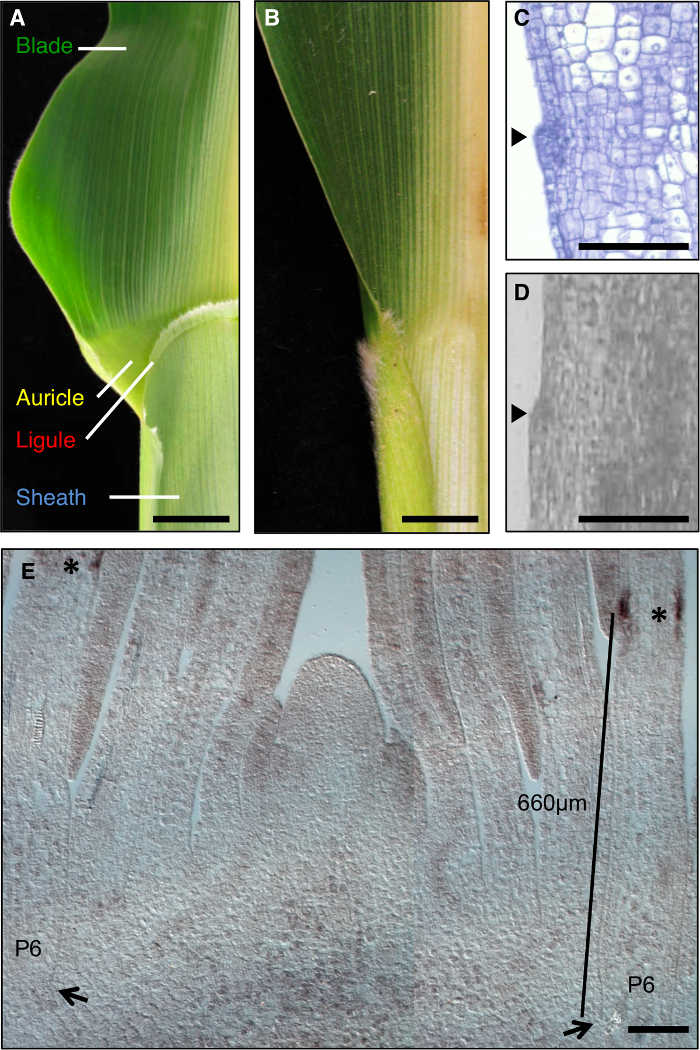
図1:野生型と liguleless1-R トウモロコシの葉。 (A)小舌と耳介の構造を示す成熟した野生型の葉のブレードシース境界領域。小舌と耳介の構造が存在しないことを示す成熟liguleless1-Rの葉の(B)ブレード・シース界領域。 AとBでの葉は中肋に沿って半分にカットされています。 (C)野生型の葉の原基の縦断面図。サンプルを処理し、組織学的分析のために染色されています。開始小舌は葉(矢じり)の面から突出するバンプとして明らかです。 (D)縦宗派野生型の葉の原基を介してイオン。本文中に記載されているように、サンプルは、LMのために処理されています。アローヘッドは、小舌を開始示しています。シュート頂側面縦断面のin situハイブリダイゼーション(E)LG1。アスタリスクは、P6の葉原基のPLBでLG1転写産物の蓄積を示しています。矢印は、P6原基のベースを示しています。バーでは、PLBへの原基のベースから測定値を示しています。 AとBの中のスケールバーは20ミリメートルを=。 CEでのスケールバー=100μmです。この図は、参照6(植物生物学者の著作権協会)から変更されています。 この図の拡大版をご覧になるにはこちらをクリックしてください。
本研究では、LM RNA-のSeqを差動葉原基の他の部分に、IDEにブレードシース境界相対で(DE)に発現する遺伝子群を同定しました野生型の兄弟に比べてLG1-R変異体でDEですntify遺伝子。 LM-RNAのSeqは、特定の細胞または細胞のドメイン7に転写物の蓄積を定量化する方法です。直動システムは、レーザーやデジタルカメラと顕微鏡を組み合わせます。切片組織は、スライド上にマウントし、顕微鏡を通して見られます。 LMソフトウェアは、典型的には、ユーザが顕微解剖のために任意の選択した領域を概説することを可能にする描画ツールが含まれています。線に沿って、レーザーカット、および選択された組織は、スライドをオフに放り出さし、スライド上に懸架チューブにされています。 LMは、ユーザが特定の細胞層を含む正確なドメイン、さらには単一細胞8,9を顕微解剖することができます。次いで、RNAを顕微解剖組織から抽出することができます。その後、RNA-のSeq成分が抽出されたRNA 10から生成されたcDNAライブラリーを配列決定するために、次世代シーケンシングを利用して、= "外部参照"> 11。
LM RNA-seqのの主な利点は、転写物の正確に定義されたドメイン内の蓄積と同時に全体のトランスクリプトーム7のプロファイルを作成する能力を定量する能力です。技術は、関心領域は、多くの場合、微小な初期発生イベントをプローブに特に適しています。以前の研究は、植物9の発生過程を研究するためのマイクロアレイ技術、12、13と組み合わさLMを利用しています。 RNA-SEQが低発現される遺伝子を含む、広いダイナミックレンジにわたって転写を定量化するという利点を有し、かつ事前配列情報は、11を 10に必要とされません。また、LM RNA-のSeqは、遺伝子冗長性または喪失型の致死性に突然変異誘発画面でミスの可能性があることを発育に重要な遺伝子を強調するために可能性を秘めています機能変異体。
このような狭いsheath1(NS1)とカップ状cotyledon2(CUC2)などの発達に重要な遺伝子は、多くの場合、ただ1つまたは少数の細胞17の特異的な発現パターンを持っている18、19、20。多くは成熟した臓器に発生初期の段階でのみ発現し、されていません。全体の臓器や大規模なドメインを分析すると、これらの細胞特異的転写産物が希釈され、より多くの従来の分析で検出されない場合があります。正確に定義されたドメインの分析を可能にすることにより、LM RNA-のSeqは、これらの組織特異的遺伝子を同定し、定量化することを可能にします。
ここに記載した実験の成功に重要な要因は、分析のための適切な発達段階とドメインの選択を導いた徹底的な組織学的分析、および正確なmeasuremeましたLMための細胞組織のドメインのヌクレオチド。同等のドメインは、すべての複製のためにサンプリングしたことを確認するために、組織は、同じ発達段階での葉原基から回収し、顕微解剖されたドメインは、新興小舌( 図2)のような形態学的なランドマークと比較して測定しました。いくつかの遺伝子は、葉の基部の先端からの勾配で発現することが知られています。葉の近位-遠位軸に沿った異なる場所からサンプリングの正確なドメイン、変化を測定することにより、最小値( 図3A)に維持しました。同じサイズのドメインをmicrodissectingすることにより、細胞特異的転写物の希釈差によるばらつきも( 図3B)に減少しました。茎頂の側面縦断面は全てmicrodissectionsのために使用しました。これらは、( 図4)中肋マージンの軸に垂直な部分です。 SAMを含むセクションのみを使用することと同等の横方向の領域を確保し葉原基が分析されます。
処理され、LMのために切片サンプルでは、小舌伸長の最初の形態学的兆候が原因向軸表皮( 図1D、 図2)における周縁の細胞分裂に向軸側のバンプです。新興小舌を確実plastochron 7段葉原基で識別することができることを判断しました。私たちは、新興小舌と耳介を形成することになるすぐ遠位の細胞を含む全小舌の領域で発現する遺伝子、に興味を持っていました。同等の組織の選択が行われたことを確実にするために、小舌のバンプは、形態学的ランドマークとして使用し、小舌バンプを中心100μmの矩形はLM( 図2A、2B)のために選択しました。前ブレードとプレシースの同等サイズの矩形が同じ葉原基から選択されました。
liguleless変異植物の分析は異なるchalleを発表しましたNGE; LG1-R変異体は、したがって、この形態学的特徴は、LMための領域を選択するために使用することができなかった、小舌を形成しません。代わりに、野生型の葉の原基におけるLG1転写物の蓄積の領域を決定し、このドメインを含むであろう領域を定義しました。以前の研究は、PLBの位置は、成長条件に依存して変化することを示したので、これらの予備的な分析は、最終的な分析のために使用したのと同じ植え付けから苗木上で実施しました。 in situハイブリダイゼーションはLG1転写物がPLB P6の葉原基( 図1E)に蓄積することが示されました。私たちは、LG1式(紫の長方形、 図2A)のドメインを包含し、野生型とLG1-R植物からこれらの同等の領域を撮影した葉原基のベースからドメイン400〜900ミクロンを選択しました。 transcripを比較した場合、遺伝的背景と成長条件の変化を最小限に抑えるために、LG1-R及び野生型植物におけるT蓄積は、変異体と野生型同胞の分離ファミリーを使用しました。
Protocol
Representative Results
Discussion
実験計画は、RNA-seqの実験において重要な要因です。重要な考慮事項は、正確なドメイン(複数可)および発生段階(複数可)を分析するために、どのような比較が行われるです。出力は、典型的には2つ以上の条件との間でDEされている遺伝子のリストであるので、比較の観点から考えることが重要です。すべての実験と同様に、一度に1つの変数を変更することが重要です。例えば、比較す?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
著者はコラボレーションを継続的かつ小舌開発について議論を刺激するためのS.メルルーサに感謝します。この作品は、国立科学財団助成MCB 1052051およびIOS-1848478によってサポートされています。
Materials
| Diethyl pyrocarbonate | Sigma-Aldrich | 159220 | Used for RNase treatment of solutions |
| Razor blades | Electron Microscopy Sciences | 72000 | |
| RNase Zap | Sigma-Aldrich | R2020-250ML | RNase decontamination solution |
| Ethanol absolute 200 proof | Fisher Scientific | BP28184 | |
| Acetic acid, glacial | Sigma-Aldrich | A6283 | |
| Glass vials – 22mL | VWR | 470206-384 | |
| Xylenes, histological grade | Sigma-Aldrich | 534056-4L | |
| Paraplast plus | Sigma-Aldrich | P3683-1KG | |
| Disposable base molds, 15 x 15 x 5mm | VWR | 15154-072 | |
| Embedding rings | VWR | 15154-303 | |
| Silica gel packets | Electron Microscopy Sciences | 71206-01 | Desiccant for storage of paraffin blocks |
| Oven | Fisher Scientific | 15-103-0503 | Oven must maintain temperature of 60° C |
| Paraffin embedding station | Leica | EG1160 | |
| Microtome | Leica | RM2235 | |
| Slide warmer | Electron Microscopy Sciences | 71317-10 | |
| Coplin jars | Electron Microscopy Sciences | 70316-02 | |
| Laser microdissector | Zeiss | ||
| KIMWIPES™ Delicate Task Wipers | Kimberly-Clark Professional | 34120 | Lint-free wipes for wicking excess solutions from microscope slides |
| Membrane Slide 1.0 PEN | Zeiss | 415190-9041-000 | Slides for laser microdissection |
| Adhesive Cap 200 opaque | Zeiss | 415190-9181-000 | Tubes for laser microdissection |
| PicoPure RNA Isolation Kit | ThermoFisher Scientific | KIT0204 |
References
- Becraft, P. W., Bongard-Pierce, D. K., Sylvester, A. W., Poethig, R. S., Freeling, M. The liguleless-1 gene acts tissue specifically in maize leaf development. Dev Biol. 141 (1), 220-232 (1990).
- Sylvester, A. W., Cande, W. Z., Freeling, M. Division and differentiation during normal and liguleless-1 maize leaf development. Development. 110 (3), 985-1000 (1990).
- Moreno, M. A., Harper, L. C., Krueger, R. W., Dellaporta, S. L., Freeling, M. liguleless1 encodes a nuclear-localized protein required for induction of ligules and auricles during maize leaf organogenesis. Genes Dev. 11 (5), 616-628 (1997).
- Emerson, R. A. The inheritance of the ligule and auricle of corn leaves. Neb. Agr. Exp. Sta. An. Rep. 25, 81-85 (1912).
- Moon, J., Candela, H., Hake, S. The Liguleless narrow mutation affects proximal-distal signaling and leaf growth. Development. 140 (2), 405-412 (2013).
- Johnston, R., et al. Transcriptomic Analyses Indicate That Maize Ligule Development Recapitulates Gene Expression Patterns That Occur during Lateral Organ Initiation. Plant Cell. 26 (12), 4718-4732 (2014).
- Schmid, M. W., et al. A powerful method for transcriptional profiling of specific cell types in eukaryotes: laser-assisted microdissection and RNA sequencing. PLoS One. 7 (1), e29685 (2012).
- Kerk, N. M., Ceserani, T., Tausta, S. L., Sussex, I. M., Nelson, T. M. Laser capture microdissection of cells from plant tissues. Plant Physiol. 132 (1), 27-35 (2003).
- Nakazono, M., Qiu, F., Borsuk, L. A., Schnable, P. S. Laser-capture microdissection, a tool for the global analysis of gene expression in specific plant cell types: identification of genes expressed differentially in epidermal cells or vascular tissues of maize. Plant Cell. 15 (3), 583-596 (2003).
- Marioni, J. C., Mason, C. E., Mane, S. M., Stephens, M., Gilad, Y. RNA-seq: an assessment of technical reproducibility and comparison with gene expression arrays. Genome Res. 18 (9), 1509-1517 (2008).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nat Rev Genet. 10 (1), 57-63 (2009).
- Brooks, L., et al. Microdissection of shoot meristem functional domains. PLoS Genet. 5 (5), e1000476 (2009).
- Cai, S., Lashbrook, C. C. Stamen abscission zone transcriptome profiling reveals new candidates for abscission control: enhanced retention of floral organs in transgenic plants overexpressing Arabidopsis ZINC FINGER PROTEIN2. Plant Physiol. 146 (3), 1305-1321 (2008).
- Li, P., et al. The developmental dynamics of the maize leaf transcriptome. Nat Genet. 42 (12), 1060-1067 (2010).
- Eveland, A. L., et al. Regulatory modules controlling maize inflorescence architecture. Genome Res. 24 (3), 431-443 (2014).
- Takacs, E. M., et al. Ontogeny of the maize shoot apical meristem. Plant Cell. 24 (8), 3219-3234 (2012).
- Aida, M., Ishida, T., Tasaka, M. Shoot apical meristem and cotyledon formation during Arabidopsis embryogenesis: interaction among the CUP-SHAPED COTYLEDON and SHOOT MERISTEMLESS genes. Development. 126 (8), 1563-1570 (1999).
- Ishida, T., Aida, M., Takada, S., Tasaka, M. Involvement of CUP-SHAPED COTYLEDON genes in gynoecium and ovule development in Arabidopsis thaliana. Plant Cell Physiol. 41 (1), 60-67 (2000).
- Takada, S., Hibara, K., Ishida, T., Tasaka, M. The CUP-SHAPED COTYLEDON1 gene of Arabidopsis regulates shoot apical meristem formation. Development. 128 (7), 1127-1135 (2001).
- Nardmann, J., Ji, J., Werr, W., Scanlon, M. J. The maize duplicate genes narrow sheath1 and narrow sheath2 encode a conserved homeobox gene function in a lateral domain of shoot apical meristems. Development. 131 (12), 2827-2839 (2004).
- Bonner, W. A., Hulett, H. R., Sweet, R. G., Herzenberg, L. A. Fluorescence activated cell sorting. Rev Sci Instrum. 43 (3), 404-409 (1972).
- Birnbaum, K., et al. A gene expression map of the Arabidopsis root. Science. 302 (5652), 1956-1960 (2003).
- Brady, S. M., et al. A high-resolution root spatiotemporal map reveals dominant expression patterns. Science. 318 (5851), 801-806 (2007).
- Carter, A. D., Bonyadi, R., Gifford, M. L. The use of fluorescence-activated cell sorting in studying plant development and environmental responses. Int J Dev Biol. 57 (6-8), 545-552 (2013).
- Ruzin, S. E. . Plant microtechnique and microscopy. , (1999).
- Jackson, D., Veit, B., Hake, S. Expression of maize KNOTTED1 related homeobox genes in the shoot apical meristem predicts patterns of morphogenesis in the vegetative shoot. Development. 120, 405-413 (1994).
- Javelle, M., Marco, C. F., Timmermans, M. In situ hybridization for the precise localization of transcripts in plants. J Vis Exp. (57), e3328 (2011).
- Day, R. C., McNoe, L., Macknight, R. C. Evaluation of global RNA amplification and its use for high-throughput transcript analysis of laser-microdissected endosperm. Int J Plant Genomics. , 61028 (2007).

