Experimental Design for Laser mikrodisseksjon RNA-Seq: Lærdommer fra en analyse av Mais Leaf Development
Summary
Many developmentally important genes have cell- or tissue-specific expression patterns. This paper describes LM RNA-seq experiments to identify genes that are differentially expressed at the maize leaf blade-sheath boundary and in lg1-R mutants compared to wild-type. The experimental considerations discussed here apply to transcriptomic analyses of other developmental phenomena.
Abstract
Gener med viktige roller i utviklingen har ofte romlig og / eller tidsmessig begrenset uttrykk mønstre. Ofte disse genet transkripsjoner ikke blir oppdaget eller ikke identifisert som forskjellig uttrykt (DE) i transcriptomic analyser av hele anlegget organer. Laser mikrodisseksjon RNA-Seq (LM RNA-Seq) er et kraftig verktøy for å identifisere gener som er DE i spesifikke utviklings domener. Men til valg av cellulære domener microdissect og sammenligne, og nøyaktigheten av microdissections er avgjørende for å lykkes i forsøkene. Her to eksempler illustrerer design hensyn til transcriptomics eksperimenter; en LM RNA-seq analyse for å identifisere gener som er de langs mais blad proksimale-distale akse, og et andre eksperiment for å identifisere gener som er i DE liguleless1-R (LG1-R) mutanter sammenlignet med vill-type. Sentrale elementer som bidro til suksessen til disse eksperimentene ble rede histologiske og i situ hybridisering analyser for regionen som skal bli analysert, valg av blad primordia ved ekvivalente utviklingsstadier, bruk av morfologiske landemerker for å velge områder for mikrodisseksjon, og mikrodisseksjon av nøyaktig målt domener. Denne artikkelen gir en detaljert protokoll for analyse av utviklings domener ved LM RNA-Seq. Dataene presentert her illustrerer hvordan regionen valgt for mikrodisseksjon vil påvirke de oppnådde resultater.
Introduction
Mais blad er en ideell modell for å studere dannelsen av utviklingsfelt under morphogenesis, som den har en tydelig grense mellom bladet og skjede som er mottagelig for genetisk disseksjon (figur 1A). Under de tidlige stadier av blad utvikling, en lineær band av mindre celler, preligule band (PLB), subdivides bladet primordium inn i pre-blad og pre-kappe domener. En pannelugg-lignende ligule og trekantede ytterøret utvikles fra PLB (Figur 1A, C, D). Genetiske skjermer har identifisert mutasjoner som forstyrrer blad kappe grense. For eksempel recessive liguleless1 (LG1) mutasjoner slette ligule og ytterøret 1, 2, 3, 4 (figur 1B). In situ hybridisering viste at LG1 transkripsjon akkumulerer på PLB og nye ligule, noe som gjør det til et utmerket markør for ligule utvikling 5, 6 (figur 1E).
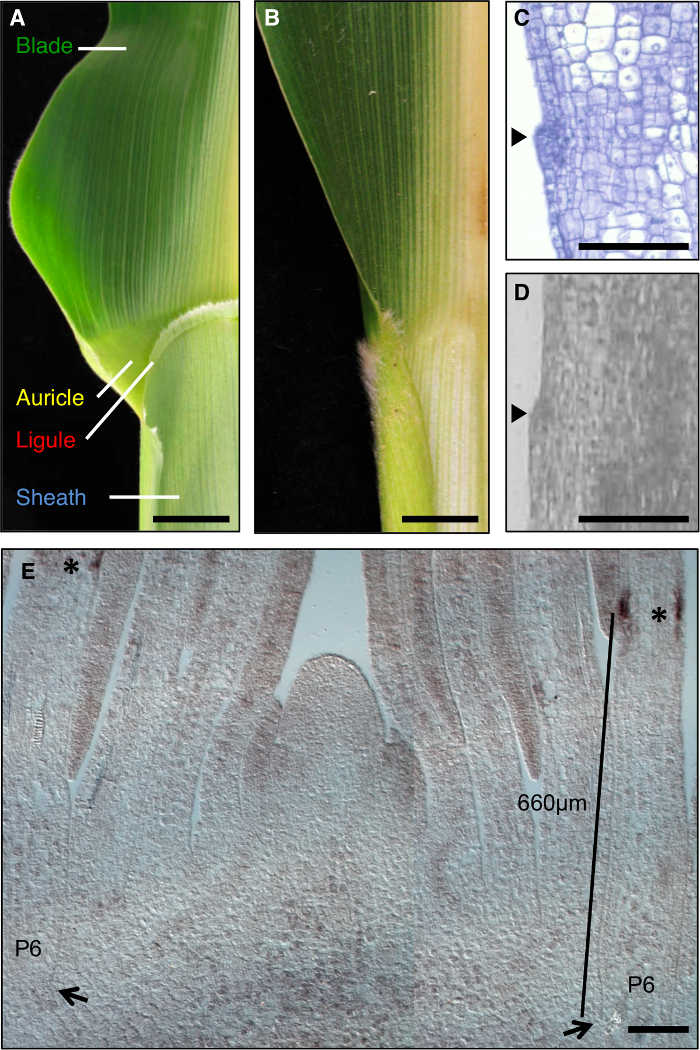
Figur 1: Wild-type og liguleless1-R mais blader. (A) Blade-kappe grenseområdet av modent villtype blad som viser ligule og aurikkel strukturer. (B) Blade-slire grenseområdet av modne liguleless1-R blad som viser fravær av ligule og auricle strukturer. Blader i A og B har blitt halvert langs midrib. (C) Lengdesnitt gjennom villtype blad primordium. Prøve har blitt behandlet og farget for histologisk analyse. Den initierende ligule er tydelig som en brak stikker ut fra planet til bladet (pilspiss). (D) Longitudinal sektion gjennom villtype blad primordium. Prøve er blitt behandlet for LM som beskrevet i teksten. Arrowhead viser initiere ligule. (E) LG1 in situ hybridisering av shoot apex lateral lengdesnitt. Stjernene viser LG1 transkripsjon opphopning på PLB av P6 blad primordium. Piler indikerer base av P6 primordium. Linjen indikerer målingen fra bunnen av primordium til PLB. Skala barer i A og B = 20 mm. Skala barer i CE = 100 mikrometer. Dette tallet har blitt modifisert fra referanse 6 (Copyright American Society of Plant Biologer). Klikk her for å se en større versjon av dette tallet.
I denne studien ble LM RNA-Seq anvendes til å identifisere en rekke gener som er differensielt uttrykt (DE) ved blad-kappe grense i forhold til andre deler av bladet primordium og til Ide ntify gener som er i DE LG1-R-mutanter i forhold til villtype-søsken. LM RNA-Seq er en metode for å kvantifisere transkripsjonen akkumulering i spesifikke celler eller cellulære domener 7. LM systemer kombinerer en laser og et mikroskop med et digitalt kamera. Kåret vev er montert på objektglass og sett gjennom mikroskop. LM programvaren inneholder vanligvis tegneverktøy som tillater brukeren å skissere noen utvalgte region for mikrodisseksjon. Laser kutt langs linjen, og det valgte vevet er slynget ut av lysbildet og inn i et rør suspendert over raset. LM tillater brukeren å microdissect presise domener, inkludert spesifikke cellelagene og til og med enkeltceller 8, 9. RNA kan deretter ekstraheres fra microdissected vev. Deretter benytter RNA-Seq komponent neste generasjons sekvense å sekvensere cDNA bibliotek generert fra den utpakkede RNA 10,= "xref"> 11.
Viktige fordeler med LM RNA-seq er evnen til å kvantifisere transkripsjon opphopning i presist definerte domener og kapasitet til å profilere hele transkriptomet samtidig 7. Teknikken er spesielt egnet til sondering tidlige utviklings hendelser hvor regionen av interesse er ofte mikroskopisk. Tidligere studier har benyttet LM kombinert med mikromatriser å studere utviklingsmessige prosesser i planter 9, 12, 13. RNA-Seq har fordelen av å kvantifisere transkripsjoner over et bredt dynamisk område, inkludert lav uttrykte gener, og før sekvensinformasjon er ikke påkrevd 10, 11. Videre har LM RNA-Seq potensial til å markere utviklingsmessig viktige gener som kan være savnet i mutagenese skjermer på grunn av genetisk redundans eller til dødelighet av tapet-of-funksjon mutant.
Utviklingsmessig viktige gener, for eksempel smale sheath1 (ns1) og cup-formet cotyledon2 (CUC2), har ofte spesifikke uttrykk mønstre av bare ett eller noen få celler 17, 18, 19, 20. Mange er uttrykt bare i løpet av tidlige utviklingsstadier, og ikke i den modne organ. Når hele organer eller store domener er analysert, er disse cellespesifikke transkripsjoner utvannet og kan ikke påvises i mer konvensjonelle analyser. Ved å tillate analyser av nøyaktig definerte områder, muliggjør LM RNA-Seq Disse vevs-spesifikke gener kan identifiseres og kvantifiseres.
Avgjørende faktorene i suksess for eksperimentene beskrevet her var en grundig histologisk analyse som guidet valg av riktig utviklingsstadiet og domene for analyse, og nøyaktig måling avnt av celle-vev domener for LM. For å sikre at tilsvarende domener ble prøvetatt for alle replikater, ble vev hentet fra bladet primordia på samme utviklingsstadiet og de microdissected domenene ble målt i forhold til morfologiske landemerker, for eksempel den nye ligule (figur 2). Det er kjent at noen gener som skal uttrykkes i en gradient fra spissen til foten av bladet. Ved å måle nøyaktige domener, variasjoner som skyldes sampling fra forskjellige steder langs bladet proksimale-distale akse ble holdt på et minimum (figur 3A). Ved microdissecting domener av samme størrelse, for å variasjon på grunn av differensial fortynning av celle-spesifikke transkripter ble også redusert (figur 3B). Lateral lengdesnitt av skyte apex ble brukt for alle microdissections. Disse er deler som er vinkelrett på midrib-margin akse (figur 4). Ved hjelp av bare deler som inkluderer SAM sikrer at tilsvarende side regioneneblad primordia analyseres.
I prøver bearbeidet og seksjonert for LM, den første morfologiske tegn på ligule utvekst er en kul på adaxial side grunnet periclinal celledelinger i adaxial epidermis (figur 1D, figur 2). Det ble bestemt at den nye ligule kan bli pålitelig identifiseres ved plastochron 7 scenen blad primordia. Vi var interessert i genene kommer til uttrykk i hele ligule regionen, inkludert den nye ligule og cellene umiddelbart distale som vil danne atriet. For å sikre at tilsvarende vev valg ble gjort, ble det ligule bump anvendt som en morfologisk fjell og et 100 um rektangel sentrert på ligule bump ble valgt for LM (figur 2A, 2B). Tilsvarende store rektangler av pre-blad og pre-slire ble valgt ut fra de samme blad primordia.
Analyser av liguleless mutant planter presentert en annen Challenge; LG1-R mutanter danner ikke et ligule, derfor er dette morfologiske funksjonen kan ikke brukes til å velge området for LM. I stedet ble domenet til LG1 transkript akkumulering i vill-type blad primordia bestemmes, og en region som ville omfatte dette domenet ble definert. Disse preliminære analyser ble utført på frøplanter fra det samme plante som ble anvendt for den endelige analysen, siden tidligere arbeid har vist at plasseringen av PLB varierer avhengig av vekstbetingelsene. In situ hybridisering indikerte at LG1 transkripsjoner akkumuleres i PLB av P6 blad primordia (figur 1E). Vi har valgt et domene 400-900 um fra undersiden av bladet primordia som omfattet domenet til LG1 uttrykket (purpur rektangler, figur 2A) og fanges opp de tilsvarende områder fra villtype og LG1-R planter. For å minimere variasjon i genetiske bakgrunn og vekstvilkår når man sammenligner transcript akkumulering i LG1-R og planter av vill type, segregere familier av mutanter og villtype-søsken ble anvendt.
Protocol
Representative Results
Discussion
Eksperimentell design er en kritisk faktor i RNA-seq eksperimenter. Viktige betraktninger er den nøyaktige domene (r) og utviklingstrinn (e) som skal analyseres, og hvilke sammenligninger vil bli gjort. Det er viktig å tenke i form av sammenligninger, fordi produksjonen er vanligvis en liste av gener som er de mellom to eller flere betingelser. Som med alle eksperimenter, er det viktig å forandre bare en variabel om gangen. For eksempel, når man sammenligner ulike blad domener, går på samme alder og utviklingsstad…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Forfatterne takker S. Hake for løpende samarbeid og stimulerende diskusjoner om ligule utvikling. Dette arbeidet er støttet av National Science Foundation Grants MCB 1052051 og IOS-1848478.
Materials
| Diethyl pyrocarbonate | Sigma-Aldrich | 159220 | Used for RNase treatment of solutions |
| Razor blades | Electron Microscopy Sciences | 72000 | |
| RNase Zap | Sigma-Aldrich | R2020-250ML | RNase decontamination solution |
| Ethanol absolute 200 proof | Fisher Scientific | BP28184 | |
| Acetic acid, glacial | Sigma-Aldrich | A6283 | |
| Glass vials – 22mL | VWR | 470206-384 | |
| Xylenes, histological grade | Sigma-Aldrich | 534056-4L | |
| Paraplast plus | Sigma-Aldrich | P3683-1KG | |
| Disposable base molds, 15 x 15 x 5mm | VWR | 15154-072 | |
| Embedding rings | VWR | 15154-303 | |
| Silica gel packets | Electron Microscopy Sciences | 71206-01 | Desiccant for storage of paraffin blocks |
| Oven | Fisher Scientific | 15-103-0503 | Oven must maintain temperature of 60° C |
| Paraffin embedding station | Leica | EG1160 | |
| Microtome | Leica | RM2235 | |
| Slide warmer | Electron Microscopy Sciences | 71317-10 | |
| Coplin jars | Electron Microscopy Sciences | 70316-02 | |
| Laser microdissector | Zeiss | ||
| KIMWIPES™ Delicate Task Wipers | Kimberly-Clark Professional | 34120 | Lint-free wipes for wicking excess solutions from microscope slides |
| Membrane Slide 1.0 PEN | Zeiss | 415190-9041-000 | Slides for laser microdissection |
| Adhesive Cap 200 opaque | Zeiss | 415190-9181-000 | Tubes for laser microdissection |
| PicoPure RNA Isolation Kit | ThermoFisher Scientific | KIT0204 |
References
- Becraft, P. W., Bongard-Pierce, D. K., Sylvester, A. W., Poethig, R. S., Freeling, M. The liguleless-1 gene acts tissue specifically in maize leaf development. Dev Biol. 141 (1), 220-232 (1990).
- Sylvester, A. W., Cande, W. Z., Freeling, M. Division and differentiation during normal and liguleless-1 maize leaf development. Development. 110 (3), 985-1000 (1990).
- Moreno, M. A., Harper, L. C., Krueger, R. W., Dellaporta, S. L., Freeling, M. liguleless1 encodes a nuclear-localized protein required for induction of ligules and auricles during maize leaf organogenesis. Genes Dev. 11 (5), 616-628 (1997).
- Emerson, R. A. The inheritance of the ligule and auricle of corn leaves. Neb. Agr. Exp. Sta. An. Rep. 25, 81-85 (1912).
- Moon, J., Candela, H., Hake, S. The Liguleless narrow mutation affects proximal-distal signaling and leaf growth. Development. 140 (2), 405-412 (2013).
- Johnston, R., et al. Transcriptomic Analyses Indicate That Maize Ligule Development Recapitulates Gene Expression Patterns That Occur during Lateral Organ Initiation. Plant Cell. 26 (12), 4718-4732 (2014).
- Schmid, M. W., et al. A powerful method for transcriptional profiling of specific cell types in eukaryotes: laser-assisted microdissection and RNA sequencing. PLoS One. 7 (1), e29685 (2012).
- Kerk, N. M., Ceserani, T., Tausta, S. L., Sussex, I. M., Nelson, T. M. Laser capture microdissection of cells from plant tissues. Plant Physiol. 132 (1), 27-35 (2003).
- Nakazono, M., Qiu, F., Borsuk, L. A., Schnable, P. S. Laser-capture microdissection, a tool for the global analysis of gene expression in specific plant cell types: identification of genes expressed differentially in epidermal cells or vascular tissues of maize. Plant Cell. 15 (3), 583-596 (2003).
- Marioni, J. C., Mason, C. E., Mane, S. M., Stephens, M., Gilad, Y. RNA-seq: an assessment of technical reproducibility and comparison with gene expression arrays. Genome Res. 18 (9), 1509-1517 (2008).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nat Rev Genet. 10 (1), 57-63 (2009).
- Brooks, L., et al. Microdissection of shoot meristem functional domains. PLoS Genet. 5 (5), e1000476 (2009).
- Cai, S., Lashbrook, C. C. Stamen abscission zone transcriptome profiling reveals new candidates for abscission control: enhanced retention of floral organs in transgenic plants overexpressing Arabidopsis ZINC FINGER PROTEIN2. Plant Physiol. 146 (3), 1305-1321 (2008).
- Li, P., et al. The developmental dynamics of the maize leaf transcriptome. Nat Genet. 42 (12), 1060-1067 (2010).
- Eveland, A. L., et al. Regulatory modules controlling maize inflorescence architecture. Genome Res. 24 (3), 431-443 (2014).
- Takacs, E. M., et al. Ontogeny of the maize shoot apical meristem. Plant Cell. 24 (8), 3219-3234 (2012).
- Aida, M., Ishida, T., Tasaka, M. Shoot apical meristem and cotyledon formation during Arabidopsis embryogenesis: interaction among the CUP-SHAPED COTYLEDON and SHOOT MERISTEMLESS genes. Development. 126 (8), 1563-1570 (1999).
- Ishida, T., Aida, M., Takada, S., Tasaka, M. Involvement of CUP-SHAPED COTYLEDON genes in gynoecium and ovule development in Arabidopsis thaliana. Plant Cell Physiol. 41 (1), 60-67 (2000).
- Takada, S., Hibara, K., Ishida, T., Tasaka, M. The CUP-SHAPED COTYLEDON1 gene of Arabidopsis regulates shoot apical meristem formation. Development. 128 (7), 1127-1135 (2001).
- Nardmann, J., Ji, J., Werr, W., Scanlon, M. J. The maize duplicate genes narrow sheath1 and narrow sheath2 encode a conserved homeobox gene function in a lateral domain of shoot apical meristems. Development. 131 (12), 2827-2839 (2004).
- Bonner, W. A., Hulett, H. R., Sweet, R. G., Herzenberg, L. A. Fluorescence activated cell sorting. Rev Sci Instrum. 43 (3), 404-409 (1972).
- Birnbaum, K., et al. A gene expression map of the Arabidopsis root. Science. 302 (5652), 1956-1960 (2003).
- Brady, S. M., et al. A high-resolution root spatiotemporal map reveals dominant expression patterns. Science. 318 (5851), 801-806 (2007).
- Carter, A. D., Bonyadi, R., Gifford, M. L. The use of fluorescence-activated cell sorting in studying plant development and environmental responses. Int J Dev Biol. 57 (6-8), 545-552 (2013).
- Ruzin, S. E. . Plant microtechnique and microscopy. , (1999).
- Jackson, D., Veit, B., Hake, S. Expression of maize KNOTTED1 related homeobox genes in the shoot apical meristem predicts patterns of morphogenesis in the vegetative shoot. Development. 120, 405-413 (1994).
- Javelle, M., Marco, C. F., Timmermans, M. In situ hybridization for the precise localization of transcripts in plants. J Vis Exp. (57), e3328 (2011).
- Day, R. C., McNoe, L., Macknight, R. C. Evaluation of global RNA amplification and its use for high-throughput transcript analysis of laser-microdissected endosperm. Int J Plant Genomics. , 61028 (2007).

