Экспериментальный дизайн для лазерных микродиссекция РНК-Seq: уроки из анализа развития листьев кукурузы
Summary
Many developmentally important genes have cell- or tissue-specific expression patterns. This paper describes LM RNA-seq experiments to identify genes that are differentially expressed at the maize leaf blade-sheath boundary and in lg1-R mutants compared to wild-type. The experimental considerations discussed here apply to transcriptomic analyses of other developmental phenomena.
Abstract
Гены с важную роль в развитии часто имеют в пространстве и / или во времени ограниченные паттерны экспрессии. Часто эти генные транскрипты не обнаруживаются или не определяются как дифференциально экспрессируются (DE) в транскриптомных анализа целых органов растений. Лазерная микродиссекция-Seq РНК (LM-Seq РНК) является мощным инструментом для идентификации генов, которые DE в конкретных областях развития. Тем не менее, выбор клеточных доменов microdissect и сравнить, и точность microdissections имеют решающее значение для успеха экспериментов. Вот два примера иллюстрируют конструктивные соображения для транскриптомике экспериментов; Л.М. РНК-сл анализ , чтобы идентифицировать гены, которые – де вдоль листа кукурузы проксимальной-дистальной оси, а второй эксперимент , чтобы идентифицировать гены, которые в DE liguleless1-R (lg1-R) Мутанты по сравнению с диким типом. Ключевые элементы , которые внесли свой вклад в успех этих экспериментов были подробно гистологическое и в сиTu гибридизация анализ области для анализа, выбор листа зачатков на эквивалентных стадиях развития, использование морфологических ориентиров для выбора регионов для микродиссекции и микродиссекции точно измеренных доменов. В настоящем документе приводится подробный протокол для анализа областей развития с помощью LM РНК-Seq. Представленные здесь данные показывают, как область выбрана для микродиссекции будет влиять на полученные результаты.
Introduction
Лист кукурузы является идеальной моделью для исследования образования полей развития в процессе морфогенеза, поскольку она имеет четкую границу между лопаткой и оболочкой, которая поддается генетической диссекции (рис 1А). На ранних стадиях развития листьев, линейной полосы более мелких ячеек, то preligule группа (PLB), подразделяет листовой зачаток в предварительно лопаточной и предварительно оболочки доменов. Бахромой, как Язычок и треугольные ушки развиваются из PLB (рис 1А, C, D). Генетические экраны выявили мутации, которые нарушают границу лопастного оболочки. Например, рецессивный liguleless1 (lg1) мутации удаления язычка и ушные раковины 1, 2, 3, 4 (Фигура 1В). В гибридизация показало , что lg1 транскрипт накапливается в РLB и возникающих язычка, что делает его отличным маркером для язычка развития 5, 6 (рис 1E).
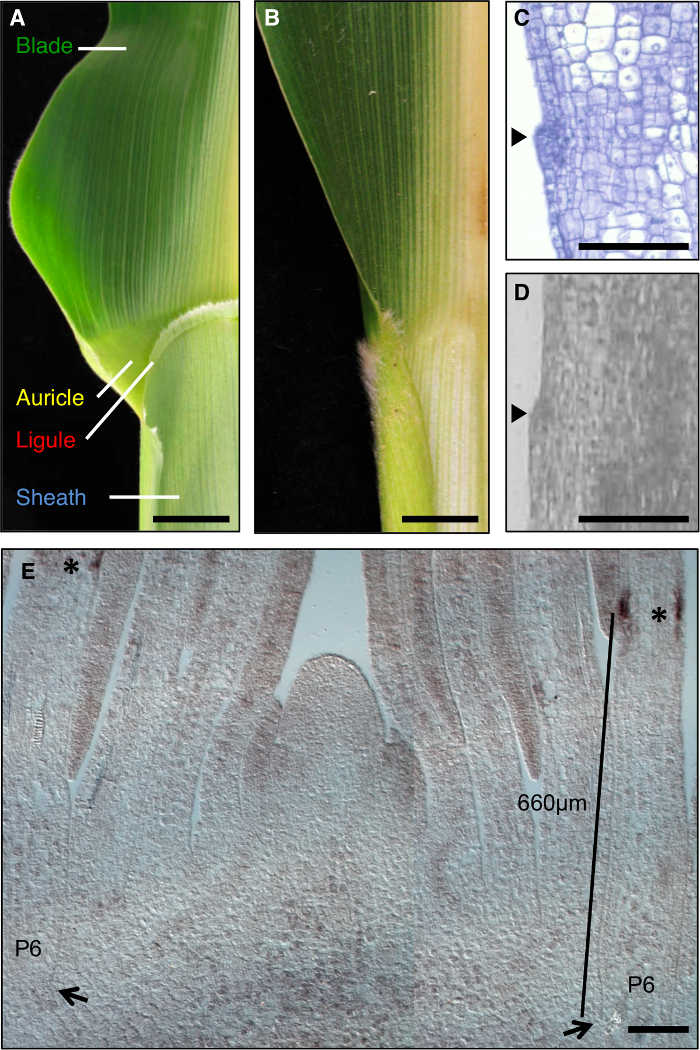
Рисунок 1: дикого типа и liguleless1-R листьев кукурузы. (А) граница Блейд-оболочка область зрелого дикого типа листа , показывающий язычка и предсердия структур. (B) граница Блейд-оболочка область зрелого liguleless1-R листьев показывая отсутствие язычка и ушной раковины структур. Листья в А и В были сокращены в два раза вдоль жилки. (С) Продольный разрез через лист примордия дикого типа. Образец был обработан и окрашивают для гистологического анализа. Инициирующий Язычок очевидно как выпуклость, выступающую из плоскости листа (стрелки). (D) Продольная разделиона через лист зачатка дикого типа. Образец был обработан для LM, как описано в тексте. Arrowhead указывает на инициирование язычка. (E) lg1 на месте гибридизации побегов верхушки корня бокового продольного разреза в. Звездочки показывают lg1 накопление транскриптов в PLB от P6 листа зачатка. Стрелки указывают основание P6 зачатка. Бар показывает измерение от основания зачатка к ИПР. Шкала баров в А и В = 20 мм. Шкала баров в CE = 100 мкм. Эта цифра была изменена из ссылки 6 (Авторское право Американского общества биологов растений). Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
В этом исследовании, Л.М. Секвенирование РНК был использован, чтобы идентифицировать набор генов, которые дифференциально экспрессируются (DE) на границе лопастного оболочки по отношению к другим частям листа зачатка и Ide ntify гены, которые DE в lg1-R мутантов по отношению к братьев и сестер дикого типа. LM-РНК Seq представляет собой метод количественного накопления транскриптов в специфических клетках или клеточных доменов 7. LM системы сочетают в себе лазер и микроскоп с цифровой камерой. Секционного ткань монтируется на салазках и просмотрены через микроскоп. Программное обеспечение LM обычно включает в себя инструменты для рисования, которые позволяют пользователю выделить любую выбранную область для микродиссекции. Разрезы лазерные вдоль линии, и выбранная ткань катапультировался за пределы слайда и в трубу, подвешенную над слайде. LM позволяет пользователю microdissect точные домены, в том числе специфических клеточных слоев и даже отдельных клеток 8, 9. РНК затем может быть извлечено из микродиссекции ткани. Затем РНК-Seq компонент использует следующего поколения последовательности в последовательности кДНК – библиотек , сгенерированные из извлеченной РНК 10,= "Xref"> 11.
Основные преимущества LM Секвенирование РНК являются способность количественного накопления транскриптов в четко определенных областях и способность к профилю весь транскриптом одновременно 7. Метод особенно подходит для зондирования ранних событий в области развития, где область интереса часто микроскопическими. Предыдущие исследования использовали LM в сочетании с микрочипов технологии для изучения процессов развития растений 9, 12, 13. РНК-Seq имеет преимущество количественной оценки транскриптов в широком динамическом диапазоне, в том числе с низкой экспрессируемых генов, и информация до последовательности не требуется 10, 11. Более того, LM-Seq РНК имеет потенциал, чтобы выделить важные гены уровню развития, которые могут быть пропущены в экранах мутагенеза из-за генетической избыточности или к летальности потерь посещающихфункция мутант.
Развивающих важные гены, такие как узкий sheath1 (ns1) и чашеобразной cotyledon2 (cuc2), часто имеют специфические паттерны экспрессии только один или несколько ячеек 17, 18, 19, 20. Многие выражаются только на ранних стадиях развития, а не в зрелом органе. Когда целые органы или крупные домены анализируются, эти клеточные специфические транскрипты разбавлены и не могут быть обнаружены в более обычных анализах. Допустив анализ точно определенных областей, LM-Seq РНК позволяет эти гены тканеспецифические быть идентифицированы и количественно.
Решающие факторы в успехе экспериментов, описанных здесь, были тщательный гистологический анализ, который руководствовался выбор соответствующей стадии развития и области для анализа и точного MeasureMeнт доменов клеточной ткани для LM. Для того, чтобы гарантировать , что эквивалентные домены были отобраны для всех повторах, ткань была собрана из листьев зачатков на той же стадии развития и микродиссекции домены были измерены относительно морфологических ориентиров , таких как формирующейся язычка (рис 2). Известно, что некоторые гены экспрессируются в градиенте от кончика к основанию листа. Измеряя точные домены, изменение из – за выборки из разных мест вдоль листа проксимальной-дистальной оси была сведена к минимальной (рис 3А). По microdissecting домены одного и того же размера, изменение за счет дифференциальной разбавление клеток специфических транскриптов также была снижена (фигура 3В). Боковые продольные участки верхушки побега были использованы для всех microdissections. Эти участки, перпендикулярные оси жилка рентабельностью (Рисунок 4). Используя только разделы, которые включают SAM гарантирует, что эквивалентные боковые участкилистовые зачатки анализируются.
В образцах , обработанных и секционного для LM, первым морфологическим признаком язычка выроста является шишка на адаксиальную стороне из – за периклинальных клеточных делений в адаксиальной эпидермиса (рис 1D, Рисунок 2). Было установлено, что формирующаяся Язычок может быть надежно идентифицирован на plastochron 7 этап листьев зачатков. Мы были заинтересованы в генах, выраженных во всем регионе язычка, включая формирующейся язычка и клетки немедленно дистальных, которые будут образовывать ушную раковину. Для того , чтобы гарантировать , что выборы эквивалентные ткани были сделаны, Язычок шишка был использован в качестве ориентира морфологического и прямоугольник 100 мкм с серединой на Язычок бугорок был выбран для LM (рис 2А, 2В). Эквивалент размера прямоугольниками предварительного лезвия и предварительной оболочки были выбраны из тех же листьев зачатков.
Анализ liguleless мутантных растений представлены различные ChalleНге; lg1-R мутанты не образуют язычка, поэтому эта морфологическая особенность не может быть использована для выбора региона для LM. Вместо этого область lg1 накопления транскриптов в листьев зачатков дикого типа была определена, и была определена область , которая охватывала бы эту область. Эти предварительные анализы проводили на проростках из того же посадки, которые были использованы для окончательного анализа, поскольку предыдущие исследования показали, что местоположение PLB изменяется в зависимости от условий роста. В гибридизация показали , что lg1 транскрипты накапливаются в PLB Р6 листьев зачатков (рис 1E). Мы выбрали домен 400-900 мкм от основания листа зачатков , который охватывает область lg1 выражения (фиолетовый прямоугольник, рисунок 2А) и захватил эти эквивалентные области из дикого типа и lg1-R растений. Чтобы свести к минимуму изменение генетического фона и условий роста при сравнении transcripбыли использованы т накопление в lg1-R и растений дикого типа, семейства разделения мутантов , братьев и сестер дикого типа.
Protocol
Representative Results
Discussion
Экспериментальный дизайн является решающим фактором в РНК-сл экспериментов. Основные соображения точный домен (ы) и этап (ы) развития, которые будут проанализированы, и какие сравнения будут сделаны. Крайне важно, чтобы думать с точки зрения сравнения, так как выход, как правило, список ?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Авторы выражают благодарность С. хека за постоянное сотрудничество и стимулирующие дискуссии о развитии язычка. Эта работа поддерживается Национальным научным грантам Фонда MCB 1052051 и IOS-1848478.
Materials
| Diethyl pyrocarbonate | Sigma-Aldrich | 159220 | Used for RNase treatment of solutions |
| Razor blades | Electron Microscopy Sciences | 72000 | |
| RNase Zap | Sigma-Aldrich | R2020-250ML | RNase decontamination solution |
| Ethanol absolute 200 proof | Fisher Scientific | BP28184 | |
| Acetic acid, glacial | Sigma-Aldrich | A6283 | |
| Glass vials – 22mL | VWR | 470206-384 | |
| Xylenes, histological grade | Sigma-Aldrich | 534056-4L | |
| Paraplast plus | Sigma-Aldrich | P3683-1KG | |
| Disposable base molds, 15 x 15 x 5mm | VWR | 15154-072 | |
| Embedding rings | VWR | 15154-303 | |
| Silica gel packets | Electron Microscopy Sciences | 71206-01 | Desiccant for storage of paraffin blocks |
| Oven | Fisher Scientific | 15-103-0503 | Oven must maintain temperature of 60° C |
| Paraffin embedding station | Leica | EG1160 | |
| Microtome | Leica | RM2235 | |
| Slide warmer | Electron Microscopy Sciences | 71317-10 | |
| Coplin jars | Electron Microscopy Sciences | 70316-02 | |
| Laser microdissector | Zeiss | ||
| KIMWIPES™ Delicate Task Wipers | Kimberly-Clark Professional | 34120 | Lint-free wipes for wicking excess solutions from microscope slides |
| Membrane Slide 1.0 PEN | Zeiss | 415190-9041-000 | Slides for laser microdissection |
| Adhesive Cap 200 opaque | Zeiss | 415190-9181-000 | Tubes for laser microdissection |
| PicoPure RNA Isolation Kit | ThermoFisher Scientific | KIT0204 |
References
- Becraft, P. W., Bongard-Pierce, D. K., Sylvester, A. W., Poethig, R. S., Freeling, M. The liguleless-1 gene acts tissue specifically in maize leaf development. Dev Biol. 141 (1), 220-232 (1990).
- Sylvester, A. W., Cande, W. Z., Freeling, M. Division and differentiation during normal and liguleless-1 maize leaf development. Development. 110 (3), 985-1000 (1990).
- Moreno, M. A., Harper, L. C., Krueger, R. W., Dellaporta, S. L., Freeling, M. liguleless1 encodes a nuclear-localized protein required for induction of ligules and auricles during maize leaf organogenesis. Genes Dev. 11 (5), 616-628 (1997).
- Emerson, R. A. The inheritance of the ligule and auricle of corn leaves. Neb. Agr. Exp. Sta. An. Rep. 25, 81-85 (1912).
- Moon, J., Candela, H., Hake, S. The Liguleless narrow mutation affects proximal-distal signaling and leaf growth. Development. 140 (2), 405-412 (2013).
- Johnston, R., et al. Transcriptomic Analyses Indicate That Maize Ligule Development Recapitulates Gene Expression Patterns That Occur during Lateral Organ Initiation. Plant Cell. 26 (12), 4718-4732 (2014).
- Schmid, M. W., et al. A powerful method for transcriptional profiling of specific cell types in eukaryotes: laser-assisted microdissection and RNA sequencing. PLoS One. 7 (1), e29685 (2012).
- Kerk, N. M., Ceserani, T., Tausta, S. L., Sussex, I. M., Nelson, T. M. Laser capture microdissection of cells from plant tissues. Plant Physiol. 132 (1), 27-35 (2003).
- Nakazono, M., Qiu, F., Borsuk, L. A., Schnable, P. S. Laser-capture microdissection, a tool for the global analysis of gene expression in specific plant cell types: identification of genes expressed differentially in epidermal cells or vascular tissues of maize. Plant Cell. 15 (3), 583-596 (2003).
- Marioni, J. C., Mason, C. E., Mane, S. M., Stephens, M., Gilad, Y. RNA-seq: an assessment of technical reproducibility and comparison with gene expression arrays. Genome Res. 18 (9), 1509-1517 (2008).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nat Rev Genet. 10 (1), 57-63 (2009).
- Brooks, L., et al. Microdissection of shoot meristem functional domains. PLoS Genet. 5 (5), e1000476 (2009).
- Cai, S., Lashbrook, C. C. Stamen abscission zone transcriptome profiling reveals new candidates for abscission control: enhanced retention of floral organs in transgenic plants overexpressing Arabidopsis ZINC FINGER PROTEIN2. Plant Physiol. 146 (3), 1305-1321 (2008).
- Li, P., et al. The developmental dynamics of the maize leaf transcriptome. Nat Genet. 42 (12), 1060-1067 (2010).
- Eveland, A. L., et al. Regulatory modules controlling maize inflorescence architecture. Genome Res. 24 (3), 431-443 (2014).
- Takacs, E. M., et al. Ontogeny of the maize shoot apical meristem. Plant Cell. 24 (8), 3219-3234 (2012).
- Aida, M., Ishida, T., Tasaka, M. Shoot apical meristem and cotyledon formation during Arabidopsis embryogenesis: interaction among the CUP-SHAPED COTYLEDON and SHOOT MERISTEMLESS genes. Development. 126 (8), 1563-1570 (1999).
- Ishida, T., Aida, M., Takada, S., Tasaka, M. Involvement of CUP-SHAPED COTYLEDON genes in gynoecium and ovule development in Arabidopsis thaliana. Plant Cell Physiol. 41 (1), 60-67 (2000).
- Takada, S., Hibara, K., Ishida, T., Tasaka, M. The CUP-SHAPED COTYLEDON1 gene of Arabidopsis regulates shoot apical meristem formation. Development. 128 (7), 1127-1135 (2001).
- Nardmann, J., Ji, J., Werr, W., Scanlon, M. J. The maize duplicate genes narrow sheath1 and narrow sheath2 encode a conserved homeobox gene function in a lateral domain of shoot apical meristems. Development. 131 (12), 2827-2839 (2004).
- Bonner, W. A., Hulett, H. R., Sweet, R. G., Herzenberg, L. A. Fluorescence activated cell sorting. Rev Sci Instrum. 43 (3), 404-409 (1972).
- Birnbaum, K., et al. A gene expression map of the Arabidopsis root. Science. 302 (5652), 1956-1960 (2003).
- Brady, S. M., et al. A high-resolution root spatiotemporal map reveals dominant expression patterns. Science. 318 (5851), 801-806 (2007).
- Carter, A. D., Bonyadi, R., Gifford, M. L. The use of fluorescence-activated cell sorting in studying plant development and environmental responses. Int J Dev Biol. 57 (6-8), 545-552 (2013).
- Ruzin, S. E. . Plant microtechnique and microscopy. , (1999).
- Jackson, D., Veit, B., Hake, S. Expression of maize KNOTTED1 related homeobox genes in the shoot apical meristem predicts patterns of morphogenesis in the vegetative shoot. Development. 120, 405-413 (1994).
- Javelle, M., Marco, C. F., Timmermans, M. In situ hybridization for the precise localization of transcripts in plants. J Vis Exp. (57), e3328 (2011).
- Day, R. C., McNoe, L., Macknight, R. C. Evaluation of global RNA amplification and its use for high-throughput transcript analysis of laser-microdissected endosperm. Int J Plant Genomics. , 61028 (2007).

